
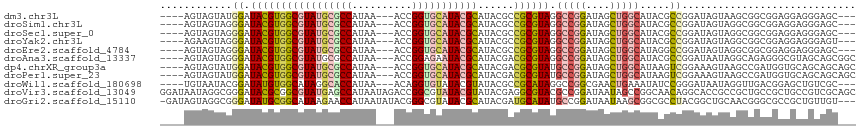
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 8,208,795 – 8,208,901 |
| Length | 106 |
| Max. P | 0.987615 |

| Location | 8,208,795 – 8,208,901 |
|---|---|
| Length | 106 |
| Sequences | 11 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 75.96 |
| Shannon entropy | 0.50719 |
| G+C content | 0.57606 |
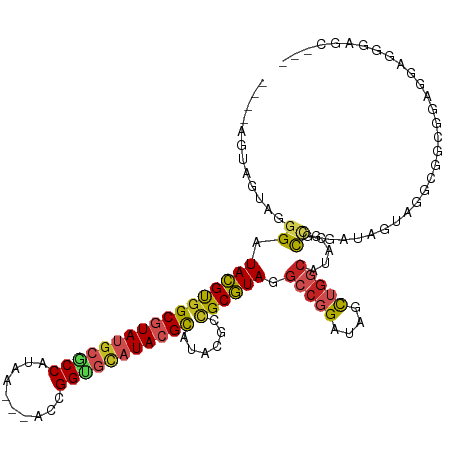
| Mean single sequence MFE | -43.84 |
| Consensus MFE | -24.75 |
| Energy contribution | -25.05 |
| Covariance contribution | 0.30 |
| Combinations/Pair | 1.42 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.56 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.29 |
| SVM RNA-class probability | 0.987615 |
| Prediction | RNA |
| WARNING | Out of training range. z-scores are NOT reliable. |
Download alignment: ClustalW | MAF
>dm3.chr3L 8208795 106 - 24543557 ----AGUAGUAUGGAUACGUGGCGUAUGCGCCAUAA---ACCGGUGCAUACGCAUACGCCGCGUAGGCCGGAUAGCUGGCAUACGCCGGAUAGUAAGCGGCGGAGGAGGGAGC--- ----.....((((....))))(((((((((((....---...)))))))))))...(((((((((.(((((....))))).)))((......))..))))))...........--- ( -44.40, z-score = -2.13, R) >droSim1.chr3L 7672624 106 - 22553184 ----AGUAGUAGGGAUACGUGGCGUAUGCGCCAUAA---ACCGGUGCAUACGCAUACGCCGCGUAGGCCGGAUAGCUGGCAUACGCCGGAUAGUAGGCGGCGGAGGAGGGAGC--- ----.............(((((((((((((((....---...))))))))))).))))(((((((.(((((....))))).)))(((........))).))))..........--- ( -46.60, z-score = -2.68, R) >droSec1.super_0 494825 106 - 21120651 ----AGUAGUAGGGAUACGUGGCGUAUGCGCCAUAA---ACCGGUGCAUACGCAUACGCCGCGUAGGCCGGAUAGCUGGCAUACGCCGGAUAGUAGGCGGCGGAGGAGGGAGC--- ----.............(((((((((((((((....---...))))))))))).))))(((((((.(((((....))))).)))(((........))).))))..........--- ( -46.60, z-score = -2.68, R) >droYak2.chr3L 20508150 106 + 24197627 ----AGAAGUAGGGAUACGUGGCGUAUGCGCCAUAA---ACCGGUGCAUACGCAUACGCCGCGUAGGCCGGAUAGCUGGCAUACGCCGGAUAGUAGGCGGCGGAGGAGGGAGU--- ----.............(((((((((((((((....---...))))))))))).))))(((((((.(((((....))))).)))(((........))).))))..........--- ( -46.60, z-score = -3.14, R) >droEre2.scaffold_4784 19937964 106 + 25762168 ----AGUAGUAGGGAUACGUGGCGUAUGCGCCAUAA---ACCGGUGCAUACGCAUACGCCGCGUAGGCCGGAUAGCUGGCAUAGGCCGGAUAGUAGGCGGCGGAGGAGGGAGC--- ----.............(((((((((((((((....---...))))))))))).))))((((....(((((....)))))....(((........))).))))..........--- ( -43.60, z-score = -2.06, R) >droAna3.scaffold_13337 3212030 109 - 23293914 ----AGUAGUAGGGAUACGUGGCGUAUGCGCCAUAA---ACCGGAGAAUACGCAUACGACGCGUAGGCCGGAUAGCUGGCAUACGCCGGAUAAUAGGCAGAGGGCGUAGCAGCGGC ----....(((....)))((((((....))))))..---.((((....(((((.......)))))..))))...((((.(.((((((...............)))))))))))... ( -39.26, z-score = -1.55, R) >dp4.chrXR_group3a 1368215 109 + 1468910 ----AGUAGUAUGGAUACGUGGCGUAUGCGCCAUAA---ACCGGUGCAUACGCAUACGACGCGUAUGCCGGAUAGCUGGCAUAAGUCGGAAAGUAAGCCGAUGGUGCAGCAGCAGC ----.(((.(((((.(((...(((((((((((....---...)))))))))))...((((...((((((((....)))))))).))))....)))..)).))).)))......... ( -46.30, z-score = -3.15, R) >droPer1.super_23 1559112 109 + 1662726 ----AGUAGUAUGGAUACGUGGCGUAUGCGCCAUAA---ACCGGUGCAUACGCAUACGACGCGUAUGCCGGAUAGCUGGCAUAAGUCGGAAAGUAAGCCGAUGGUGCAGCAGCAGC ----.(((.(((((.(((...(((((((((((....---...)))))))))))...((((...((((((((....)))))))).))))....)))..)).))).)))......... ( -46.30, z-score = -3.15, R) >droWil1.scaffold_180698 2151780 106 - 11422946 ----UGUAAUACGGAUAUGUGGCAUAGGCACCAUAA---ACAGGUGUAUACGUAUACGCCGCAUAGGCCGGCGAACUGAAAUAUCCGGGAUAAUAGGUUGACGGAGCUGUCGC--- ----.......(((.((((((((....(((((....---...)))))....(....)))))))))..)))((((...(.....((((.(((.....)))..)))).)..))))--- ( -32.10, z-score = -0.99, R) >droVir3.scaffold_13049 22629691 116 + 25233164 GGAUAAUAGGCGGGAUACGCGGCGUAUGAGCCAUAAUAGACCGGCGUAUACGUAUACGAGGCGUACGCCGGAUAAUAGCCGGCAACAGGCACCGCCGCUGCCGCUGCCGUCGCAGC ........(((((.......(((......)))........(((((((..((((.......)))))))))))......((((....).))).)))))(((((.((....)).))))) ( -48.20, z-score = -1.05, R) >droGri2.scaffold_15110 20966690 112 + 24565398 -GAUAGUAGGCGGGAUAUGCGGCAUAAGAACCAUAAUAUACGGGCGUAUACGCAUACGAUGCAUAUGCCGGAUAAUAAGCGGCGCCUACGGCUGCAACGGGCGCCGCUGUUGU--- -.........(((.((((((((........))........((.(((....)))...))..)))))).))).((((((.((((((((..((.......))))))))))))))))--- ( -42.30, z-score = -1.31, R) >consensus ____AGUAGUAGGGAUACGUGGCGUAUGCGCCAUAA___ACCGGUGCAUACGCAUACGCCGCGUAGGCCGGAUAGCUGGCAUACGCCGGAUAGUAGGCGGCGGAGGAGGGAGC___ ............((.(((((((((((((((((..........)))))))))))......)))))).(((((....))))).....))............................. (-24.75 = -25.05 + 0.30)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:11:02 2011