| Sequence ID | dm3.chr3L |
|---|---|
| Location | 7,796,592 – 7,796,687 |
| Length | 95 |
| Max. P | 0.991260 |

| Location | 7,796,592 – 7,796,687 |
|---|---|
| Length | 95 |
| Sequences | 11 |
| Columns | 121 |
| Reading direction | forward |
| Mean pairwise identity | 71.55 |
| Shannon entropy | 0.54199 |
| G+C content | 0.65569 |
| Mean single sequence MFE | -47.50 |
| Consensus MFE | -27.21 |
| Energy contribution | -27.55 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.06 |
| Structure conservation index | 0.57 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.47 |
| SVM RNA-class probability | 0.991260 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
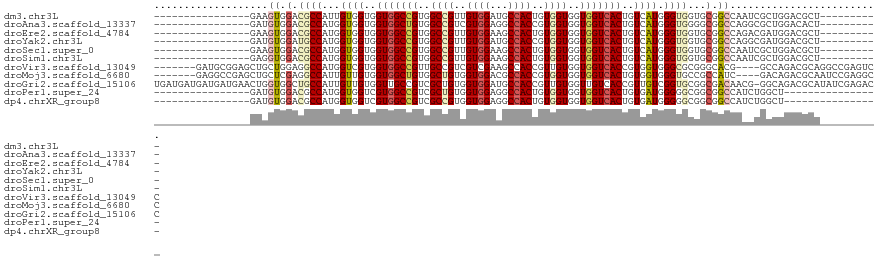
>dm3.chr3L 7796592 95 + 24543557 ----------------GAAGUGGACGCCAUUGUGGUGGUGGCCGUGGCCGUUGUGGAUGCCACUGUGGUGGUGGUCACUGUCAUGGGUGGUGCGGCCAAUCGCUGGACGCU---------- ----------------..((((..((((((..(((..(((((((..((((..((((...))))..))))..)))))))..)))..).))))((((....)))).)..))))---------- ( -44.50, z-score = -2.08, R) >droAna3.scaffold_13337 10588635 95 + 23293914 ----------------GAUGUGGACGCCAUGGUGGUGGUGGCUGUGGCCGUCGUGGAGGCCACCGUGGUGGUGGUCACUGUCAUGGGUGGGGCGGCCAGGCGCUGGACACU---------- ----------------...(((..((((.(.((((..(((((..(.((((..((((...))))..)))).)..)))))..)))).)....)))).((((...)))).))).---------- ( -43.50, z-score = -0.58, R) >droEre2.scaffold_4784 22207181 95 - 25762168 ----------------GAAGUGGACGCCAUGGUGGUGGUGGCCGUGGCCGUUGUGGAAGCCACUGUGGUGGUGGUCACUGUCAUGGGUGGUGCGGCCAGACGAUGGACGCU---------- ----------------........((((...((((..(((((((..((((..((((...))))..))))..)))))))..)))).))))..(((.(((.....))).))).---------- ( -43.10, z-score = -2.06, R) >droYak2.chr3L 8392182 95 + 24197627 ----------------GAUGUGGAUGCCAUGGUGGUGGUGGCCGUGGCCGUUGUGGAUGCCACCGUGGUGGUGGUCACUGUCAUGGGUGGUGCGGCCAGGCGAUGGACGCU---------- ----------------.........(((((.((((..(((((((..((((..((((...))))..))))..)))))))..)))).).))))(((.(((.....))).))).---------- ( -42.60, z-score = -1.05, R) >droSec1.super_0 103250 95 + 21120651 ----------------GAAGUGGACGCCAUGGUGGUGGUGGCCGUGGCCGUUGUGGAAGCCACUGUGGUGGUGGUCACUGUCAUGGGUGGUGCGGCCAAUCGCUGGACGCU---------- ----------------..((((..((((((.((((..(((((((..((((..((((...))))..))))..)))))))..)))).).))))((((....)))).)..))))---------- ( -43.80, z-score = -1.90, R) >droSim1.chr3L 7283980 95 + 22553184 ----------------GAGGUGGACGCCAUGGUGGUGGUGGCCGUGGCCGUUGUGGAAGCCACUGUGGUGGUGGUCACUGUCAUGGGUGGUGCGGCCAAUCGCUGGACGCU---------- ----------------..(((.(.((((((.((((..(((((((..((((..((((...))))..))))..)))))))..)))).).)))))).)))....((.....)).---------- ( -45.20, z-score = -2.07, R) >droVir3.scaffold_13049 20591068 110 + 25233164 -------GAUGCGGAGCUGCUGGAGGCCAUGGUCGUGGUGGCCGUUGCCGUCGUCGAAGCCACCGUUGUGGUGGUCACCGUGGUGGGCGCGGGCACG----GCCAGACGCAGGCCGAGUCC -------(((.(((..((((.....(((((....)))))((((((.(((..((((...((((((.....))))))(((....)))))))..))))))----)))....)))).))).))). ( -53.50, z-score = -0.61, R) >droMoj3.scaffold_6680 8300348 110 + 24764193 -------GAGGCCGAGCUGCUCGAGGCCAUUGUUGUGGUGGCUGUGGCUGUGGUGGACGCCACCGUGGUGGUGGUCACUGUGGUGGGUGCCGCCAUC----GACAGACGCAAUCCGAGGCC -------..(((((((...)))...((..(((((((((((((..(.((((..(((..((((((....))))))..)))..)))).)..))))))).)----)))))..)).......)))) ( -53.50, z-score = -1.74, R) >droGri2.scaffold_15106 40992 120 + 101566 UGAUGAUGAUGAUGAACUGGUGGCUGCCAUUGUUGUGGUUGCCGUCGCUGUGGUGGAUGCCACCGUUGUGGUUGUCACCGUUGUCGGUGCGGCGACAACG-GGCAGACGCAUAUCGAGACC .........(((((....(..(((.(((((....))))).)))..)......(((..((((..((((((.((((.(((((....))))))))).))))))-))))..))).)))))..... ( -50.10, z-score = -1.97, R) >droPer1.super_24 1356070 89 + 1556852 ----------------GAUGUGGACGCCAUGGUGGUCGUGGCCGUCGCUGUGGUGGAGGCCACUGUGGUGGUGGUCACUGUGAUGGGGGCGGCGGCCAUCUGGCU---------------- ----------------.........((((.(((((((((.(((.(((..(..((((..(((((....)))))..))))..)..))).))).))))))))))))).---------------- ( -50.70, z-score = -4.39, R) >dp4.chrXR_group8 7517925 89 - 9212921 ----------------GAUGUGGACGCCAUGGUGGUCGUGGCCGUCGCCGUGGUGGAGGCCACUGUGGUGGUGGUCACUGUGAUGGGGGCGGCGGCCAUCUGGCU---------------- ----------------.........((((.(((((((((.(((.(((.((..((((..(((((....)))))..))))..)).))).))).))))))))))))).---------------- ( -52.00, z-score = -4.18, R) >consensus ________________GAGGUGGACGCCAUGGUGGUGGUGGCCGUGGCCGUUGUGGAAGCCACUGUGGUGGUGGUCACUGUCAUGGGUGGGGCGGCCAAGCGCUGGACGCU__________ ...................((.(.((((...((((..(((((((..((((..((((...))))..))))..)))))))..)))).))))...).))......................... (-27.21 = -27.55 + 0.33)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:10:10 2011