
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 6,238,090 – 6,238,204 |
| Length | 114 |
| Max. P | 0.836519 |

| Location | 6,238,090 – 6,238,204 |
|---|---|
| Length | 114 |
| Sequences | 14 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 72.37 |
| Shannon entropy | 0.61987 |
| G+C content | 0.52128 |
| Mean single sequence MFE | -40.46 |
| Consensus MFE | -14.98 |
| Energy contribution | -14.09 |
| Covariance contribution | -0.89 |
| Combinations/Pair | 1.72 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.37 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.86 |
| SVM RNA-class probability | 0.836519 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 6238090 114 - 24543557 AUUAUCUACAGUGAGUUCACGUCCAUGUUGGUGGACCGCGCCAUGCAGUUUGGCACCAGUUCAAAUGUUCAUGCCCUGCCACUUUUCGGAGGUGGUAAAACACUGUAAGUACUC--- ..((..(((((((...((((.(((.....(((((.....((((.......))))..(((..((........))..))))))))....))).)))).....)))))))..))...--- ( -33.70, z-score = -1.05, R) >droMoj3.scaffold_6680 14528448 114 + 24764193 AUUGUCUACAGUGAGUUUACGGCGAUGUUGGUGGAUCGUGCCAUGCAAGUGGGCACCAGCUCAACGGUGCAUGCCCUGCCCAUUUUCGGGGGCGGCAAGAAACUGUGAGUGCCC--- ..(((((((.(((....)))((((..(((....)))..))))......)))))))...(((((.((((...((((..((((........))))))))....)))))))))....--- ( -36.50, z-score = 1.19, R) >droAna3.scaffold_13337 2590657 117 + 23293914 AUCAUCUACAGCGAGUUCACCUCUAUGCUAGUGGACAGGGCCAUGGUCUUUGGGACCAGCUCCAAGGUCCAUGCCUUGCCACUUUUUGGUGGCGGAAAAACGCUGUAAGUAUCAAAC ......(((((((..(((.((.(((....(((((.((((((.((((.(((((((......))))))).)))))))))))))))...))).)).)))....))))))).......... ( -50.40, z-score = -4.91, R) >droEre2.scaffold_4784 8926683 114 - 25762168 AUCAUCUACAGUGAGUUCACGUCCAUGUUGGUGGACCGUGCCAUGCAGUUUGGCACCAGCUCAAAUGUCCAUGCGCUUCCACUCUUCGGAGGUGGUAAAACACUGUAAGUGCUC--- ..((..(((((((...((((.(((.....((((((..((((.(((..(((((((....)).)))))...))))))).))))))....))).)))).....)))))))..))...--- ( -40.10, z-score = -2.36, R) >droYak2.chr3L 6818252 114 - 24197627 AUCAUCUACAGUGAGUUCACAUCCAUGCUGGUGGACCGGGCCAUGCAGUUUGGCACCAGCUCAAAGGUCCAUGCCCUUCCACUCUUCGGAGGUGGUAAAACACUGUAAGUGCUC--- ..((..(((((((.............(((((((..((((((......)))))))))))))...........(((((((((.......))))).))))...)))))))..))...--- ( -39.70, z-score = -1.64, R) >droSec1.super_2 6204079 114 - 7591821 AUUAUCUACAGUGAGUUCACGUCCAUGUUGGUGGACCGUGCCAUGCAGUUUGGCACCAGCUCAAAUGUCCAUGCCCUGCCACUUUUCGGAGGUGGUAAAACACUGUAAGUAUUC--- ..((..(((((((((((...((((((....)))))).((((((.......)))))).)))))..............(((((((((...)))))))))....))))))..))...--- ( -37.70, z-score = -2.20, R) >droSim1.chr3L 5731688 114 - 22553184 AUUAUCUACAGUGAGUUCACGUCCAUGUUGGUGGACCGGGCCAUGCAGUUUGGCACCAGCUCAAAUGUCCAUGCCCUGCCACUUUUCGGAGGUGGUAAAACCCUGUAAGUAUUC--- ..((..(((((.(.((((((.(((.....(((((.(.((((.(((..(((((((....)).)))))...))))))).))))))....))).))))....))))))))..))...--- ( -37.90, z-score = -1.75, R) >anoGam1.chr2L 17534690 111 - 48795086 AUUCUGUACGGUGAGUUUACCACGCUGCUCGUCGAUCGUACCAUCGGCAUCGGCAAAUCGACCGACACGGCCAUCCUGUCGAUAUUCGGUGGCGGCAGAGUGCUGUAAGUA------ (((((((..((((....)))).(((..((.((((((......))))))(((((((....(.(((...))).)....)))))))....))..))))))))))..........------ ( -35.60, z-score = 0.10, R) >droWil1.scaffold_180949 637869 111 + 6375548 AUCAUUUAUAGUGAAUUCACAUCCAUGCUGGUGGAUCGUGCCAUGGACUAUGGUACUAGCUCCAAUGUAAAUGCCUUGCCUCUAUUUGGCGGUGGCAAAGUAUUGUGAGUA------ ..........(((....)))((((((....)))))).((((((((...))))))))..(((((((((....(((((((((.......))))).))))...))))).)))).------ ( -35.90, z-score = -1.99, R) >droPer1.super_23 253458 110 - 1662726 AUCAUUUACAGCGAGUUCACGUCGAUGCUGGUGGACCGGGCCAUGGAGUUCGGGACGAGCUCGAAGGUCCACGCGUUGCCCCUCUUCGGGGGCGGAAAGUCGCUGUAAGU------- ...(((((((((((.((..(...(((((.((....))(((((.(.(((((((...))))))).).)))))..)))))((((((....)))))))..)).)))))))))))------- ( -54.60, z-score = -4.02, R) >dp4.chrXR_group3a 250270 110 - 1468910 AUCAUUUACAGCGAGUUCACGUCGAUGCUGGUGGACCGGGCCAUGGAGUUCGGGACGAGCUCGAAGGUCCACGCGUUGCCCCUCUUCGGGGGCGGAAAGUCGCUGUAAGU------- ...(((((((((((.((..(...(((((.((....))(((((.(.(((((((...))))))).).)))))..)))))((((((....)))))))..)).)))))))))))------- ( -54.60, z-score = -4.02, R) >droVir3.scaffold_13049 10542141 111 - 25233164 AUUGUUUAUAGUGAAUUUACUGCCAUGUUGGUGGAUCGUGCCAUGCAAGUGGGCACCAGCUCGACGGUACAUGCCCUGCCCCUUUUGGGAGGCGGCAAGAAAUUGUGAGUA------ ....((((((((.......(..((.....))..).((.((((........(((((...(((....)))...))))).(((((.....)).))))))).)).))))))))..------ ( -33.50, z-score = 0.28, R) >droGri2.scaffold_15110 15863299 110 + 24565398 AUUGUAUACAGUGAAUUCACCGCCAUGCUGGUGGACAGGGCCAUACAAGUGGGCACCAGCUCAACGGUGCACGCCCUGCCCAUCUUUGGGGGCGGCAAGAAACUGUAAGU------- ......((((((.......(((((.....(((((.(((((((((....))).(((((........)))))..)))))).)))))......)))))......))))))...------- ( -45.92, z-score = -2.22, R) >apiMel3.Group2 2680582 114 - 14465785 AUACAAUACGGCGAGUUCACCACGUUGCUGGUGGAUCGAAACAUGAAGAAUCACACGAGCACGCCGACCCUAAUAAUGAAGUGGUUCGGUGGAGGAAAGGUCUUGUAAGUCUUG--- ...(((.((.(((((...(((....((((.(((((((..........).))).))).))))((((((((((........)).)).)))))).......))))))))..)).)))--- ( -30.30, z-score = -0.25, R) >consensus AUUAUCUACAGUGAGUUCACGUCCAUGCUGGUGGACCGGGCCAUGCAGUUUGGCACCAGCUCAAAGGUCCAUGCCCUGCCACUUUUCGGAGGCGGCAAAACACUGUAAGUA______ ......(((((((.((((((..........)))))).......................................(((((..........))))).....))))))).......... (-14.98 = -14.09 + -0.89)

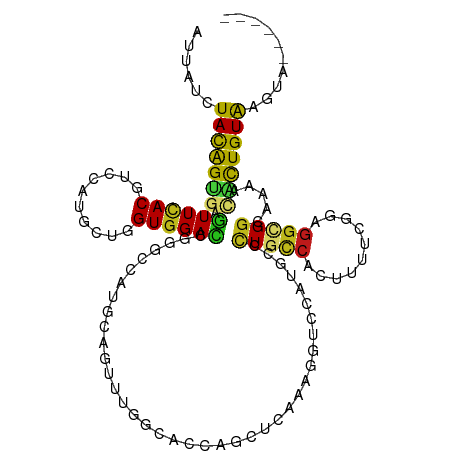
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:06:46 2011