
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 4,781,002 – 4,781,121 |
| Length | 119 |
| Max. P | 0.719771 |

| Location | 4,781,002 – 4,781,104 |
|---|---|
| Length | 102 |
| Sequences | 12 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 91.67 |
| Shannon entropy | 0.17042 |
| G+C content | 0.48625 |
| Mean single sequence MFE | -35.03 |
| Consensus MFE | -25.10 |
| Energy contribution | -25.11 |
| Covariance contribution | 0.01 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.72 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.22 |
| SVM RNA-class probability | 0.598713 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 4781002 102 - 24543557 -GGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAAGGCUUAAAUGG-----CCCGACGACGUGAA-------- -(.((((((((.(((.(((......)))))))))))))).).....((((((((((.((((......(((((.....)))))....)))-----).))..))))))))-------- ( -35.30, z-score = -2.57, R) >droEre2.scaffold_4784 7497479 102 - 25762168 -GGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAAGGCUUAAAUGG-----CCCGGCGACGUGAA-------- -(.((((((((.(((.(((......)))))))))))))).).....((((((((((.((((......(((((.....)))))....)))-----).)))..)))))))-------- ( -35.70, z-score = -2.01, R) >droYak2.chr3L 5363591 102 - 24197627 -GGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAAGGCUUAAAUGG-----CCCGGCGACGUGAA-------- -(.((((((((.(((.(((......)))))))))))))).).....((((((((((.((((......(((((.....)))))....)))-----).)))..)))))))-------- ( -35.70, z-score = -2.01, R) >droSim1.chr3L 4313426 102 - 22553184 -GGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAAGGCUUAAAUGG-----CCCGACGACGUGAA-------- -(.((((((((.(((.(((......)))))))))))))).).....((((((((((.((((......(((((.....)))))....)))-----).))..))))))))-------- ( -35.30, z-score = -2.57, R) >droSec1.super_2 4767280 102 - 7591821 -GGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAAGGCUUAAAUGG-----CCCGACGACGUGAA-------- -(.((((((((.(((.(((......)))))))))))))).).....((((((((((.((((......(((((.....)))))....)))-----).))..))))))))-------- ( -35.30, z-score = -2.57, R) >droAna3.scaffold_13337 2868925 102 - 23293914 -GGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAAGGCUUAAAUGG-----CCCGACGACGUGAG-------- -(.((((((((.(((.(((......)))))))))))))).)......(((((((((.((((......(((((.....)))))....)))-----).))..))))))).-------- ( -35.10, z-score = -2.50, R) >dp4.chrXR_group8 2922853 112 - 9212921 GGGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAAGGCUUAAAUGG----CCCCGACGACGUGAGGCAGCGCC (((((((.....(((.(((......))))))....(((((((((......)))..))))))......(((((.....)))))....)))----))))..((.(....)....)).. ( -37.70, z-score = -0.95, R) >droPer1.super_24 1422243 112 + 1556852 AGGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUUCGCGCCACUAAGUGGCCUGCUUAAGGCUUAAAUGG----CCCCGACGACGUGAGGCAGCGCC .((((((.....(((.(((......))))))....(((((((((......)))..))))))......(((((.....)))))....)))----)))((....(....)....)).. ( -34.50, z-score = -0.32, R) >droWil1.scaffold_180698 883633 103 + 11422946 GGAGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGAGCACAGAUUUAUGUUCGCGCCACUAAGUGGCCUGCUUAAGGCUUAAAUGC-----UGCGACGACGUGAG-------- .(..(((((((.(((.(((......)))))))))))))..)......(((((((((((((((...)))))........(((......))-----)))))..)))))).-------- ( -30.60, z-score = -1.48, R) >droVir3.scaffold_12758 462553 107 - 972128 -GAGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGCGCACAGAUUUAUGUCUGUGGCACUAAGUGGCCUGCUUAAGGCUCAAAUGGCCGACGCCGUCGACGUGAG-------- -..((((((((.(((.(((......)))))))))))))).(((((((....))))))).(((....((((((.....)))).)).(((((....)))))....)))..-------- ( -37.50, z-score = -2.38, R) >droMoj3.scaffold_6680 1561022 101 - 24764193 -CGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGAGCACAGAUUUAUGUCUGUGCCACUAAGUGGGCUGCUUAAGGCUUAAAUGG------CCGACGACGUGAG-------- -...(((((((.(((.(((......))))))))))))).((((((((....))))))))(((...((.((((..(((.....)))..))------)).))...)))..-------- ( -35.10, z-score = -3.39, R) >droGri2.scaffold_15110 23580016 102 + 24565398 GGGUCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGCGCACAGAUUUAUGUCUGUGUCACUAAGUGGCCUGCUUAAGGCUUAAAUGG------CCGACGACGUGAG-------- ..(((.......(((.(((......))))))...(((((((((((((....))))))))..(.....(((((.....))))).....))------)))).))).....-------- ( -32.60, z-score = -2.37, R) >consensus _GGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAAGGCUUAAAUGG_____CCCGACGACGUGAG________ .........(((((..(((......)))((((...(((((((((......)))..))))))(.....(((((.....))))).....).......))))...)))))......... (-25.10 = -25.11 + 0.01)


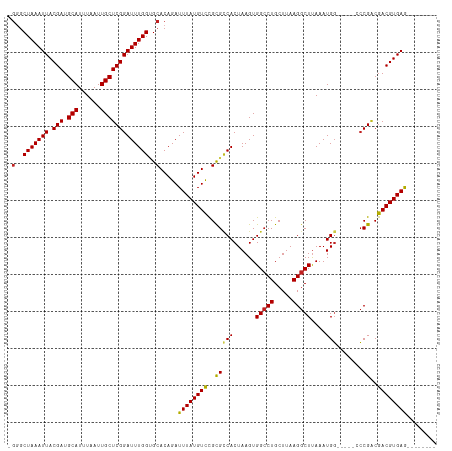
| Location | 4,781,027 – 4,781,121 |
|---|---|
| Length | 94 |
| Sequences | 12 |
| Columns | 107 |
| Reading direction | reverse |
| Mean pairwise identity | 81.71 |
| Shannon entropy | 0.40041 |
| G+C content | 0.48311 |
| Mean single sequence MFE | -30.28 |
| Consensus MFE | -18.51 |
| Energy contribution | -18.46 |
| Covariance contribution | -0.06 |
| Combinations/Pair | 1.22 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.61 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.50 |
| SVM RNA-class probability | 0.719771 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr3L 4781027 94 - 24543557 ----------GAAGGGUACUACG-GACC--GGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAA ----------.((((((.(....-))))--((((((.....(((.(((......))))))....(((((((((......)))..)))))).....)))))).))).. ( -29.00, z-score = -1.61, R) >droEre2.scaffold_4784 7497504 94 - 25762168 ----------GACGGGGACUCCG--GACCGG-GCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAA ----------..(((.....)))--((.(((-((((.....(((.(((......))))))....(((((((((......)))..)))))).....))))))).)).. ( -28.80, z-score = -1.10, R) >droYak2.chr3L 5363616 96 - 24197627 ----------GACGGGGAAAGCCCGGGCC-GGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAA ----------..((((.....))))(((.-((((((.....(((.(((......))))))....(((((((((......)))..)))))).....)))))))))... ( -33.20, z-score = -1.26, R) >droSim1.chr3L 4313451 94 - 22553184 ----------GAAGGGUGCUCCG-GACC--GGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAA ----------.....((((.(((-((((--(((((((((........)))))...)))))).))))).))))............(((((((...)))))..)).... ( -29.70, z-score = -1.04, R) >droSec1.super_2 4767305 94 - 7591821 ----------GAAGGGUGCUCCG-GACC--GGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAA ----------.....((((.(((-((((--(((((((((........)))))...)))))).))))).))))............(((((((...)))))..)).... ( -29.70, z-score = -1.04, R) >droAna3.scaffold_13337 2868950 106 - 23293914 GGAGGAUCCUGGCGGUUGGCAAGCAGACC-GGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAA (((.((..(((((((((.(....).))))-..((((((((.(((.(((......)))))))))))))))).)))..))...)))(((((((...)))))..)).... ( -33.50, z-score = -0.42, R) >dp4.chrXR_group8 2922887 98 - 9212921 --------UGGGAGAGUAGUCCU-GGUCCGGGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAA --------..(((..((((..((-(.....(.((((((((.(((.(((......)))))))))))))).).)))..)))).)))(((((((...)))))..)).... ( -30.30, z-score = -0.64, R) >droPer1.super_24 1422277 98 + 1556852 --------UGGGAGAGUAGUCCU-GGUCCAGGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUUCGCGCCACUAAGUGGCCUGCUUAA --------.....((((((.(((-(...))))((((((((.(((.(((......))))))))))))))(((((......)))..))(((((...))))))))))).. ( -28.80, z-score = -0.64, R) >droWil1.scaffold_180698 883658 105 + 11422946 --ACAACCUGGACGUAGACUGAGAAGACCGGAGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGAGCACAGAUUUAUGUUCGCGCCACUAAGUGGCCUGCUUAA --.......((((((((((((.(.....).(..(((((((.(((.(((......)))))))))))))..).))).)))))))))(((((((...)))))..)).... ( -29.80, z-score = -2.05, R) >droVir3.scaffold_12758 462583 84 - 972128 ---------------------CCUUGCC--GAGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGCGCACAGAUUUAUGUCUGUGGCACUAAGUGGCCUGCUUAA ---------------------...((((--(.((((((((.(((.(((......)))))))))))))).)((((((....)))))))))).(((((.....))))). ( -29.10, z-score = -3.11, R) >droMoj3.scaffold_6680 1561046 83 - 24764193 ----------------------UUCGCC--CGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGAGCACAGAUUUAUGUCUGUGCCACUAAGUGGGCUGCUUAA ----------------------...(((--((.(((((((.(((.(((......))))))))))))).((((((((....)))))))).......)))))....... ( -31.30, z-score = -3.92, R) >droGri2.scaffold_15110 23580040 95 + 24565398 -----------GCAACAACGGCAGCGACG-GGUCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGCGCACAGAUUUAUGUCUGUGUCACUAAGUGGCCUGCUUAA -----------........((((((.((.-.(.(((((((.(((.(((......))))))))))))))((((((((....))))))))......)).).)))))... ( -30.20, z-score = -2.14, R) >consensus __________GAAGGGUACUCCG_GGCCC_GGGCUAAAUUACGAUGCAUUUAAUUGCUCGGAUUUGGUGCACAGAUUUAUGUCCGCGCCACUAAGUGGCCUGCUUAA ..............................((((.......(((.(((......))))))((((((.....))))))...))))(((((((...)))))..)).... (-18.51 = -18.46 + -0.06)

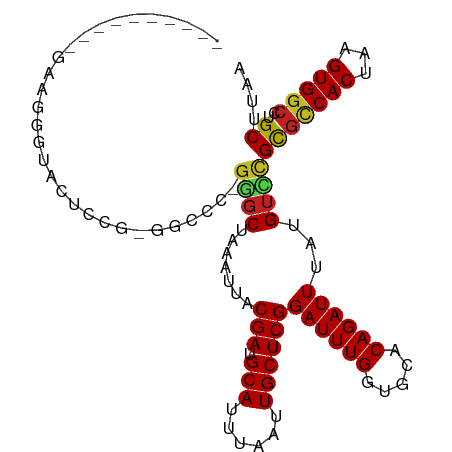

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 23:03:38 2011