
| Sequence ID | dm3.chr3L |
|---|---|
| Location | 3,060,561 – 3,060,709 |
| Length | 148 |
| Max. P | 0.995904 |

| Location | 3,060,561 – 3,060,709 |
|---|---|
| Length | 148 |
| Sequences | 5 |
| Columns | 148 |
| Reading direction | forward |
| Mean pairwise identity | 97.57 |
| Shannon entropy | 0.04239 |
| G+C content | 0.41486 |
| Mean single sequence MFE | -45.76 |
| Consensus MFE | -44.58 |
| Energy contribution | -45.02 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.13 |
| Structure conservation index | 0.97 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.86 |
| SVM RNA-class probability | 0.995904 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
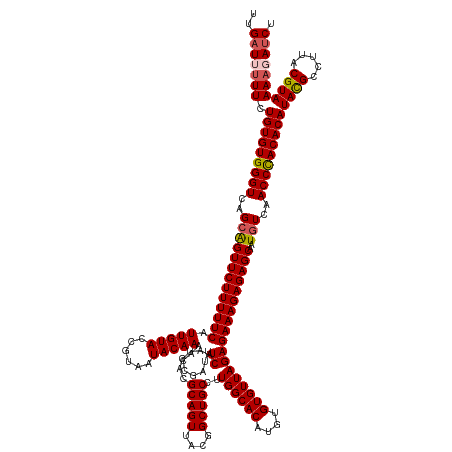
>dm3.chr3L 3060561 148 + 24543557 UUGAUUUUUCUGUGUGGGUCAGCAGUUCUUUUUCAUUGUACCGUAAUACAAAACCGAUUUCAAUGACGCAGUUACGGCUGCCUUGGCACAUGUGUGUUAGAGAAAGAGAGCAAUGUCAACCCACACAUACGCCUUACGUAAAAGAUCU ..(((((((.(((((((((..(((((((((((((.(((((......)))))........((......(((((....)))))..((((((....))))))))))))))))))..)))..)))))))))((((.....))))))))))). ( -49.00, z-score = -4.05, R) >droSim1.chr3L 2591869 148 + 22553184 UUGAUUUUUCUGUGUGGGUCAGCAGUUCUUUUUCAUUGUACCGUAAUACAAAACCGAUUUCAAUGACGCAGUUACGGCUGCCUUGGCACAUGUGUGUUAGAGAAAGAGAGCAAUGUCAACCCACACAUACGCCUUACGUAAAAGAUCU ..(((((((.(((((((((..(((((((((((((.(((((......)))))........((......(((((....)))))..((((((....))))))))))))))))))..)))..)))))))))((((.....))))))))))). ( -49.00, z-score = -4.05, R) >droSec1.super_2 3062602 148 + 7591821 UUGAUUUUUCUGUGUGGGUCAGAAGUUCUUUUUCAUUGUACCGUAAUACAAAACCUAUUUCAAUGACGCAGUUACGGCUGCCUUGGCACAUGUGUGUUAGAGAAAGAGAGCAAUGUCAACCCACACAUACGCCUUACGUAAAAGAUCU ..(((((((.(((((((((..((.((((((((((.(((((......)))))........((......(((((....)))))..((((((....))))))))))))))))))....)).)))))))))((((.....))))))))))). ( -46.20, z-score = -3.65, R) >droYak2.chr3L 15318904 148 - 24197627 UUGAUAGUUCUGUGUGGGUCAGCAGUUCUUUUUCAUUGUACCGUAAUACAAAACCGAUUUCAAUGACGCAGUUACGGCUGCCUUGGCACAUGUGUGUUAGAGAAAGAGAGCAAUGUCAACCUACACAUACGCCUUACGUAAAAGAUCU ..(((.....(((((((((..(((((((((((((.(((((......)))))........((......(((((....)))))..((((((....))))))))))))))))))..)))..)))))))))((((.....))))....))). ( -43.70, z-score = -2.33, R) >droEre2.scaffold_4784 3076584 148 + 25762168 UUGUUAUUUCUGUGUGGGUCAGCGGUUCUUUUUCAUUGUACCGUAAUACAAAACCGAUUUCAAUGACGCAGUUACGGCUGCCUUGGCACAUGUGUGUUAGAGAAAGAGAGCAAUGUCAACCUACACAUAUGCCUUACGUAAAAGAUCU ........(((((((((((..((.((((((((((.(((((......)))))........((......(((((....)))))..((((((....))))))))))))))))))...))..)))))))).((((.....))))..)))... ( -40.90, z-score = -1.57, R) >consensus UUGAUUUUUCUGUGUGGGUCAGCAGUUCUUUUUCAUUGUACCGUAAUACAAAACCGAUUUCAAUGACGCAGUUACGGCUGCCUUGGCACAUGUGUGUUAGAGAAAGAGAGCAAUGUCAACCCACACAUACGCCUUACGUAAAAGAUCU ..(((((((.(((((((((..(((((((((((((.(((((......)))))........((......(((((....)))))..((((((....))))))))))))))))))..)))..)))))))))((((.....))))))))))). (-44.58 = -45.02 + 0.44)
| Location | 3,060,561 – 3,060,709 |
|---|---|
| Length | 148 |
| Sequences | 5 |
| Columns | 148 |
| Reading direction | reverse |
| Mean pairwise identity | 97.57 |
| Shannon entropy | 0.04239 |
| G+C content | 0.41486 |
| Mean single sequence MFE | -39.94 |
| Consensus MFE | -35.82 |
| Energy contribution | -37.22 |
| Covariance contribution | 1.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.90 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.580248 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
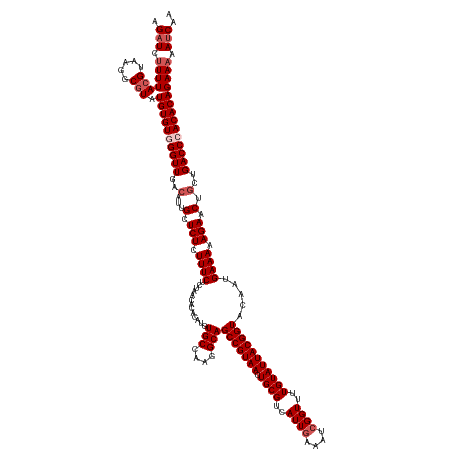
>dm3.chr3L 3060561 148 - 24543557 AGAUCUUUUACGUAAGGCGUAUGUGUGGGUUGACAUUGCUCUCUUUCUCUAACACACAUGUGCCAAGGCAGCCGUAACUGCGUCAUUGAAAUCGGUUUUGUAUUACGGUACAAUGAAAAAGAACUGCUGACCCACACAGAAAAAUCAA .(((.(((((((.....))).((((((((((..((..(.(((.((((.............(((....)))(((((((.((((..((((....))))..))))))))))).....)))).))).)))..)))))))))))))).))).. ( -43.40, z-score = -2.33, R) >droSim1.chr3L 2591869 148 - 22553184 AGAUCUUUUACGUAAGGCGUAUGUGUGGGUUGACAUUGCUCUCUUUCUCUAACACACAUGUGCCAAGGCAGCCGUAACUGCGUCAUUGAAAUCGGUUUUGUAUUACGGUACAAUGAAAAAGAACUGCUGACCCACACAGAAAAAUCAA .(((.(((((((.....))).((((((((((..((..(.(((.((((.............(((....)))(((((((.((((..((((....))))..))))))))))).....)))).))).)))..)))))))))))))).))).. ( -43.40, z-score = -2.33, R) >droSec1.super_2 3062602 148 - 7591821 AGAUCUUUUACGUAAGGCGUAUGUGUGGGUUGACAUUGCUCUCUUUCUCUAACACACAUGUGCCAAGGCAGCCGUAACUGCGUCAUUGAAAUAGGUUUUGUAUUACGGUACAAUGAAAAAGAACUUCUGACCCACACAGAAAAAUCAA .(((.(((((((.....))).((((((((((((....(.(((.((((.............(((....)))(((((((.((((..(((......)))..))))))))))).....)))).))).).)).)))))))))))))).))).. ( -39.10, z-score = -1.49, R) >droYak2.chr3L 15318904 148 + 24197627 AGAUCUUUUACGUAAGGCGUAUGUGUAGGUUGACAUUGCUCUCUUUCUCUAACACACAUGUGCCAAGGCAGCCGUAACUGCGUCAUUGAAAUCGGUUUUGUAUUACGGUACAAUGAAAAAGAACUGCUGACCCACACAGAACUAUCAA .(((....((((.....))))(((((.((((..((..(.(((.((((.............(((....)))(((((((.((((..((((....))))..))))))))))).....)))).))).)))..)))).))))).....))).. ( -36.00, z-score = -0.37, R) >droEre2.scaffold_4784 3076584 148 - 25762168 AGAUCUUUUACGUAAGGCAUAUGUGUAGGUUGACAUUGCUCUCUUUCUCUAACACACAUGUGCCAAGGCAGCCGUAACUGCGUCAUUGAAAUCGGUUUUGUAUUACGGUACAAUGAAAAAGAACCGCUGACCCACACAGAAAUAACAA ...((((((.(((..((((((((((((((..((........))..))).....)))))))))))......(((((((.((((..((((....))))..)))))))))))...))).))))))....(((.......)))......... ( -37.80, z-score = -1.34, R) >consensus AGAUCUUUUACGUAAGGCGUAUGUGUGGGUUGACAUUGCUCUCUUUCUCUAACACACAUGUGCCAAGGCAGCCGUAACUGCGUCAUUGAAAUCGGUUUUGUAUUACGGUACAAUGAAAAAGAACUGCUGACCCACACAGAAAAAUCAA .(((.(((((((.....))).((((((((((..(...(.(((.((((.............(((....)))(((((((.((((..((((....))))..))))))))))).....)))).))).).)..)))))))))))))).))).. (-35.82 = -37.22 + 1.40)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:59:32 2011