| Sequence ID | dm3.chr3L |
|---|---|
| Location | 2,794,528 – 2,794,627 |
| Length | 99 |
| Max. P | 0.999454 |

| Location | 2,794,528 – 2,794,627 |
|---|---|
| Length | 99 |
| Sequences | 12 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 83.15 |
| Shannon entropy | 0.36287 |
| G+C content | 0.59900 |
| Mean single sequence MFE | -37.84 |
| Consensus MFE | -28.51 |
| Energy contribution | -28.35 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.75 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.37 |
| SVM RNA-class probability | 0.932072 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
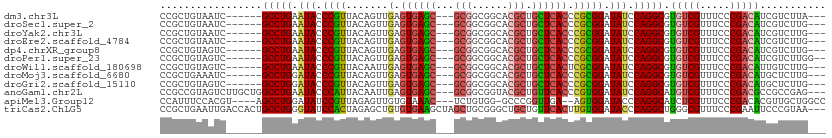
>dm3.chr3L 2794528 99 + 24543557 CCGCUGUAAUC------GCCUGAAUACCCGUUACAGUUGAGUGAGC---GCGGCGGCACGCUGCUCACCCGCGGAUAUCCAGGCGUGUCGUUUCCCGACAUCGUCUUA--- ((((.....((------(.((((((....))).))).)))((((((---(((......))).))))))..))))......((((((((((.....))))).)))))..--- ( -36.10, z-score = -1.49, R) >droSec1.super_2 2815201 99 + 7591821 CCGCUGUAAUC------GCCUGAAUACCCGUUACAGUUGAGUGAGC---GCGGCGGCACGCUGCUCACCCGCGGAUAUCCAGGCGUGUCGUUUCCCGACAUCGUCUUG--- ((((.....((------(.((((((....))).))).)))((((((---(((......))).))))))..))))......((((((((((.....))))).)))))..--- ( -36.10, z-score = -1.49, R) >droYak2.chr3L 15055843 99 - 24197627 CCGCUGUAAUC------GCCUGAAUACCCGUUACAGUUGAGUGAGC---GCGGCGGCACGCUGCUCACCCGCGGAUAUCCAGGCGUGUCGUUUCCCGACAUCGUCUUG--- ((((.....((------(.((((((....))).))).)))((((((---(((......))).))))))..))))......((((((((((.....))))).)))))..--- ( -36.10, z-score = -1.49, R) >droEre2.scaffold_4784 2819776 99 + 25762168 CCGCUGUAAUC------GCCUGAAUACCCGUUACAGUUGAGUGAGC---GCGGCGGCACGCUGCUCACCCGCGGAUAUCCAGGCGUGUCGUUUCCCGACAUCGUCUUG--- ((((.....((------(.((((((....))).))).)))((((((---(((......))).))))))..))))......((((((((((.....))))).)))))..--- ( -36.10, z-score = -1.49, R) >dp4.chrXR_group8 3617655 99 + 9212921 CCGCUGUAGUC------GCCUGAAUACCCGUUACAGUUGAGUGAGC---GCGGCGGCACGCUGCUCACCCGCGGAUAUCCAGGCGUGUCGUUUCCCGACAUCGUCUUG--- ((((.....((------(.((((((....))).))).)))((((((---(((......))).))))))..))))......((((((((((.....))))).)))))..--- ( -36.00, z-score = -1.11, R) >droPer1.super_23 1532460 100 + 1662726 CCGCUGUAGUC------GCCUGAAUACCCGUUACAGUUGAGUGAGC---GCGGCGGCACGCUGCUCACCCGCGGAUAUCCAGGCGUGUCGUUUCCCGACAUCGUCUUGG-- ((((.....((------(.((((((....))).))).)))((((((---(((......))).))))))..))))....((((((((((((.....))))).)).)))))-- ( -37.80, z-score = -1.19, R) >droWil1.scaffold_180698 263797 99 - 11422946 CCGCUGUAGUC------GCCUGAAUACCCGUUACAAUUGAGUGAGC---GCGGCGGCACGCUGCUCACUCGCGGAUAUCCAGGCGUGUCGUUUCCCGACAUUGUCUUG--- ..........(------(((((.(((.((((.......((((((((---(((......))).)))))))))))).))).))))))(((((.....)))))........--- ( -40.21, z-score = -2.85, R) >droMoj3.scaffold_6680 2781977 99 - 24764193 CCGCUGAAAUC------GCCUGGAUACCCGUUACAGUUGAGUGAGC---GCGGCGGCACGCUGCUCACCCGCGGAUAUCCAGGCGUGUCGUUUCCCGACAUGCUCUUG--- ..((.......------(((((((((.((((.......(.((((((---(((......))).)))))).))))).)))))))))((((((.....)))))))).....--- ( -44.21, z-score = -3.53, R) >droGri2.scaffold_15110 19331995 99 + 24565398 CCGCUGUAGUC------GCCUGGAUACCCGUUACAGUUGAGUGAGC---GCGGCGGCACGCUGCUCACCCGCGGAUAUCCAGGCGUGUCGUUUCCCGACAUGCUCUUG--- ..((.......------(((((((((.((((.......(.((((((---(((......))).)))))).))))).)))))))))((((((.....)))))))).....--- ( -44.21, z-score = -3.01, R) >anoGam1.chr2L 5319007 105 - 48795086 CCGCCGUAGUCUUGCUGGCCUGAAUACCCAUUACAAUUGAGUGAGC---GCGGCGGUACGCUGUUCACCCGUGGAUAUCCAGGCAUGUCGUUUCCCGACGCCGCCGAG--- ..((.((..........(((((.(((.((((.......(.((((((---(((......))).)))))).))))).))).)))))..((((.....)))))).))....--- ( -32.01, z-score = 0.10, R) >apiMel3.Group12 2033812 101 + 9182753 CCAUUUCCACGU----AGCCUGGAUAUCCGUUAGAGUUGUGUAAAC---UCUGUGG-GCCCGGUUGA--AGUGGAUACCCAGGCAUCUCGUUUCCCGACACGUUGCUGGCC (((((((.((..----.((((((....))..(((((((.....)))---)))).))-))...)).))--)))))....((((.((...(((........))).)))))).. ( -30.80, z-score = -0.62, R) >triCas2.ChLG5 3248756 108 + 18847211 CCGCUGAAUUGACCACUGCCUGGGUAUCCACUAGAGCUGUGUGAAGCUAGCUGCGGGCUGCUGUUCACUUGUGGAUACCCAGGCUGGGCGUUUCCCGAAUUCCCGUAA--- .((..(((.((.((...((((((((((((((.......(((...(((.(((.....))))))...)))..))))))))))))))..)))).))).))...........--- ( -44.50, z-score = -3.03, R) >consensus CCGCUGUAAUC______GCCUGAAUACCCGUUACAGUUGAGUGAGC___GCGGCGGCACGCUGCUCACCCGCGGAUAUCCAGGCGUGUCGUUUCCCGACAUCGUCUUG___ .................(((((.(((.((((.......(.((((((...(((......))).)))))).))))).))).))))).(((((.....)))))........... (-28.51 = -28.35 + -0.16)
| Location | 2,794,528 – 2,794,627 |
|---|---|
| Length | 99 |
| Sequences | 12 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 83.15 |
| Shannon entropy | 0.36287 |
| G+C content | 0.59900 |
| Mean single sequence MFE | -45.61 |
| Consensus MFE | -36.43 |
| Energy contribution | -36.79 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.24 |
| Mean z-score | -3.94 |
| Structure conservation index | 0.80 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.90 |
| SVM RNA-class probability | 0.999454 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
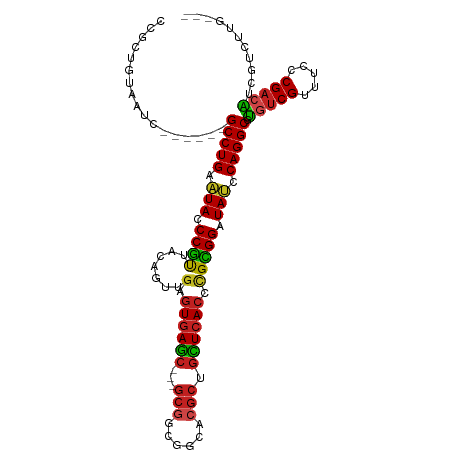
>dm3.chr3L 2794528 99 - 24543557 ---UAAGACGAUGUCGGGAAACGACACGCCUGGAUAUCCGCGGGUGAGCAGCGUGCCGCCGC---GCUCACUCAACUGUAACGGGUAUUCAGGC------GAUUACAGCGG ---.....((.(((((.....)))))((((((((((((((.((((((((.(((......)))---))))))))........)))))))))))))------).......)). ( -46.20, z-score = -4.11, R) >droSec1.super_2 2815201 99 - 7591821 ---CAAGACGAUGUCGGGAAACGACACGCCUGGAUAUCCGCGGGUGAGCAGCGUGCCGCCGC---GCUCACUCAACUGUAACGGGUAUUCAGGC------GAUUACAGCGG ---.....((.(((((.....)))))((((((((((((((.((((((((.(((......)))---))))))))........)))))))))))))------).......)). ( -46.20, z-score = -4.09, R) >droYak2.chr3L 15055843 99 + 24197627 ---CAAGACGAUGUCGGGAAACGACACGCCUGGAUAUCCGCGGGUGAGCAGCGUGCCGCCGC---GCUCACUCAACUGUAACGGGUAUUCAGGC------GAUUACAGCGG ---.....((.(((((.....)))))((((((((((((((.((((((((.(((......)))---))))))))........)))))))))))))------).......)). ( -46.20, z-score = -4.09, R) >droEre2.scaffold_4784 2819776 99 - 25762168 ---CAAGACGAUGUCGGGAAACGACACGCCUGGAUAUCCGCGGGUGAGCAGCGUGCCGCCGC---GCUCACUCAACUGUAACGGGUAUUCAGGC------GAUUACAGCGG ---.....((.(((((.....)))))((((((((((((((.((((((((.(((......)))---))))))))........)))))))))))))------).......)). ( -46.20, z-score = -4.09, R) >dp4.chrXR_group8 3617655 99 - 9212921 ---CAAGACGAUGUCGGGAAACGACACGCCUGGAUAUCCGCGGGUGAGCAGCGUGCCGCCGC---GCUCACUCAACUGUAACGGGUAUUCAGGC------GACUACAGCGG ---.....((.(((((.....)))))((((((((((((((.((((((((.(((......)))---))))))))........)))))))))))))------).......)). ( -46.20, z-score = -3.94, R) >droPer1.super_23 1532460 100 - 1662726 --CCAAGACGAUGUCGGGAAACGACACGCCUGGAUAUCCGCGGGUGAGCAGCGUGCCGCCGC---GCUCACUCAACUGUAACGGGUAUUCAGGC------GACUACAGCGG --......((.(((((.....)))))((((((((((((((.((((((((.(((......)))---))))))))........)))))))))))))------).......)). ( -46.20, z-score = -3.67, R) >droWil1.scaffold_180698 263797 99 + 11422946 ---CAAGACAAUGUCGGGAAACGACACGCCUGGAUAUCCGCGAGUGAGCAGCGUGCCGCCGC---GCUCACUCAAUUGUAACGGGUAUUCAGGC------GACUACAGCGG ---........(((((.....)))))((((((((((((((.((((((((.(((......)))---))))))))........)))))))))))))------).......... ( -46.20, z-score = -4.77, R) >droMoj3.scaffold_6680 2781977 99 + 24764193 ---CAAGAGCAUGUCGGGAAACGACACGCCUGGAUAUCCGCGGGUGAGCAGCGUGCCGCCGC---GCUCACUCAACUGUAACGGGUAUCCAGGC------GAUUUCAGCGG ---.....((.(((((.....)))))((((((((((((((.((((((((.(((......)))---))))))))........)))))))))))))------)......)).. ( -50.80, z-score = -5.02, R) >droGri2.scaffold_15110 19331995 99 - 24565398 ---CAAGAGCAUGUCGGGAAACGACACGCCUGGAUAUCCGCGGGUGAGCAGCGUGCCGCCGC---GCUCACUCAACUGUAACGGGUAUCCAGGC------GACUACAGCGG ---.....((.(((((.....)))))((((((((((((((.((((((((.(((......)))---))))))))........)))))))))))))------)......)).. ( -50.80, z-score = -4.97, R) >anoGam1.chr2L 5319007 105 + 48795086 ---CUCGGCGGCGUCGGGAAACGACAUGCCUGGAUAUCCACGGGUGAACAGCGUACCGCCGC---GCUCACUCAAUUGUAAUGGGUAUUCAGGCCAGCAAGACUACGGCGG ---..((.((..(((.(....).....(((((((((((((.((((((...((((......))---))))))))........)))))))))))))......)))..)).)). ( -40.20, z-score = -1.75, R) >apiMel3.Group12 2033812 101 - 9182753 GGCCAGCAACGUGUCGGGAAACGAGAUGCCUGGGUAUCCACU--UCAACCGGGC-CCACAGA---GUUUACACAACUCUAACGGAUAUCCAGGCU----ACGUGGAAAUGG ..(((.(.(((((((.(....)..)))((((((((((((...--......(...-.)..(((---(((.....))))))...)))))))))))).----)))).)...))) ( -36.90, z-score = -2.67, R) >triCas2.ChLG5 3248756 108 - 18847211 ---UUACGGGAAUUCGGGAAACGCCCAGCCUGGGUAUCCACAAGUGAACAGCAGCCCGCAGCUAGCUUCACACAGCUCUAGUGGAUACCCAGGCAGUGGUCAAUUCAGCGG ---...((.(((((.(((.....))).((((((((((((((..((((..((((((.....))).)))))))..(....).)))))))))))))).......)))))..)). ( -45.20, z-score = -4.06, R) >consensus ___CAAGACGAUGUCGGGAAACGACACGCCUGGAUAUCCGCGGGUGAGCAGCGUGCCGCCGC___GCUCACUCAACUGUAACGGGUAUUCAGGC______GAUUACAGCGG ...........(((((.....))))).(((((((((((((.((((((((.(((......)))...))))))))........)))))))))))))................. (-36.43 = -36.79 + 0.36)


Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:58:30 2011