| Sequence ID | dm3.chr3L |
|---|---|
| Location | 2,105,427 – 2,105,547 |
| Length | 120 |
| Max. P | 0.911459 |

| Location | 2,105,427 – 2,105,547 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.83 |
| Shannon entropy | 0.50676 |
| G+C content | 0.54167 |
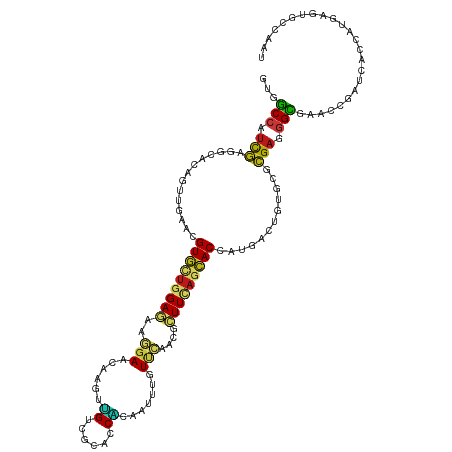
| Mean single sequence MFE | -47.99 |
| Consensus MFE | -14.24 |
| Energy contribution | -13.70 |
| Covariance contribution | -0.54 |
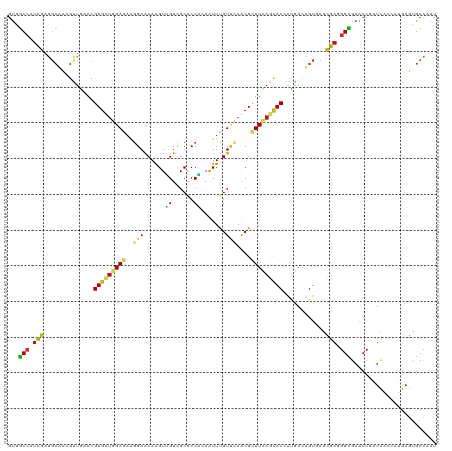
| Combinations/Pair | 1.65 |
| Mean z-score | -2.88 |
| Structure conservation index | 0.30 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.22 |
| SVM RNA-class probability | 0.911459 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
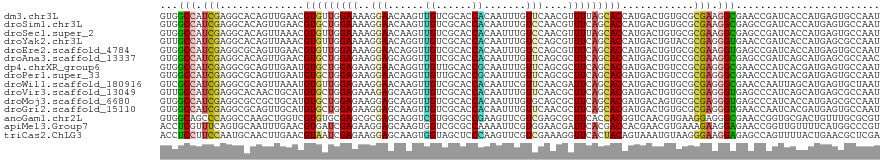
>dm3.chr3L 2105427 120 - 24543557 GUGGCCAUCGAGGCACAGUUGAACGUGUUGGAAAAGGAACAAGUUGUCGCACCACAAUUUGUUCAACGUUUUAGCACCAUGACUGUGCGCGAAGGCGAACCGAUCACCAUGAGUGCCAAU ((((..((((..(((((((((...(((((((((...(((((((((((......)))))))))))....)))))))))..)))))))))((....))....))))..)))).......... ( -45.80, z-score = -3.49, R) >droSim1.chr3L 1669159 120 - 22553184 GUGGCCAUCGAGGCACAGUUGAACGUGCUGGAAAAGGAACAAGUUGUCGCACCACAAUUUGUCCAACGUUUCAGCACCAUGACUGUGCGCGAAGGCGAGCCGAUCACCAUGAGUGCCAAU .(((((.(((..(((((((((...(((((((((..(((.((((((((......)))))))))))....)))))))))..))))))))).))).((.((.....)).))....).)))).. ( -48.30, z-score = -3.54, R) >droSec1.super_2 2136186 120 - 7591821 GUGGCCAUCGAGGCACAGUUAAACGUGUUGGAAAAGGAACAAGUUGUCGCACCACAAUUUGUCCAACGUUUUAGCACCAUGACUGUGCGCGAAGGCGAGCCGAUCACCAUGAGUGCCAAU .(((((.(((..(((((((((...(((((((((..(((.((((((((......)))))))))))....)))))))))..))))))))).))).((.((.....)).))....).)))).. ( -45.10, z-score = -3.43, R) >droYak2.chr3L 2063209 120 - 24197627 GUUGCCAUCGAGGCACAGUUAAACGUGUUGGAAAAGGAACAGGUUGUCGCACCACAAUUUGUCCAGCGUUUCAGCACCAUGACUGUACGCGAGGGUGAACCGAUCACCAUGAGCGCCAAU ...........((((((((((...(((((((((..(((.((((((((......)))))))))))....)))))))))..)))))))..((...(((((.....)))))....)))))... ( -43.20, z-score = -2.77, R) >droEre2.scaffold_4784 2104171 120 - 25762168 GUGGCCAUCGAGGCGCAGUUGAACGUGUUGGAAAAGGAACAGGUUGUCGCACCACAAUUUGUCCAGCGUUUCAGCACCAUGACUGUGCGCGAAGGUGAGCCGAUCACCAUGAGUGCCAAU .(((((.(((..(((((((((...(((((((((..(((.((((((((......)))))))))))....)))))))))..))))))))).))).(((((.....)))))....).)))).. ( -49.00, z-score = -3.19, R) >droAna3.scaffold_13337 22574509 120 + 23293914 GUGGCCAUCGAGGCACAGUUGAACGUGCUGGAGAAGGAGCAGGUUGUCGCACCACAAUUUGUUCAGCGCUUCAGCACCAUGACUGUCCGCGAAGGCGAGCCGAUCAGCAUGAGCGCCAAC .((((......((((((((((...(((((((((...(((((((((((......)))))))))))....)))))))))..))))))).(((....))).))).....((....)))))).. ( -51.40, z-score = -3.23, R) >dp4.chrXR_group6 12015509 120 - 13314419 GUGGCCAUCGAGGCGCAGUUGAAUGUGCUGGAGAAGGAACAGGUUGUUGCACCGCAAUUUGUUCAGCGCUUCAGCACGAUGACUGUCCGCGAGGGCGAACCCAUCACGAUGAGUGCCAAU .(((((((((.((.((((((...((((((((((...(((((((((((......)))))))))))....))))))))))..))))))))..(((((....))).)).)))))...)))).. ( -53.30, z-score = -3.75, R) >droPer1.super_33 36387 120 - 967471 GUGGCCAUCGAGGCGCAGUUGAAUGUGCUGGAGAAGGAACAGGUUGUUGCACCGCAAUUUGUUCAGCGCUUCAGCACGAUGACUGUCCGCGAGGGCGAACCCAUCACGAUGAGUGCCAAU .(((((((((.((.((((((...((((((((((...(((((((((((......)))))))))))....))))))))))..))))))))..(((((....))).)).)))))...)))).. ( -53.30, z-score = -3.75, R) >droWil1.scaffold_180916 1411533 120 + 2700594 GUCGCCAUCGAGGCGCAGUUAAAUGUGUUGGAGAAGGAACAAGUUGUCGCACCACAAUUCGUUCAACGAUUCAGCACCAUGACUGUGCGCGAGGGCGAACCAAUUAGCAUGAGUGCUAAU .(((((.(((..(((((((((...((((((((....((((.((((((......)))))).)))).....))))))))..))))))))).))).)))))....(((((((....))))))) ( -50.80, z-score = -5.16, R) >droVir3.scaffold_13049 13229506 120 + 25233164 GUUGCCAUCGAGGCACAACUGCAUGUGCUGGAGAAAGAGCAAGUUGUCGCACCACAAUUUGUUCAACGCUUCAGCACGAUGACUGUGCGCGAGGGUGAGCCCAUCAGCAUGAGCGCCAAU .(..((.(((..(((((....((((((((((((...(((((((((((......)))))))))))....))))))))).)))..))))).))).))..)((.(((....))).))...... ( -47.60, z-score = -2.40, R) >droMoj3.scaffold_6680 8817136 120 - 24764193 GUGGCCAUCGAGGCGCCGCUGCAUGUGCUGGAGAAGGAGCAGGUUGUCGCACCACAAUUUGUGCAGCGCUUCAGCACGAUGACAGUGCGCGAGGGUGAGCCCAUCACCAUGAGCGCCAAU .((((..(((..((((.((((((((((((((((...(.(((((((((......))))))))).)....))))))))).))).))))))))...(((((.....))))).)))..)))).. ( -52.80, z-score = -1.93, R) >droGri2.scaffold_15110 13121571 120 + 24565398 GUGGCCAUCGAGGCGCAGUUGCAUGUGCUGGAGAAGGAGCAAGUUGUCGCACCACAAUUUGUUCAACGCUUCAGCACGAUGACUGUGCGCGAGGGUGAACCAAUCACGAUGAGUGCCAAU .(((((((((..((((((((...((((((((((...(((((((((((......)))))))))))....))))))))))..))))))))..((.((....))..)).)))))...)))).. ( -55.20, z-score = -4.39, R) >anoGam1.chr2L 1724408 120 + 48795086 GUGGCAGCCCAGGCCAAGCUGGUCGUGUGCGAGCGCGAGCAGGUCGUGGCGCCGAAGUUCGUCGAGCGCUUCACCACGGUCAACGUGAAGGAGGGCGAACCGGUGCGACUGUUUGCGCGU ......((.((((((.....)))).)).))..((((((((((.((...((((((..(((((((.....((((..((((.....))))..))))))))))))))))))))))))))))).. ( -52.80, z-score = -0.12, R) >apiMel3.Group7 5704716 120 - 9974240 ACCUCGUUUCAGUGCAAUUUGAACGUGAUCGAGAAGGAGCAAGUGGUCGCGCCAAAAUUCGUGGAACGAUUCACGACCACGAACGUGAAAGAAGGAGAACCGGUUGUUUUCAUGGCCCGU ..((((..(((((.(.....).)).))).))))..((.((..(((((((.(.......(((.....)))..).)))))))...((((((((..((....)).....)))))))))))).. ( -32.80, z-score = 0.44, R) >triCas2.ChLG3 2720777 120 + 32080666 ACCUCCUUCCAAUGCAACUUGAACGUAAUCGAGAAGGAGCAAGUGGUAGCUCCCAAGUUCGUCGAAAGGUUCACUACAGUAAAUGUAAGGGAAGGAGAGCCAGUUUUACUGAACGCUCGA ...(((((((..((((((((((((.(..((((((((((((........)))))....))).)))).).)))))....)))...))))..)))))))(((((((.....)))...)))).. ( -38.50, z-score = -2.52, R) >consensus GUGGCCAUCGAGGCACAGUUGAACGUGCUGGAGAAGGAACAAGUUGUCGCACCACAAUUUGUUCAACGCUUCAGCACCAUGACUGUGCGCGAGGGCGAACCGAUCACCAUGAGUGCCAAU ...(((.(((..............(((((((((..(((......((......)).......)))....)))))))))............))).)))........................ (-14.24 = -13.70 + -0.54)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:57:04 2011