
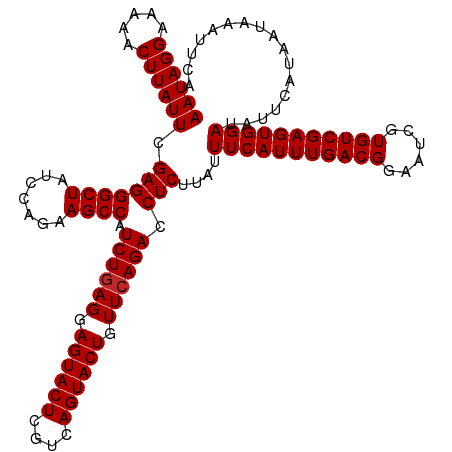
| Sequence ID | dm3.chr3LHet |
|---|---|
| Location | 207,439 – 207,559 |
| Length | 120 |
| Max. P | 0.986598 |

| Location | 207,439 – 207,559 |
|---|---|
| Length | 120 |
| Sequences | 10 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 99.83 |
| Shannon entropy | 0.00391 |
| G+C content | 0.39167 |
| Mean single sequence MFE | -34.08 |
| Consensus MFE | -34.15 |
| Energy contribution | -34.25 |
| Covariance contribution | 0.10 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.39 |
| Structure conservation index | 1.00 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.24 |
| SVM RNA-class probability | 0.986598 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
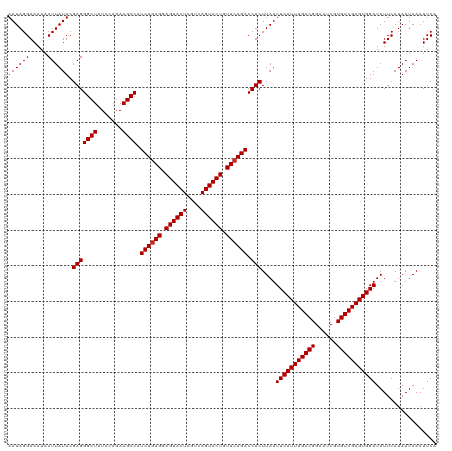
>dm3.chr3LHet 207439 120 + 2555491 AAUAGGAAAAACUUAUUCGAGGGCUAUCCAGAAGCCAUCUGAGGAGUACUCGUCAGUACUGUUCAGACCUCUUAUUUCAUUUGACGGAAUCGUGUCGAGUGGAUAUUCAUAAUAAAUUCA ((((((.....)))))).(((((((.......)))).((((((.((((((....)))))).)))))).)))....(((((((((((......)))))))))))................. ( -34.70, z-score = -2.54, R) >droSec1.super_75 9250 120 - 156374 AAUAGGAAAAACUUAUUCGAGGGCUAUCCAGAAGCCAUCUGAGGAGUACUCGUCAGUACUGUUCAGACCUCUUAUUUCAUUUGACGGAAUCGUGUCGAGUGGAUAUUCAUAAUAAAUUCA ((((((.....)))))).(((((((.......)))).((((((.((((((....)))))).)))))).)))....(((((((((((......)))))))))))................. ( -34.70, z-score = -2.54, R) >droYak2.chrUh 605932 120 + 720513 AAUAGGAAAAACUUAUUCGAGGGCUAUCCAGAAGCCAUCUGAGGAGUACUCGUCAGUACUGUUCAGACCUCUUAUUUCAUUUGACGGAAUCGUGUCGAGUGGAUAUUCAUAAUAAAUUCA ((((((.....)))))).(((((((.......)))).((((((.((((((....)))))).)))))).)))....(((((((((((......)))))))))))................. ( -34.70, z-score = -2.54, R) >droEre2.scaffold_4784 23818004 120 + 25762168 AAUAGGAAAAACUUAUUCGAGGGCUAUCCAGAAGCCAUCUGAGGAGUACUCGUCAGUACUGUUCAGACCUCUUAUUUCAUUUGACGGAAUCGUGUCGAGUGGAUAUUCAUAAUAAAUUCA ((((((.....)))))).(((((((.......)))).((((((.((((((....)))))).)))))).)))....(((((((((((......)))))))))))................. ( -34.70, z-score = -2.54, R) >droAna3.scaffold_13082 1939874 120 + 3919855 AAUAGGAAAAACUUAUUCGAGGGCUAUCCAGAAGCCAUCUCAGGAGUACUCGUCAGUACUGUUCAGACCUCUUAUUUCAUUUGACGGAAUCGUGUCGAGUGGAUAUUCAUAAUAAAUUCA ((((((.....)))))).(((((((.......)))).(((.((.((((((....)))))).)).))).)))....(((((((((((......)))))))))))................. ( -28.50, z-score = -0.97, R) >droPer1.super_6 516575 120 + 6141320 AAUAGGAAAAACUUAUUCGAGGGCUAUCCAGAAGCCAUCUGAGGAGUACUCGUCAGUACUGUUCAGACCUCUUAUUUCAUUUGACGGAAUCGUGUCGAGUGGAUAUUCAUAAUAAAUUCA ((((((.....)))))).(((((((.......)))).((((((.((((((....)))))).)))))).)))....(((((((((((......)))))))))))................. ( -34.70, z-score = -2.54, R) >droWil1.scaffold_181089 692243 120 - 12369635 AAUAGGAAAAACUUAUUCGAGGGCUAUCCAGAAGCCAUCUGAGGAGUACUCGUCAGUACUGUUCAGACCUCUUAUUUCAUUUGACGGAAUCGUGUCGAGUGGAUAUUCAUAAUAAAUUCA ((((((.....)))))).(((((((.......)))).((((((.((((((....)))))).)))))).)))....(((((((((((......)))))))))))................. ( -34.70, z-score = -2.54, R) >droVir3.scaffold_13047 346397 120 + 19223366 AAUAGGAAAAACUUAUUCGAGGGCUAUCCAGAAGCCAUCUGAGGAGUACUCGUCAGUACUGUUCAGACCUCUUAUUUCAUUUGACGGAAUCGUGUCGAGUGGAUAUUCAUAAUAAAUUCA ((((((.....)))))).(((((((.......)))).((((((.((((((....)))))).)))))).)))....(((((((((((......)))))))))))................. ( -34.70, z-score = -2.54, R) >droMoj3.scaffold_6540 33186680 120 - 34148556 AAUAGGAAAAACUUAUUCGAGGGCUAUCCAGAAGCCAUCUGAGGAGUACUCGUCAGUACUGUUCAGACCUCUUAUUUCAUUUGACGGAAUCGUGUCGAGUGGAUAUUCAUAAUAAAUUCA ((((((.....)))))).(((((((.......)))).((((((.((((((....)))))).)))))).)))....(((((((((((......)))))))))))................. ( -34.70, z-score = -2.54, R) >droGri2.scaffold_14906 339384 120 - 14172833 AAUAGGAAAAACUUAUUCGAGGGCUAUCCAGAAGCCAUCUGAGGAGUACUCGUCAGUACUGUUCAGACCUCUUAUUUCAUUUGACGGAAUCGUGUCGAGUGGAUAUUCAUAAUAAAUUCA ((((((.....)))))).(((((((.......)))).((((((.((((((....)))))).)))))).)))....(((((((((((......)))))))))))................. ( -34.70, z-score = -2.54, R) >consensus AAUAGGAAAAACUUAUUCGAGGGCUAUCCAGAAGCCAUCUGAGGAGUACUCGUCAGUACUGUUCAGACCUCUUAUUUCAUUUGACGGAAUCGUGUCGAGUGGAUAUUCAUAAUAAAUUCA ((((((.....)))))).(((((((.......)))).((((((.((((((....)))))).)))))).)))....(((((((((((......)))))))))))................. (-34.15 = -34.25 + 0.10)

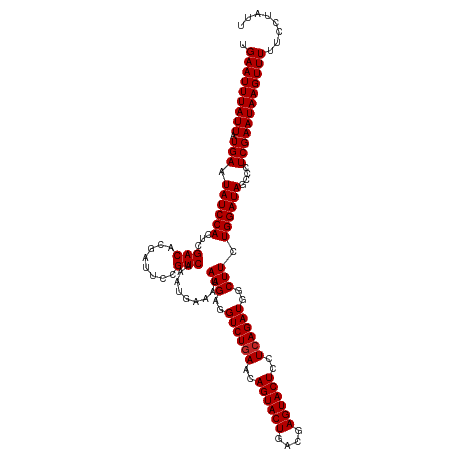
| Location | 207,439 – 207,559 |
|---|---|
| Length | 120 |
| Sequences | 10 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 99.83 |
| Shannon entropy | 0.00391 |
| G+C content | 0.39167 |
| Mean single sequence MFE | -29.24 |
| Consensus MFE | -29.05 |
| Energy contribution | -29.15 |
| Covariance contribution | 0.10 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.99 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.42 |
| SVM RNA-class probability | 0.937830 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
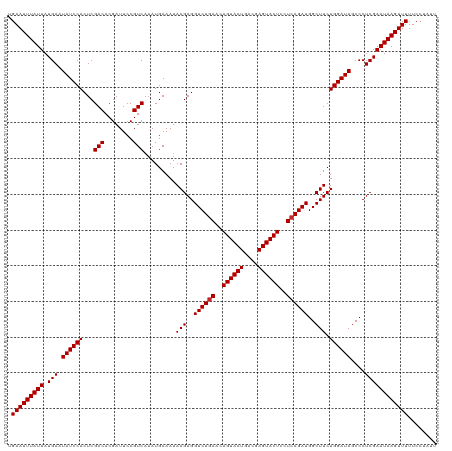
>dm3.chr3LHet 207439 120 - 2555491 UGAAUUUAUUAUGAAUAUCCACUCGACACGAUUCCGUCAAAUGAAAUAAGAGGUCUGAACAGUACUGACGAGUACUCCUCAGAUGGCUUCUGGAUAGCCCUCGAAUAAGUUUUUCCUAUU .(((((((((.(((.((((((...(((........))).........(((..((((((..((((((....))))))..))))))..))).))))))....))))))))))))........ ( -29.60, z-score = -1.92, R) >droSec1.super_75 9250 120 + 156374 UGAAUUUAUUAUGAAUAUCCACUCGACACGAUUCCGUCAAAUGAAAUAAGAGGUCUGAACAGUACUGACGAGUACUCCUCAGAUGGCUUCUGGAUAGCCCUCGAAUAAGUUUUUCCUAUU .(((((((((.(((.((((((...(((........))).........(((..((((((..((((((....))))))..))))))..))).))))))....))))))))))))........ ( -29.60, z-score = -1.92, R) >droYak2.chrUh 605932 120 - 720513 UGAAUUUAUUAUGAAUAUCCACUCGACACGAUUCCGUCAAAUGAAAUAAGAGGUCUGAACAGUACUGACGAGUACUCCUCAGAUGGCUUCUGGAUAGCCCUCGAAUAAGUUUUUCCUAUU .(((((((((.(((.((((((...(((........))).........(((..((((((..((((((....))))))..))))))..))).))))))....))))))))))))........ ( -29.60, z-score = -1.92, R) >droEre2.scaffold_4784 23818004 120 - 25762168 UGAAUUUAUUAUGAAUAUCCACUCGACACGAUUCCGUCAAAUGAAAUAAGAGGUCUGAACAGUACUGACGAGUACUCCUCAGAUGGCUUCUGGAUAGCCCUCGAAUAAGUUUUUCCUAUU .(((((((((.(((.((((((...(((........))).........(((..((((((..((((((....))))))..))))))..))).))))))....))))))))))))........ ( -29.60, z-score = -1.92, R) >droAna3.scaffold_13082 1939874 120 - 3919855 UGAAUUUAUUAUGAAUAUCCACUCGACACGAUUCCGUCAAAUGAAAUAAGAGGUCUGAACAGUACUGACGAGUACUCCUGAGAUGGCUUCUGGAUAGCCCUCGAAUAAGUUUUUCCUAUU .(((((((((.(((.(((((....(((........)))..........(((((((.....((((((....))))))........))))))))))))....))))))))))))........ ( -26.02, z-score = -0.79, R) >droPer1.super_6 516575 120 - 6141320 UGAAUUUAUUAUGAAUAUCCACUCGACACGAUUCCGUCAAAUGAAAUAAGAGGUCUGAACAGUACUGACGAGUACUCCUCAGAUGGCUUCUGGAUAGCCCUCGAAUAAGUUUUUCCUAUU .(((((((((.(((.((((((...(((........))).........(((..((((((..((((((....))))))..))))))..))).))))))....))))))))))))........ ( -29.60, z-score = -1.92, R) >droWil1.scaffold_181089 692243 120 + 12369635 UGAAUUUAUUAUGAAUAUCCACUCGACACGAUUCCGUCAAAUGAAAUAAGAGGUCUGAACAGUACUGACGAGUACUCCUCAGAUGGCUUCUGGAUAGCCCUCGAAUAAGUUUUUCCUAUU .(((((((((.(((.((((((...(((........))).........(((..((((((..((((((....))))))..))))))..))).))))))....))))))))))))........ ( -29.60, z-score = -1.92, R) >droVir3.scaffold_13047 346397 120 - 19223366 UGAAUUUAUUAUGAAUAUCCACUCGACACGAUUCCGUCAAAUGAAAUAAGAGGUCUGAACAGUACUGACGAGUACUCCUCAGAUGGCUUCUGGAUAGCCCUCGAAUAAGUUUUUCCUAUU .(((((((((.(((.((((((...(((........))).........(((..((((((..((((((....))))))..))))))..))).))))))....))))))))))))........ ( -29.60, z-score = -1.92, R) >droMoj3.scaffold_6540 33186680 120 + 34148556 UGAAUUUAUUAUGAAUAUCCACUCGACACGAUUCCGUCAAAUGAAAUAAGAGGUCUGAACAGUACUGACGAGUACUCCUCAGAUGGCUUCUGGAUAGCCCUCGAAUAAGUUUUUCCUAUU .(((((((((.(((.((((((...(((........))).........(((..((((((..((((((....))))))..))))))..))).))))))....))))))))))))........ ( -29.60, z-score = -1.92, R) >droGri2.scaffold_14906 339384 120 + 14172833 UGAAUUUAUUAUGAAUAUCCACUCGACACGAUUCCGUCAAAUGAAAUAAGAGGUCUGAACAGUACUGACGAGUACUCCUCAGAUGGCUUCUGGAUAGCCCUCGAAUAAGUUUUUCCUAUU .(((((((((.(((.((((((...(((........))).........(((..((((((..((((((....))))))..))))))..))).))))))....))))))))))))........ ( -29.60, z-score = -1.92, R) >consensus UGAAUUUAUUAUGAAUAUCCACUCGACACGAUUCCGUCAAAUGAAAUAAGAGGUCUGAACAGUACUGACGAGUACUCCUCAGAUGGCUUCUGGAUAGCCCUCGAAUAAGUUUUUCCUAUU .(((((((((.(((.((((((...(((........))).........(((..((((((..((((((....))))))..))))))..))).))))))....))))))))))))........ (-29.05 = -29.15 + 0.10)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:50:11 2011