
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 20,982,183 – 20,982,291 |
| Length | 108 |
| Max. P | 0.994351 |

| Location | 20,982,183 – 20,982,291 |
|---|---|
| Length | 108 |
| Sequences | 11 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 82.54 |
| Shannon entropy | 0.38447 |
| G+C content | 0.66449 |
| Mean single sequence MFE | -53.31 |
| Consensus MFE | -33.90 |
| Energy contribution | -33.92 |
| Covariance contribution | 0.02 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.64 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.98 |
| SVM RNA-class probability | 0.865612 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
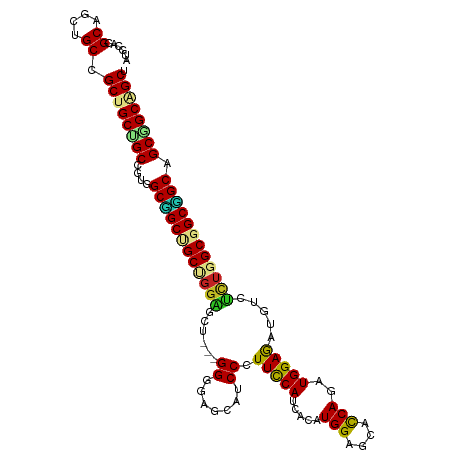
>dm3.chr2R 20982183 108 + 21146708 AGCUGCCGCUGCAGCCGCCAAAGACAUCUCCAUCUGGUGCUCCAUAUGAUGGAAGGGAUGCUCCCC---AGAUCCAGCAGCCGCCACGGCAGCAGCAGCAGCUGCGUGCAU ((((((.(((((.((((...........(((((((((....)))...))))))..((.((((....---......)))).))....)))).))))).))))))........ ( -42.60, z-score = -0.55, R) >droSim1.chr2R 19433990 108 + 19596830 AGCUGCCGCGGCAGCCGCCAAAGACAUCUCCAUCUGGUGCUCCAUAUGAUGGAAGGGAUGCUCCCC---AGAUCCAGCAGCCGCCACGGCAGCAGCCGCAGCUGCGUGCAU .(((((((.(((.((.((..........(((((((((....)))...)))))).(((......)))---.......)).)).))).))))))).(((((....))).)).. ( -48.80, z-score = -2.13, R) >droSec1.super_23 838426 108 + 989336 AGCUGCCGCGGCAGCCGCCAAAGACAUCUCCAUCUGGUGCUCCAUAUGAUGGAAGGGAUGCUCCCC---AGAUCCAGCAGCCGCCACGGCAGCAGCCGCAGCUGCGUGCAU .(((((((.(((.((.((..........(((((((((....)))...)))))).(((......)))---.......)).)).))).))))))).(((((....))).)).. ( -48.80, z-score = -2.13, R) >droYak2.chr2R 20952212 108 + 21139217 AGCGGCCGCUGCAGCCGCCAGAGACAUCUCCAUCUGGUGCUCCAUAUGAUGGAAGGGGUGCUCCCC---AGAUCCAGCAGCCGCCACGGCAGCAGCCGCAGCUGCGUGCAU .(.(((.((((((((.((((((..........)))))))))........((((.((((....))))---...))))))))).))).).((((((((....))))).))).. ( -49.60, z-score = -1.56, R) >droEre2.scaffold_4845 22390850 108 + 22589142 AGCUGCCGCUGCAGCCGCCAAAGACAUCUCCAUCUGGUGCUCCAUAUGAUGGAAGGGGUGCUCACC---AGAUCCAGCAGCCGCCACGGCAGCAGCCGCAGCUGCGUGCAU ((((((.(((((.((((...........(((((((((....)))...)))))).((((((((....---......)))).)).)).)))).))))).))))))........ ( -45.10, z-score = -0.80, R) >droAna3.scaffold_13266 12289443 105 + 19884421 UGCUGCAGCGGCCGCGGCCAGGGACAUCUCCAUCUGGUGCUCCAUGUGGUGGAAGGGAUGGUCGCC---AGCC---GCGGCUGCUGCUGCAGCGGCGGCGGCGGCGUGCAU ...((((((.((((((((((....(.(((((((.(((....))).)))).))).)...))))))).---.(((---((.(((((....))))).)))))))).)).)))). ( -60.80, z-score = -1.95, R) >dp4.chr3 10092220 108 + 19779522 GGCCGCAGCCGCCGCCGCCAGCGACAUCUCCAUCUGGUGCUCCAUGUGGUGGAAGGGAUGCUCCCC---AGCGCCGGCGGCAGCCACGGCCGCAGCGGCUGCGGCGUGCAU .(((((((((((.((((((.(((.(.(((((((.(((....))).)))).))).(((......)))---.)))).)))))).((....))....)))))))))))...... ( -62.90, z-score = -2.24, R) >droPer1.super_4 5421305 108 + 7162766 GGCCGCAGCCGCCGCCGCCAGCGACAUCUCCAUCUGGUGCUCCAUGUGGUGGAAGGGAUGCUCCCC---AGCGCCGGCGGCAGCCACGGCCGCAGCGGCUGCGGCGUGCAU .(((((((((((.((((((.(((.(.(((((((.(((....))).)))).))).(((......)))---.)))).)))))).((....))....)))))))))))...... ( -62.90, z-score = -2.24, R) >droWil1.scaffold_180700 5236200 111 - 6630534 UGCUGCAGCAGCUGCCGCCAGUGACAUUUCCAUCUGAUGUUCCAUGUGAUGAAACGGAUGCUCGCCGCUUGCUCCAGCGGCAGCAACGGCUGCUGCUGCUGCAGCAUGCAU ((((((((((((.((.(((.(..(((((.......)))))..)...............((((.((((((......))))))))))..))).)).))))))))))))..... ( -55.30, z-score = -3.41, R) >droVir3.scaffold_12875 18288345 108 - 20611582 CGCUGCGGCGGCCGCGGCCAGCGACAUUUCCAUUUGGUGCUCCAUGUGAUGAAACGGAUUCUCACC---GGCGCCUGCUGCGGCUACGGCGGCAGCGGCCGCGGCGUGCAU (((....)))(((((((((................((((((....((((.(((.....))))))).---)))))).(((((.((....)).))))))))))))))...... ( -56.40, z-score = -1.77, R) >droMoj3.scaffold_6496 22925383 111 + 26866924 CGCUGCUGCUGCCGCGGCCAGCGACAUUUCCAUUUGAUGCUCCAUGUGAUGGAACGGAUUCUCGCCAGCGGCGCCUGCAGCGGCAACGGCGGCAGCGGCUGCCGCAUGCAU ((((((.((.((((((((.((......(((((((..(((...)))..)))))))......)).))).)))))))..))))))(((.(((((((....)))))))..))).. ( -53.20, z-score = -1.06, R) >consensus AGCUGCCGCGGCAGCCGCCAGAGACAUCUCCAUCUGGUGCUCCAUGUGAUGGAAGGGAUGCUCCCC___AGCUCCAGCAGCCGCCACGGCAGCAGCGGCAGCUGCGUGCAU .((.((.(((((.((.(((...(((((((((((((((....)))...))))))...)))).)).............((....))...))).)).))))).)).))...... (-33.90 = -33.92 + 0.02)



| Location | 20,982,183 – 20,982,291 |
|---|---|
| Length | 108 |
| Sequences | 11 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 82.54 |
| Shannon entropy | 0.38447 |
| G+C content | 0.66449 |
| Mean single sequence MFE | -58.55 |
| Consensus MFE | -38.62 |
| Energy contribution | -37.82 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.44 |
| Mean z-score | -2.76 |
| Structure conservation index | 0.66 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.69 |
| SVM RNA-class probability | 0.994351 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 20982183 108 - 21146708 AUGCACGCAGCUGCUGCUGCUGCCGUGGCGGCUGCUGGAUCU---GGGGAGCAUCCCUUCCAUCAUAUGGAGCACCAGAUGGAGAUGUCUUUGGCGGCUGCAGCGGCAGCU ..(((.((....))))).((((((((.((((((((..((...---((((.....))))((((((...(((....)))))))))......))..)))))))).)))))))). ( -55.50, z-score = -2.28, R) >droSim1.chr2R 19433990 108 - 19596830 AUGCACGCAGCUGCGGCUGCUGCCGUGGCGGCUGCUGGAUCU---GGGGAGCAUCCCUUCCAUCAUAUGGAGCACCAGAUGGAGAUGUCUUUGGCGGCUGCCGCGGCAGCU ..((.(((....))))).(((((((((((((((((..((...---((((.....))))((((((...(((....)))))))))......))..))))))))))))))))). ( -62.80, z-score = -4.12, R) >droSec1.super_23 838426 108 - 989336 AUGCACGCAGCUGCGGCUGCUGCCGUGGCGGCUGCUGGAUCU---GGGGAGCAUCCCUUCCAUCAUAUGGAGCACCAGAUGGAGAUGUCUUUGGCGGCUGCCGCGGCAGCU ..((.(((....))))).(((((((((((((((((..((...---((((.....))))((((((...(((....)))))))))......))..))))))))))))))))). ( -62.80, z-score = -4.12, R) >droYak2.chr2R 20952212 108 - 21139217 AUGCACGCAGCUGCGGCUGCUGCCGUGGCGGCUGCUGGAUCU---GGGGAGCACCCCUUCCAUCAUAUGGAGCACCAGAUGGAGAUGUCUCUGGCGGCUGCAGCGGCCGCU ..((..(((((....))))).(((((.((((((((..((...---((((....))))(((((((...(((....)))))))))).....))..)))))))).))))).)). ( -57.50, z-score = -2.33, R) >droEre2.scaffold_4845 22390850 108 - 22589142 AUGCACGCAGCUGCGGCUGCUGCCGUGGCGGCUGCUGGAUCU---GGUGAGCACCCCUUCCAUCAUAUGGAGCACCAGAUGGAGAUGUCUUUGGCGGCUGCAGCGGCAGCU ..((.(((....))))).((((((((.(((((((((..((((---((((........((((((...))))))))))))))((((....))))))))))))).)))))))). ( -55.30, z-score = -2.23, R) >droAna3.scaffold_13266 12289443 105 - 19884421 AUGCACGCCGCCGCCGCCGCUGCAGCAGCAGCCGC---GGCU---GGCGACCAUCCCUUCCACCACAUGGAGCACCAGAUGGAGAUGUCCCUGGCCGCGGCCGCUGCAGCA .((((.((.((....)).))))))(((((.(((((---(((.---.(.(((.(((...((((.....(((....)))..)))))))))).)..)))))))).))))).... ( -53.40, z-score = -2.87, R) >dp4.chr3 10092220 108 - 19779522 AUGCACGCCGCAGCCGCUGCGGCCGUGGCUGCCGCCGGCGCU---GGGGAGCAUCCCUUCCACCACAUGGAGCACCAGAUGGAGAUGUCGCUGGCGGCGGCGGCUGCGGCC ......((((((((((((..(((....)))(((((((((((.---((((.....))))....(((..(((....)))..)))....).)))))))))))))))))))))). ( -66.20, z-score = -3.07, R) >droPer1.super_4 5421305 108 - 7162766 AUGCACGCCGCAGCCGCUGCGGCCGUGGCUGCCGCCGGCGCU---GGGGAGCAUCCCUUCCACCACAUGGAGCACCAGAUGGAGAUGUCGCUGGCGGCGGCGGCUGCGGCC ......((((((((((((..(((....)))(((((((((((.---((((.....))))....(((..(((....)))..)))....).)))))))))))))))))))))). ( -66.20, z-score = -3.07, R) >droWil1.scaffold_180700 5236200 111 + 6630534 AUGCAUGCUGCAGCAGCAGCAGCCGUUGCUGCCGCUGGAGCAAGCGGCGAGCAUCCGUUUCAUCACAUGGAACAUCAGAUGGAAAUGUCACUGGCGGCAGCUGCUGCAGCA .....((((((((((((.((.(((..((((((((((......)))))).))))...(((((((...)))))))....(((......)))...))).)).)))))))))))) ( -56.10, z-score = -2.99, R) >droVir3.scaffold_12875 18288345 108 + 20611582 AUGCACGCCGCGGCCGCUGCCGCCGUAGCCGCAGCAGGCGCC---GGUGAGAAUCCGUUUCAUCACAUGGAGCACCAAAUGGAAAUGUCGCUGGCCGCGGCCGCCGCAGCG ..((..((.(((((((((((.((....)).)))))..(((((---(((((.(....(((((((...))))))).((....))...).))))))).))))))))).)).)). ( -53.10, z-score = -1.77, R) >droMoj3.scaffold_6496 22925383 111 - 26866924 AUGCAUGCGGCAGCCGCUGCCGCCGUUGCCGCUGCAGGCGCCGCUGGCGAGAAUCCGUUCCAUCACAUGGAGCAUCAAAUGGAAAUGUCGCUGGCCGCGGCAGCAGCAGCG ......(((((((...)))))))((((((.(((((..(((..((..((((.(.((((((((((...))))))).......)))..).))))..))))).))))).)))))) ( -55.11, z-score = -1.53, R) >consensus AUGCACGCAGCUGCCGCUGCUGCCGUGGCGGCUGCUGGAGCU___GGGGAGCAUCCCUUCCAUCACAUGGAGCACCAGAUGGAGAUGUCUCUGGCGGCGGCAGCGGCAGCU ......((....)).((((((((....((((((((((((......((.......)).(((((.....(((....)))..))))).....)))))))))))).)))))))). (-38.62 = -37.82 + -0.80)


Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:49:49 2011