
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 20,587,304 – 20,587,405 |
| Length | 101 |
| Max. P | 0.993353 |

| Location | 20,587,304 – 20,587,405 |
|---|---|
| Length | 101 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 67.91 |
| Shannon entropy | 0.65037 |
| G+C content | 0.71426 |
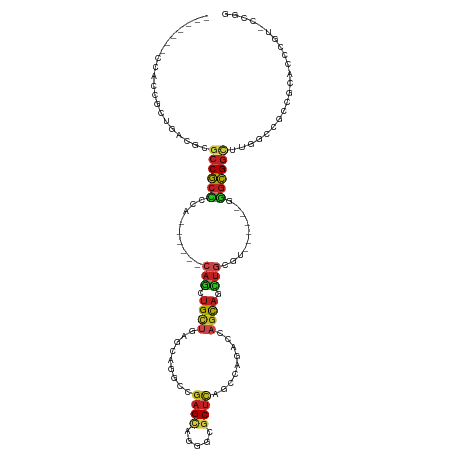
| Mean single sequence MFE | -47.27 |
| Consensus MFE | -16.67 |
| Energy contribution | -17.08 |
| Covariance contribution | 0.41 |
| Combinations/Pair | 1.59 |
| Mean z-score | -1.52 |
| Structure conservation index | 0.35 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.36 |
| SVM RNA-class probability | 0.661138 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
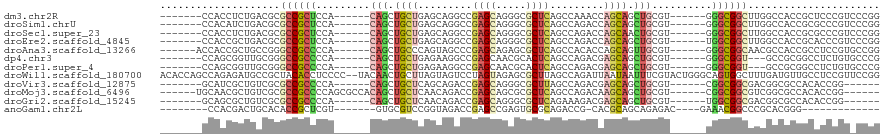
>dm3.chr2R 20587304 101 + 21146708 -------CCACCUCUGACGCGCCGCUCCA------CAGCUGCUGAGCAGGCCGAGCAGGGCGCUCAGCCAAACCAGCAGCUGCGU------GGGCGGCUUGGCCACCGCUCCCGUCCCGG -------...((...((((.((((((((.------(((((((((....(((.((((.....)))).)))....))))))))).).------)))))))..(((....)))..))))..)) ( -49.90, z-score = -1.49, R) >droSim1.chrU 6559 101 - 15797150 -------CCACAUCUGACGCGCCGCUCCA------CAGCUGCUGAGCAGGCCGAGCAGGGCGCUCAGCCAGACCAGCAGCUGCGU------GGGCGGCUUGGCCACCGCGCCCGUCCCGG -------........((((.((((((((.------(((((((((..(.(((.((((.....)))).))).)..))))))))).).------)))))))..(((......))))))).... ( -53.00, z-score = -1.76, R) >droSec1.super_23 452654 101 + 989336 -------CCACCUCUGACGCGCCGCUCCA------CAGCUGCUGAGCAGGCCGAGCAGGGCGCUCAGCCAGACCAGCAACUGCGU------GGGCGGCUUGGCCACCGCGCCCGUCCCGG -------...((...((((.((((((((.------(((.(((((..(.(((.((((.....)))).))).)..))))).))).).------)))))))..(((......)))))))..)) ( -47.10, z-score = -0.48, R) >droEre2.scaffold_4845 21956589 101 + 22589142 -------CCACCGCUGACGCGCCGCUCCA------CAGCUGCUGAGCAGGCCGAGCAGGGCGCUCAGCCAGACCAGCAGCUGCGU------UGGCGGCUUGGCCACCGCACCCGUCCCGG -------...(((..((((.((((((.(.------(((((((((..(.(((.((((.....)))).))).)..))))))))).).------.))))))...((....))...)))).))) ( -49.60, z-score = -1.22, R) >droAna3.scaffold_13266 7180008 102 + 19884421 ------ACCACCGCUGCCGGGCCGCCCCA------CAGCUGCCCAGUAGCCCGAGCAGAGCGCUCAGCCACACCAGCAGUUGCGU------GGGCGGCAACGCCACCGCCUCCGUGCCGG ------....(((..(((((((((((((.------(((((((...((.((..((((.....)))).)).))....))))))).).------)))))))...((....))..))).))))) ( -46.00, z-score = -1.50, R) >dp4.chr3 3940751 98 - 19779522 -------CCAGCGGUUGCGGGCCGCCCCA------CAGCUGCUGAGAAGGCCGAGCAACGCACUCAGCCAGACGAGCAGCUGCGU------GGGCGGU---GCCGCGGCCUCUGUGCCCG -------.(((.((((((((((((((((.------((((((((.....(((.(((.......))).))).....)))))))).).------)))))))---.)))))))).)))...... ( -57.80, z-score = -3.15, R) >droPer1.super_4 923459 98 + 7162766 -------CCAGCGGUUGCGGGCCGCCCCA------CAGCUGCUGAGAAGGCCGAGCAACGCACUCAGCCAGACGAGCAGCUGCGU------GGGCGGU---GCCGCGGCCUCUGUGCCCG -------.(((.((((((((((((((((.------((((((((.....(((.(((.......))).))).....)))))))).).------)))))))---.)))))))).)))...... ( -57.80, z-score = -3.15, R) >droWil1.scaffold_180700 2247385 118 - 6630534 ACACCAGCCAGAGAUGCCGCUACACCUCCCC--UACAACUGCUUAGUAGUCCUAGUAGAGCGCUUAGCCAGAUUAAUAAUUUCGUACUGGGCAGUGGCUUUGAUGUUGCCUCCGUUCCGG ....((((.(..((.((((((....(((..(--((..(((((...)))))..)))..))).((((((...((.........))...)))))))))))).))..)))))...(((...))) ( -28.60, z-score = 0.34, R) >droVir3.scaffold_12875 1089494 95 + 20611582 -------GCAUCGCUGUCGCGCCGCCCCA------CAGCUGCUCAGCAGACCGAGCAGGGCGCUUAGCCAGACGAGCAGCUGCGU------CGGCGGCGACGGCGCCACACCGG------ -------....(((((((..((((((.(.------(((((((((.((.......))..(((.....)))....))))))))).).------.))))))))))))).........------ ( -48.90, z-score = -2.09, R) >droMoj3.scaffold_6496 4141237 102 + 26866924 ------UGCAACGCUGUCGCGCCGCCCCAGCGCCACAGCUGCUCAACAGACCGAGCAGCGCGCUCAGCCAGACAAGCAGCUGCGU------CGGCGGCGUCGGCGCCACACCGG------ ------.....((.(((.(((((((.(((((((....(((((((........))))))))))))..(((.(((..((....))))------)))))).).)))))).))).)).------ ( -48.60, z-score = -1.59, R) >droGri2.scaffold_15245 8432503 95 - 18325388 -------GCAGCGCUGUCGCGCCGCCCCA------CAGCUGCUCAACAGACCGAGCAGGGCGCUCAGAAAGACGAGCAGCUGCGU------UGGCGGCGACGGCGCCACACCGG------ -------...((((((((..((((((.(.------(((((((((..(...(.((((.....)))).)...)..))))))))).).------.))))))))))))))........------ ( -50.00, z-score = -2.78, R) >anoGam1.chr2L 9569011 87 - 48795086 --------CCACGACUGCACACCGCUCGU-------GUGCGUCCGGUAGACCGAGCCGAGUGCGCAGACCG-CACGCAGCAGAGAC----GAAACGGCCCGCACGGG------------- --------...((...((((((.....))-------))))((((((....))).((.(.(((((.....))-))).).))...)))----....)).(((....)))------------- ( -30.00, z-score = 0.61, R) >consensus _______CCACCGCUGACGCGCCGCCCCA______CAGCUGCUGAGCAGGCCGAGCAGGGCGCUCAGCCAGACCAGCAGCUGCGU______GGGCGGCUUGGCCGCCGCACCCGU_CCGG ....................((((((.........(((.((((.........((((.....)))).........)))).)))..........))))))...................... (-16.67 = -17.08 + 0.41)
| Location | 20,587,304 – 20,587,405 |
|---|---|
| Length | 101 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 67.91 |
| Shannon entropy | 0.65037 |
| G+C content | 0.71426 |
| Mean single sequence MFE | -56.66 |
| Consensus MFE | -24.24 |
| Energy contribution | -24.60 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.57 |
| Mean z-score | -2.23 |
| Structure conservation index | 0.43 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.61 |
| SVM RNA-class probability | 0.993353 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
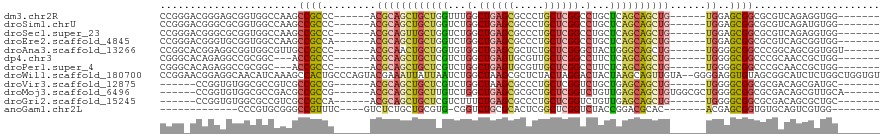
>dm3.chr2R 20587304 101 - 21146708 CCGGGACGGGAGCGGUGGCCAAGCCGCCC------ACGCAGCUGCUGGUUUGGCUGAGCGCCCUGCUCGGCCUGCUCAGCAGCUG------UGGAGCGGCGCGUCAGAGGUGG------- ((..((((.(.((((((((...)))))).------.(((((((((((((..((((((((.....)))))))).)).)))))))))------))..))..).))))...))...------- ( -60.80, z-score = -2.44, R) >droSim1.chrU 6559 101 + 15797150 CCGGGACGGGCGCGGUGGCCAAGCCGCCC------ACGCAGCUGCUGGUCUGGCUGAGCGCCCUGCUCGGCCUGCUCAGCAGCUG------UGGAGCGGCGCGUCAGAUGUGG------- ....(((((.(((((((((...)))))).------.(((((((((((..(.((((((((.....)))))))).)..)))))))))------))..))).).))))........------- ( -61.50, z-score = -2.30, R) >droSec1.super_23 452654 101 - 989336 CCGGGACGGGCGCGGUGGCCAAGCCGCCC------ACGCAGUUGCUGGUCUGGCUGAGCGCCCUGCUCGGCCUGCUCAGCAGCUG------UGGAGCGGCGCGUCAGAGGUGG------- ((..(((((.(((((((((...)))))).------.(((((((((((..(.((((((((.....)))))))).)..)))))))))------))..))).).))))...))...------- ( -61.20, z-score = -2.28, R) >droEre2.scaffold_4845 21956589 101 - 22589142 CCGGGACGGGUGCGGUGGCCAAGCCGCCA------ACGCAGCUGCUGGUCUGGCUGAGCGCCCUGCUCGGCCUGCUCAGCAGCUG------UGGAGCGGCGCGUCAGCGGUGG------- (((.(((((.(((((((((...)))))).------.(((((((((((..(.((((((((.....)))))))).)..)))))))))------))..))).).))))..)))...------- ( -64.10, z-score = -2.92, R) >droAna3.scaffold_13266 7180008 102 - 19884421 CCGGCACGGAGGCGGUGGCGUUGCCGCCC------ACGCAACUGCUGGUGUGGCUGAGCGCUCUGCUCGGGCUACUGGGCAGCUG------UGGGGCGGCCCGGCAGCGGUGGU------ (((((.(((.((((....))))(((((((------.((((.(((((.(.((((((((((.....)))).))))))).))))).))------))))))))))))))..)))....------ ( -55.10, z-score = -0.29, R) >dp4.chr3 3940751 98 + 19779522 CGGGCACAGAGGCCGCGGC---ACCGCCC------ACGCAGCUGCUCGUCUGGCUGAGUGCGUUGCUCGGCCUUCUCAGCAGCUG------UGGGGCGGCCCGCAACCGCUGG------- ......(((.((..((((.---.((((((------.((((((((((.....((((((((.....)))))))).....))))))))------)))))))).))))..)).))).------- ( -61.00, z-score = -3.44, R) >droPer1.super_4 923459 98 - 7162766 CGGGCACAGAGGCCGCGGC---ACCGCCC------ACGCAGCUGCUCGUCUGGCUGAGUGCGUUGCUCGGCCUUCUCAGCAGCUG------UGGGGCGGCCCGCAACCGCUGG------- ......(((.((..((((.---.((((((------.((((((((((.....((((((((.....)))))))).....))))))))------)))))))).))))..)).))).------- ( -61.00, z-score = -3.44, R) >droWil1.scaffold_180700 2247385 118 + 6630534 CCGGAACGGAGGCAACAUCAAAGCCACUGCCCAGUACGAAAUUAUUAAUCUGGCUAAGCGCUCUACUAGGACUACUAAGCAGUUGUA--GGGGAGGUGUAGCGGCAUCUCUGGCUGGUGU ((((..(((((((.........)))..(((((((...............)))((((.((.(((..(((.((((.......)))).))--)..))))).))))))))..)))).))))... ( -35.66, z-score = 0.09, R) >droVir3.scaffold_12875 1089494 95 - 20611582 ------CCGGUGUGGCGCCGUCGCCGCCG------ACGCAGCUGCUCGUCUGGCUAAGCGCCCUGCUCGGUCUGCUGAGCAGCUG------UGGGGCGGCGCGACAGCGAUGC------- ------...((((.((..((.(((((((.------.((((((((((((.(.(((..(((.....)))..))).).))))))))))------)).)))))))))...)).))))------- ( -55.80, z-score = -2.18, R) >droMoj3.scaffold_6496 4141237 102 - 26866924 ------CCGGUGUGGCGCCGACGCCGCCG------ACGCAGCUGCUUGUCUGGCUGAGCGCGCUGCUCGGUCUGUUGAGCAGCUGUGGCGCUGGGGCGGCGCGACAGCGUUGCA------ ------.((.(((.((((((.(.(((.((------.((((((((((((...((((((((.....))))))))...)))))))))))).)).))).))))))).))).)).....------ ( -63.20, z-score = -2.64, R) >droGri2.scaffold_15245 8432503 95 + 18325388 ------CCGGUGUGGCGCCGUCGCCGCCA------ACGCAGCUGCUCGUCUUUCUGAGCGCCCUGCUCGGUCUGUUGAGCAGCUG------UGGGGCGGCGCGACAGCGCUGC------- ------.....(..((((.(((((((((.------.((((((((((((.(...((((((.....))))))...).))))))))))------)).))))))..))).))))..)------- ( -59.30, z-score = -4.00, R) >anoGam1.chr2L 9569011 87 + 48795086 -------------CCCGUGCGGGCCGUUUC----GUCUCUGCUGCGUG-CGGUCUGCGCACUCGGCUCGGUCUACCGGACGCAC-------ACGAGCGGUGUGCAGUCGUGG-------- -------------...(((((((((((..(----((.......))).)-)))))))))).(.((((((((....)))...((((-------((.....))))))))))).).-------- ( -41.30, z-score = -0.95, R) >consensus CCGG_ACGGGGGCGGCGGCCAAGCCGCCC______ACGCAGCUGCUGGUCUGGCUGAGCGCCCUGCUCGGCCUGCUCAGCAGCUG______UGGGGCGGCGCGUCAGCGGUGG_______ .......................((((...........((((((((((...(.((((((.....)))))).)...))))))))))..........))))..................... (-24.24 = -24.60 + 0.36)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:48:59 2011