
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 20,256,009 – 20,256,103 |
| Length | 94 |
| Max. P | 0.622956 |

| Location | 20,256,009 – 20,256,103 |
|---|---|
| Length | 94 |
| Sequences | 11 |
| Columns | 101 |
| Reading direction | reverse |
| Mean pairwise identity | 84.97 |
| Shannon entropy | 0.31212 |
| G+C content | 0.40173 |
| Mean single sequence MFE | -28.56 |
| Consensus MFE | -13.74 |
| Energy contribution | -13.38 |
| Covariance contribution | -0.36 |
| Combinations/Pair | 1.30 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.48 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.622956 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 20256009 94 - 21146708 GUGGCA---CGGCCAUAACGCUGU-UGUCGUUU--GGGC-CGGGUCAAAUGUCAAGCACAUUAUUUUUGUACUUACGUACAAUAAAUAUAAAAUGCAUGAU .((((.---(((((..(((((...-.).)))).--.)))-)).))))...((((.(((...(((((((((((....)))))).))))).....))).)))) ( -29.90, z-score = -2.88, R) >droSim1.chr2R 18706815 94 - 19596830 GUGGCC---CGGCCAUAACGCCGU-UGUCGUUU--GGGC-CGGGUCAAAUGUCAAGCACAUUAUUUUUGUACUUACGUACAAUAAAUAUAAAAUGCAUGAU .(((((---(((((..(((((...-.).)))).--.)))-)))))))...((((.(((...(((((((((((....)))))).))))).....))).)))) ( -35.70, z-score = -4.20, R) >droSec1.super_23 126479 94 - 989336 GUGGCC---CGGCCAUAACGCCGU-UGUCGUUU--GGGC-CGGGUCAAAUGUCAAGCACAUUAUUUUUGUACUUACGUACAAUAAAUAUAAAAUGCAUGAU .(((((---(((((..(((((...-.).)))).--.)))-)))))))...((((.(((...(((((((((((....)))))).))))).....))).)))) ( -35.70, z-score = -4.20, R) >droEre2.scaffold_4845 21614085 94 - 22589142 GUGGCC---CGACCAUAACGCUGU-UGUCGUUU--GGGC-CGGGUCAAAUGUCAAGCACAUUAUUUUUGUACUUACGUACAAUAAAUAUAAAAUGCAUGAU .(((((---((.(((.(((((...-.).)))))--))..-)))))))...((((.(((...(((((((((((....)))))).))))).....))).)))) ( -30.80, z-score = -3.27, R) >droYak2.chr2R 20216477 97 - 21139217 GUGGCACGGCGGCCAUAGCG-CUGUUGUCGUUU--GGGC-CGGGUCAAAUGUCAAGCACAUUAUUUUUGUAUUUACGUACAAUAAAUAUAAAGUGCAUGAU .((((.((((..(((.((((-(....).)))))--))))-)).))))...((((.((((.((((..((((((....)))))).....)))).)))).)))) ( -29.60, z-score = -1.69, R) >droAna3.scaffold_13266 5719158 98 + 19884421 GUGGCCCGGCGGCCAUAACGCUGUCUGUCGUUU--GGGC-CGGGUCAAAUGUCAAGCACAUUAUUUUUGUAUUUACGUACAAUAAAUAUAAAAUGCAUGAU .(((((((((..(((.(((((.....).)))))--))))-)))))))...((((.(((...(((((((((((....)))))).))))).....))).)))) ( -33.20, z-score = -2.94, R) >dp4.chr3 1359988 93 + 19779522 GUGGCC---GGGCCAUAACGUUG--UGUCGUUU--GGGC-CGGGUCAAAUGUCAGGCACAUUAUUUUUGUAUUUACGUAUAAUAAAUAUAAAAUGCAUGAU .(((((---.((((..((((...--...)))).--.)))-).)))))...((((.(((......(((((((((((.......)))))))))))))).)))) ( -30.30, z-score = -2.86, R) >droPer1.super_34 550143 93 + 916997 GUGGCC---GGGCCAUAACGUUG--UGUCGUUU--GGGC-CGGGUCAAAUGUCAGGCACAUUAUUUUUGUAUUUACGUAUAAUAAAUAUAAAAUGCAUGAU .(((((---.((((..((((...--...)))).--.)))-).)))))...((((.(((......(((((((((((.......)))))))))))))).)))) ( -30.30, z-score = -2.86, R) >droVir3.scaffold_12875 7366520 84 - 20611582 --------GUGGCCA-AACGU----UGUCGUUU--GGGC-CGGGUCAAAUGUCAAGCACAUUAUUUUUGUGUUUACGUAUAAUAAAUAU-AAAUGCAUGAU --------.((((((-((((.----...)))))--.)))-)).((((...((.(((((((.......)))))))))((((.........-..)))).)))) ( -21.80, z-score = -1.60, R) >droMoj3.scaffold_6496 26162156 85 - 26866924 --------GUGGUCGCAACGU----UGUCGUUU--GGGC-CGGGUCAAAUGUCAAGCACAUUAUUUUUGUGUUUAUGUAUAAUAAAUAU-GAAUAGCUUAU --------..((((.(((((.----...)).))--))))-)((((..(((((.....)))))...((..(((((((.....))))))).-.))..)))).. ( -16.50, z-score = -0.03, R) >droGri2.scaffold_15245 917144 95 - 18325388 -----GUGGCCAACAAAGCGUUUGUCGUUGUUUUGGGGCGCGGGUCAAAUGUCAAGCACAUUAUUUUUAUGUUUACGUAUAAUAAAUAUAAA-UGCAUGAU -----(((.((..(((((((........))))))))).))).........((((.(((.......((((((((((.......))))))))))-))).)))) ( -20.41, z-score = -0.14, R) >consensus GUGGCC___CGGCCAUAACGCUGU_UGUCGUUU__GGGC_CGGGUCAAAUGUCAAGCACAUUAUUUUUGUAUUUACGUACAAUAAAUAUAAAAUGCAUGAU .........(((((..((((........))))....))).))........((((.(((...(((((((((((....)))))).))))).....))).)))) (-13.74 = -13.38 + -0.36)

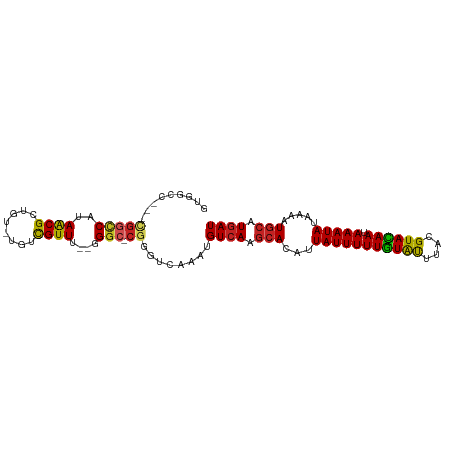
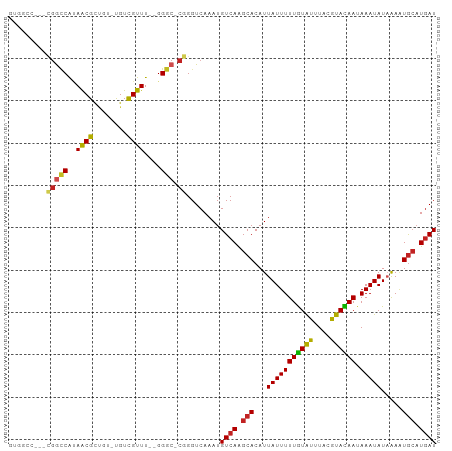
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:48:10 2011