
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 18,961,353 – 18,961,464 |
| Length | 111 |
| Max. P | 0.792903 |

| Location | 18,961,353 – 18,961,464 |
|---|---|
| Length | 111 |
| Sequences | 13 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 71.10 |
| Shannon entropy | 0.65685 |
| G+C content | 0.57587 |
| Mean single sequence MFE | -46.76 |
| Consensus MFE | -16.26 |
| Energy contribution | -15.62 |
| Covariance contribution | -0.64 |
| Combinations/Pair | 2.15 |
| Mean z-score | -1.66 |
| Structure conservation index | 0.35 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.792903 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 18961353 111 - 21146708 CAUGUGGCCAUGAGCAUCCUUGGCGGUCACCU-GUGGCUG-UGUGCACUUCAGGUGCACG-UGAAGCAGUCGCGGA--GC-UCCAAGGGCUGCUUAGAAUAAU-GCACAACUGAGGCG ......(((.(((((((((((((.(((...((-(((((((-(((((((.....)))))).-....)))))))))).--))-)))))))).)))))).......-.((....)).))). ( -45.70, z-score = -1.36, R) >droSim1.chr2R 17545063 111 - 19596830 CAUGUGGCCAUGAGCAUCCUUGGCGGCCACCU-GUGGCUG-UGUGCACUCCAGGUGCACG-UGAAGCAGUCGCGGG--GC-UCCAAGGGCUGCUUAGAAUAAU-GCACAACUGAGGCG ......(((.(((((((((((((.((((.((.-(((((((-(((((((.....)))))).-....)))))))))))--))-)))))))).)))))).......-.((....)).))). ( -51.20, z-score = -2.09, R) >droSec1.super_9 2252808 111 - 3197100 CAUGUGGCCAUGAGCAUCCUUGGCGGCCACCU-GUGGCUG-UGUGCACUCCAGGUGCACG-UGAAGCAGUCGCGGG--GC-UCCAAGGGCUGCUUAGAAUAAU-GCACAACUGAGGCG ......(((.(((((((((((((.((((.((.-(((((((-(((((((.....)))))).-....)))))))))))--))-)))))))).)))))).......-.((....)).))). ( -51.20, z-score = -2.09, R) >droYak2.chr2R 18923784 111 - 21139217 CAUGUGGCCAUGAGCAGCCUUGGCAGCCACCU-GUGGCUG-UGUGCACUCCAGGUGCACG-UGAAGCAGUCGCGGG--GC-UCCAAGGUCUGCUUAGAAUAAU-GCACAACUGAGGCG ......(((.(((((((((((((.((((.((.-(((((((-(((((((.....)))))).-....)))))))))))--))-))))))).))))))).......-.((....)).))). ( -54.70, z-score = -3.14, R) >droEre2.scaffold_4845 20324815 111 - 22589142 CAUGUGGCCAUGAGCAUCCUUGGCGGCCACCU-GUGGCUG-UGUGCACUCCAGGUGCACG-UAAUGCAGUCGCGGG--GC-UCCAAGGUCUGCUUAGAAUAAU-GCACAACUGAGGCG ......(((.((((((.((((((.((((.((.-(((((((-(((((((.....)))))).-....)))))))))))--))-)))))))..)))))).......-.((....)).))). ( -49.60, z-score = -2.22, R) >droAna3.scaffold_13266 15290602 109 - 19884421 CAUGUGGCCAUGAGCACUCUUGGCGGCCACCU-GUGGCUG-UGUGCACUUCAGGUGCACG-UGAAGCAGCUGCGGG-UGC-UCCAGGGCGUGCUUAGACUAAU-GCACAACUGAG--- ..((((....(((((((((((((.(((.((((-(..((((-(((((((.....)))))).-....)))))..))))-)))-))))))).))))))).......-.))))......--- ( -51.40, z-score = -2.21, R) >dp4.chr3 3352711 111 - 19779522 UAUGUGGUCAUGAGCGGCCCUUGCGUUCACCG-GUAGCUG-UGUGCACUUCAGGUACACG-UGAAGCAGCUUCAGG-UGC-GCUUGGGUC-GCUUAGAACAAU-GCACAACUGAGGCA ..((((((..((((((((((..(((..((((.-..(((((-(...(((...........)-))..))))))...))-)))-))..)))))-)))))..))...-.))))......... ( -45.90, z-score = -1.89, R) >droPer1.super_2 3545838 111 - 9036312 UAUGUGGUCAUGAGCGGCCCUUGCGUUCACCG-GUAGCUG-UGUGCACUUCAGGUACACG-GGAAGCAGCUUCAGG-UGC-GCUUGGGUC-GCUUAGAACAAU-GCACAACUGAGGCA ..((((((..((((((((((..(((..((((.-....(((-(((((.(....))))))))-)((((...)))).))-)))-))..)))))-)))))..))...-.))))......... ( -45.60, z-score = -1.85, R) >droWil1.scaffold_180699 2078585 116 + 2593675 UAUGUGGCCAUGAGCAAACAUGUGUUUCAUCUGGCAGCUGCUGUGGACUUUGGGUGCACAUUGAAGCAGUUGCUGAUGGC-AUUUUGUUUUGCGUAAAUCUAA-ACACAACUGAGAGA ..((((.......(((((((.((((..((((.(((((((((((((.((.....)).))).....))))))))))))))))-))..)).)))))..........-.))))......... ( -34.67, z-score = -0.41, R) >droVir3.scaffold_12875 11570948 113 + 20611582 AAUGUGGCCAUGAGCGUGCGUGACGUCUGCC---CGCCUG-UGCGCACUCCAGGUGCGCG-UUAAGCAGCUGCGGGUGGCGUCCACGAGACGCUUAGAACUAUAGCACUACUGAGACA ..(((((...(((((((.(((((((((.(((---((((((-(((((((.....)))))).-....))))..))))))))))).))))..)))))))...))))).............. ( -50.80, z-score = -2.21, R) >droMoj3.scaffold_6496 18132791 113 - 26866924 UAUGUGGCCAUGAGCACGCGCGGCGUCUGAC---CGCCUG-UGUGCACUCCAGGUGCACG-UGAAGCAGCUGCGGGCAGCGUCCACGUGUUGCUUAGAACUAUAGCACUACUGAGACA ..(((((...((((((((((.((((.(((.(---((((((-(((((((.....)))))).-....))))..)))).))))).)).)))).))))))...))))).............. ( -44.90, z-score = -0.28, R) >droGri2.scaffold_15245 15950554 113 + 18325388 AAUGUGGCCAUGAGCAUCCUUGGUGUCUGCU---CCGCUC-UGUGCACUCCAGGUGCAUG-UUAAACGGCUGUGGGUGGCGUCCAUGUGAUGCUUAGAACUAUAGCACUACUGAGGCA ..(((((...(((((((((.(((((((.((.---((((..-.((((((.....))))))(-(......)).)))))))))).))).).))))))))...)))))((.((....)))). ( -37.80, z-score = 0.09, R) >anoGam1.chr3L 19117470 95 - 41284009 CAUGUAGCCAACGGGGGGCGCACCGCUG-----GUGGGAAGUG-GAGCCCGAGGCAUACCUUCCCGCUGGUGCGCU-CGC-UGUAGAACCUGCUUACUGACGC--------------- ...((((.(.((((.(((((((((...(-----((((((((.(-..(((...)))...))))))))))))))))))-).)-))).)...))))..........--------------- ( -44.40, z-score = -1.91, R) >consensus CAUGUGGCCAUGAGCAUCCUUGGCGGCCACCU_GUGGCUG_UGUGCACUCCAGGUGCACG_UGAAGCAGCUGCGGG__GC_UCCAAGGGCUGCUUAGAACAAU_GCACAACUGAGGCA ..........((((((((((.((......((...((((((.((((.((.....)).))))......))))))..))......)).)))).))))))...................... (-16.26 = -15.62 + -0.64)

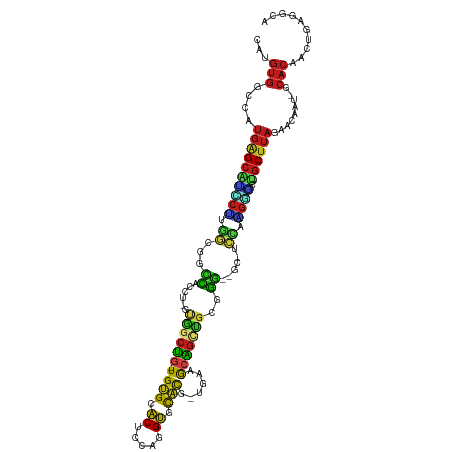
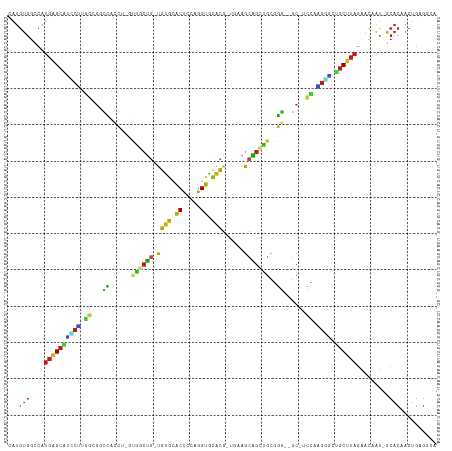
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:44:49 2011