
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 18,758,659 – 18,758,764 |
| Length | 105 |
| Max. P | 0.970370 |

| Location | 18,758,659 – 18,758,764 |
|---|---|
| Length | 105 |
| Sequences | 12 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 74.99 |
| Shannon entropy | 0.50491 |
| G+C content | 0.45411 |
| Mean single sequence MFE | -28.74 |
| Consensus MFE | -9.92 |
| Energy contribution | -9.92 |
| Covariance contribution | 0.01 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.35 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.35 |
| SVM RNA-class probability | 0.654107 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
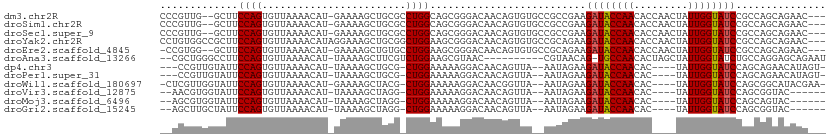
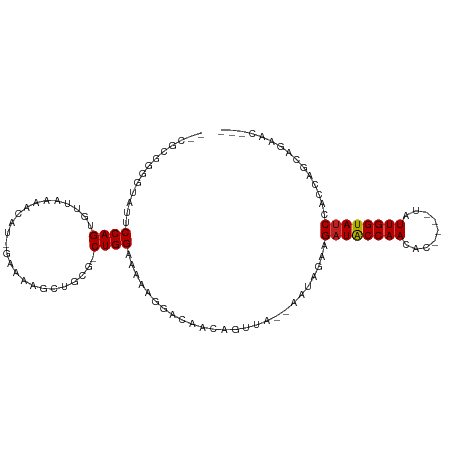
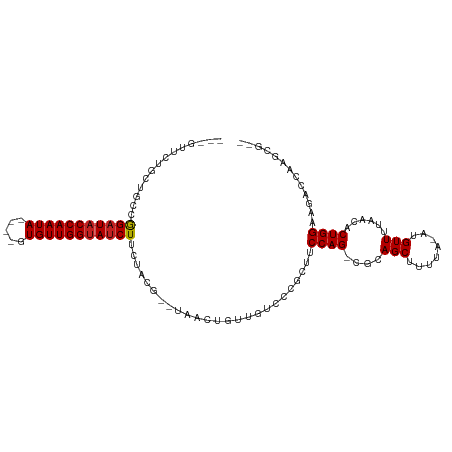
>dm3.chr2R 18758659 105 + 21146708 CCCGUUG--GCUUCCAGUGUUAAAACAU-GAAAAGCUGCGCCUGGCAGCGGGACAACAGUGUGCCGCCGAAGAUACCAACACCAACUAUUGGUAUCCGCCAGCAGAAC--- ..(((.(--((((...((((....))))-...)))))))).(((((.((((.(((....))).))))....((((((((.........)))))))).)))))......--- ( -35.20, z-score = -2.16, R) >droSim1.chr2R 17353846 105 + 19596830 CCCGUUG--GCUUCCAGUGUUAAAACAU-GAAAAGCUGCGCCUGGCAGCGGGACAACAGUGUGCCGCCGAAGAUACCAACACCAACUAUUGGUAUCCGCCAGCAGAAC--- ..(((.(--((((...((((....))))-...)))))))).(((((.((((.(((....))).))))....((((((((.........)))))))).)))))......--- ( -35.20, z-score = -2.16, R) >droSec1.super_9 2057391 105 + 3197100 CCCGUUG--GCUUCCAGUGUUAAAACAU-GAAAAGCUGCGCCUGGCAGCGGGACAACAGUGUGCCGCCGAAGAUACCAACACCAACUAUUGGUAUCCGCCAGCAGAAC--- ..(((.(--((((...((((....))))-...)))))))).(((((.((((.(((....))).))))....((((((((.........)))))))).)))))......--- ( -35.20, z-score = -2.16, R) >droYak2.chr2R 18723515 108 + 21139217 CCUGUGGCCGCUUCCAGUGUUAAAACAUAGGAAAGCUGCGGCUGGAAGCGGGACAACAGUGUGCCGCAGAAGAUACCAACACCAACUAUUGGUAUCCGCCAGCAGAAC--- .((((((((((((((.((((....)))).))))....))))))((..((((.(((....))).))))....((((((((.........))))))))..)).))))...--- ( -41.50, z-score = -3.35, R) >droEre2.scaffold_4845 20123110 104 + 22589142 -CCGUGG--GCUUCCAGUGUUAAAACAU-GAAAAGCUGUGCCUGGAAGCGGGACAACAGUGUGCCGCAGAAGAUACCAACACCAACUAUUGGUAUCCGCCAGCAGAAC--- -..((.(--((((((((.((....(((.-.......)))))))))))((((.(((....))).))))....((((((((.........)))))))).))).)).....--- ( -33.20, z-score = -1.78, R) >droAna3.scaffold_13266 15095816 97 + 19884421 --CGCUGGGCCUUCCAGUGUUAAAACAU-UAAAAGCUUCGUCUGGAAGCGUAAC----------CGUAACAG-UGCCAACACUAGCUAUUGGUAUCUGCCAGGAGCAGAAU --.(((((.....)))))((((.....(-((...(((((.....))))).))).----------..))))((-((....)))).(((.(((((....))))).)))..... ( -25.90, z-score = -0.66, R) >dp4.chr3 17457321 99 + 19779522 ---CCGUUGUAUUCCAGUGUUAAAACAU-UAAAAGCUGCG-CUGGAAAAAGGACAACAGUUA--AAUAGAAGAUACCAACAC----UAUUGGUAUCCAGCAGAACAUAGU- ---..(((((.(((((((((........-........)))-)))))).....))))).(((.--.......((((((((...----..))))))))......))).....- ( -25.43, z-score = -3.39, R) >droPer1.super_31 600834 99 - 935084 ---CCGUUGUAUUCCAGUGUUAAAACAU-UAAAAGCUGCG-CUGGAAAAAGGACAACAGUUA--AAUAGAAGAUACCAACAC----UAUUGGUAUCCAGCAGAACAUAGU- ---..(((((.(((((((((........-........)))-)))))).....))))).(((.--.......((((((((...----..))))))))......))).....- ( -25.43, z-score = -3.39, R) >droWil1.scaffold_180697 3557090 101 - 4168966 -CUCGUUGGUAUUCCAGUGUUAAAACAU-GAAAAGCUACG-CUGGAAAAAGGACAACGGUUA--AAUAGAAGAUACCAACAC----UAUUGGUAUCCAGCGGCAUACGAA- -.((((((...(((((((((........-........)))-))))))...............--.......((((((((...----..))))))))))))))........- ( -26.29, z-score = -2.53, R) >droVir3.scaffold_12875 19169056 95 + 20611582 --AACGUGGUAUUCCAGUGUUAAAACAU-UAAAAGCUAGG-CUGGAAAAAGGACAACAGUUA--AAUAGAAGAUACCAACAC----UAUUGGUAUCCAGCGGUAC------ --..(((....(((((((.(........-........).)-))))))...............--.......((((((((...----..))))))))..)))....------ ( -19.49, z-score = -1.13, R) >droMoj3.scaffold_6496 12857609 95 + 26866924 --AGCGUGGUAUUCCAGUGUUAAAACAU-UAAAAGCUAGG-CUGGAAAAAGGACAACAGUUA--AAUAGAAGAUACCAACAC----UAUUGGUAUCCAGCAGUAC------ --(((..((....))(((((....))))-)....)))..(-(((((.....(((....))).--.........((((((...----..)))))))))))).....------ ( -21.10, z-score = -1.73, R) >droGri2.scaffold_15245 13211165 95 + 18325388 --AGCUUGCUAUUCCAGUGUUAAAACAU-UAAAAGCUAGG-CUGGAAAAAGGACAACAGUUA--AAUAGAAGAUACCAACAC----UAUUGGUAUCCAGCGGUAC------ --.(((.(((.(((((((.(........-........).)-))))))...............--.......((((((((...----..)))))))).))))))..------ ( -20.99, z-score = -1.41, R) >consensus __CGCGGGGUAUUCCAGUGUUAAAACAU_GAAAAGCUGCG_CUGGAAAAAGGACAACAGUUA__AAUAGAAGAUACCAACAC____UAUUGGUAUCCACCAGCAGAAC___ .............((((.(((............))).....))))..........................((((((((.........))))))))............... ( -9.92 = -9.92 + 0.01)
| Location | 18,758,659 – 18,758,764 |
|---|---|
| Length | 105 |
| Sequences | 12 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 74.99 |
| Shannon entropy | 0.50491 |
| G+C content | 0.45411 |
| Mean single sequence MFE | -29.89 |
| Consensus MFE | -16.15 |
| Energy contribution | -16.24 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.54 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.83 |
| SVM RNA-class probability | 0.970370 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
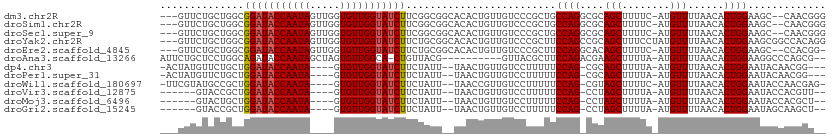
>dm3.chr2R 18758659 105 - 21146708 ---GUUCUGCUGGCGGAUACCAAUAGUUGGUGUUGGUAUCUUCGGCGGCACACUGUUGUCCCGCUGCCAGGCGCAGCUUUUC-AUGUUUUAACACUGGAAGC--CAACGGG ---((.(((((((.(((((((((((.....)))))))))))))))))).)).((((((....(((((.....)))))(((((-((((....))).)))))).--)))))). ( -41.20, z-score = -2.41, R) >droSim1.chr2R 17353846 105 - 19596830 ---GUUCUGCUGGCGGAUACCAAUAGUUGGUGUUGGUAUCUUCGGCGGCACACUGUUGUCCCGCUGCCAGGCGCAGCUUUUC-AUGUUUUAACACUGGAAGC--CAACGGG ---((.(((((((.(((((((((((.....)))))))))))))))))).)).((((((....(((((.....)))))(((((-((((....))).)))))).--)))))). ( -41.20, z-score = -2.41, R) >droSec1.super_9 2057391 105 - 3197100 ---GUUCUGCUGGCGGAUACCAAUAGUUGGUGUUGGUAUCUUCGGCGGCACACUGUUGUCCCGCUGCCAGGCGCAGCUUUUC-AUGUUUUAACACUGGAAGC--CAACGGG ---((.(((((((.(((((((((((.....)))))))))))))))))).)).((((((....(((((.....)))))(((((-((((....))).)))))).--)))))). ( -41.20, z-score = -2.41, R) >droYak2.chr2R 18723515 108 - 21139217 ---GUUCUGCUGGCGGAUACCAAUAGUUGGUGUUGGUAUCUUCUGCGGCACACUGUUGUCCCGCUUCCAGCCGCAGCUUUCCUAUGUUUUAACACUGGAAGCGGCCACAGG ---((.((((.((.(((((((((((.....))))))))))).)))))).)).((((.(..((((((((((....(((........)))......)))))))))).))))). ( -42.60, z-score = -3.09, R) >droEre2.scaffold_4845 20123110 104 - 22589142 ---GUUCUGCUGGCGGAUACCAAUAGUUGGUGUUGGUAUCUUCUGCGGCACACUGUUGUCCCGCUUCCAGGCACAGCUUUUC-AUGUUUUAACACUGGAAGC--CCACGG- ---((.((((.((.(((((((((((.....))))))))))).)))))).)).((((.(....((((((((....(((.....-..)))......))))))))--).))))- ( -34.60, z-score = -1.64, R) >droAna3.scaffold_13266 15095816 97 - 19884421 AUUCUGCUCCUGGCAGAUACCAAUAGCUAGUGUUGGCA-CUGUUACG----------GUUACGCUUCCAGACGAAGCUUUUA-AUGUUUUAACACUGGAAGGCCCAGCG-- .....(((...(((....(((..((((.((((....))-)))))).)----------))....(((((((..(((((.....-..)))))....)))))))))).))).-- ( -27.60, z-score = -1.22, R) >dp4.chr3 17457321 99 - 19779522 -ACUAUGUUCUGCUGGAUACCAAUA----GUGUUGGUAUCUUCUAUU--UAACUGUUGUCCUUUUUCCAG-CGCAGCUUUUA-AUGUUUUAACACUGGAAUACAACGG--- -.............((((((((((.----..))))))))))......--...(((((((.....((((((-.(.(((.....-..)))....).)))))).)))))))--- ( -23.50, z-score = -2.31, R) >droPer1.super_31 600834 99 + 935084 -ACUAUGUUCUGCUGGAUACCAAUA----GUGUUGGUAUCUUCUAUU--UAACUGUUGUCCUUUUUCCAG-CGCAGCUUUUA-AUGUUUUAACACUGGAAUACAACGG--- -.............((((((((((.----..))))))))))......--...(((((((.....((((((-.(.(((.....-..)))....).)))))).)))))))--- ( -23.50, z-score = -2.31, R) >droWil1.scaffold_180697 3557090 101 + 4168966 -UUCGUAUGCCGCUGGAUACCAAUA----GUGUUGGUAUCUUCUAUU--UAACCGUUGUCCUUUUUCCAG-CGUAGCUUUUC-AUGUUUUAACACUGGAAUACCAACGAG- -.............((((((((((.----..))))))))))......--....(((((......((((((-.(((((.....-..)))...)).))))))...)))))..- ( -23.90, z-score = -2.23, R) >droVir3.scaffold_12875 19169056 95 - 20611582 ------GUACCGCUGGAUACCAAUA----GUGUUGGUAUCUUCUAUU--UAACUGUUGUCCUUUUUCCAG-CCUAGCUUUUA-AUGUUUUAACACUGGAAUACCACGUU-- ------.....((((((...(((((----((..(((......)))..--..))))))).......)))))-)((((...(((-(....))))..))))...........-- ( -18.40, z-score = -1.07, R) >droMoj3.scaffold_6496 12857609 95 - 26866924 ------GUACUGCUGGAUACCAAUA----GUGUUGGUAUCUUCUAUU--UAACUGUUGUCCUUUUUCCAG-CCUAGCUUUUA-AUGUUUUAACACUGGAAUACCACGCU-- ------.....(((((....(((((----((..(((......)))..--..)))))))......((((((-...(((.....-..)))......))))))..))).)).-- ( -18.90, z-score = -1.14, R) >droGri2.scaffold_15245 13211165 95 - 18325388 ------GUACCGCUGGAUACCAAUA----GUGUUGGUAUCUUCUAUU--UAACUGUUGUCCUUUUUCCAG-CCUAGCUUUUA-AUGUUUUAACACUGGAAUAGCAAGCU-- ------.....((((((...(((((----((..(((......)))..--..))))))).......)))))-)..(((((...-.(((((((....)))))))..)))))-- ( -22.10, z-score = -1.75, R) >consensus ___GUUCUGCUGCCGGAUACCAAUA____GUGUUGGUAUCUUCUACG__UAACUGUUGUCCCGCUUCCAG_CGCAGCUUUUA_AUGUUUUAACACUGGAAGACCAAGCG__ ..............(((((((((((.....))))))))))).........................((((....(((........)))......))))............. (-16.15 = -16.24 + 0.09)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:44:23 2011