
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 18,738,268 – 18,738,384 |
| Length | 116 |
| Max. P | 0.633800 |

| Location | 18,738,268 – 18,738,382 |
|---|---|
| Length | 114 |
| Sequences | 13 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 77.41 |
| Shannon entropy | 0.52003 |
| G+C content | 0.51009 |
| Mean single sequence MFE | -42.82 |
| Consensus MFE | -14.11 |
| Energy contribution | -14.28 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.39 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.33 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.633800 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
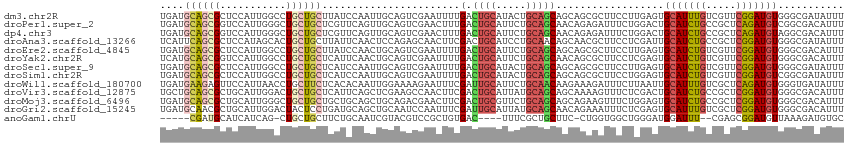
>dm3.chr2R 18738268 114 - 21146708 UGAUGCAGCGCUCCAUUGGCCUGCUGCUUAUCCAAUUGCAGUCGAAUUUUGACUGCAUACUGCAGCAGCAGCGCUUCCUUGAGUGCAUUUGUCGUUCGGAUGUGGGCGAUAUUU .......((((((....(((((((((((........(((((((((...)))))))))......)))))))).))).....))))))...(((((((((....)))))))))... ( -47.44, z-score = -3.30, R) >droPer1.super_2 4406903 114 - 9036312 UGAUGCAGCGGUCCAUUGGGCUGCUGCUCGUUCAGUUGCAGUCGAACUUUGACUGCAUUCUGCAGCAACAGAGAUUUCUGGACUGCAUCUGCCGCUCAGAUGUCGGCGACAUUU .((.((((((((((...))))))))))))(((.((.(((((((.....(((.((((.....)))))))(((......)))))))))).))(((((......).))))))).... ( -48.60, z-score = -3.04, R) >dp4.chr3 4219955 114 - 19779522 UGAUGCAGCGGUCCAUUGGGCUGCUGCUCGUUCAGUUGCAGUCGAACUUUGACUGCAUUCUGCAGCAACAGAGAUUUCUGGACUGCAUCUGCCGCUCAGAUGUAGGCGACAUUU .((.((((((((((...))))))))))))((((((.(((((((((...)))))))))..))).)))..(((......)))..(((((((((.....)))))))))......... ( -50.00, z-score = -3.56, R) >droAna3.scaffold_13266 15077365 114 - 19884421 UCAUUCAGCGCUCCAUAGCACUGCUGCUUAUUCAACUCCAGAGCAACUUCGACUGCAUCCUGCAACAGCAACGCUUCCUCGAUUGCAUCUGCCGCUCGGAUGUGGGCGAUAUUU ........(((.((((((((....))).........(((.((((.........(((.....)))...((((((......)).)))).......)))))))))))))))...... ( -28.00, z-score = 0.25, R) >droEre2.scaffold_4845 20102271 114 - 22589142 UGAUGCAGCGCUCCAUUGGCCUGCUGCUUAUCCAACUGCAGUCGAAUUUUGACUGCAUUCUGCAGCAGCAGCGCUUCCUUGAGUGCAUCUGUCGUUCGGAUGUGGGCGACAUUU .......((((((....(((((((((((........(((((((((...)))))))))......)))))))).))).....))))))...(((((((((....)))))))))... ( -48.54, z-score = -3.18, R) >droYak2.chr2R 18702741 114 - 21139217 UCAUGCAGCGGUCCAUUGGCCUGCUGCUCAUUCAACUGCAGUCGAAUUUUGACUGCAUUCUGCAGCAACAGCGCUUCCUCGAGUGCAUCUGUCGUUCGGAUGUGGGCGACAUUU ....(((((((((....)))).))))).........(((((((((...))))))))).............((((((....))))))...(((((((((....)))))))))... ( -41.30, z-score = -1.58, R) >droSec1.super_9 2037162 114 - 3197100 UGAUGCAGCGCUCCAUUGGCCUGCUGCUCAUCCAAUUGCAGUCGAAUUUUGACUGCAUACUGCAGCAGCAGCGCUUCCUUGAGUGCAUCUGUCGUUCGGAUGUGGGCGAUAUUU .......((((((....(((((((((((((......(((((((((...)))))))))...)).)))))))).))).....))))))...(((((((((....)))))))))... ( -48.00, z-score = -3.01, R) >droSim1.chr2R 17332433 114 - 19596830 UGAUGCAGCGCUCCAUUGGCCUGCUGCUCAUCCAAUUGCAGUCGAAUUUUGACUGCAUACUGCAGCAGCAGCGCUUCCUGGAGUGCAUCUGUCGUUCGGAUGUCGGCGAUAUUU ...(((.((((((((..(((((((((((((......(((((((((...)))))))))...)).)))))))).)))...))))))))(((((.....)))))....)))...... ( -51.20, z-score = -3.90, R) >droWil1.scaffold_180700 2890756 114 + 6630534 UGAUGAAGAGUUCCAUUAACCUGCUUCUCACACAAUUGGAAAAGAAUUUCGAUUGCAUUCUGCAACAAGAAAGAUUUCUUAAUUGCAUUUGUCGCUCAGAUGUGGGUGAUAUUU (((.((((.(((.....)))...)))))))..(((((((((.....))))))))).....(((((.(((((....)))))..)))))..(((((((((....)))))))))... ( -25.40, z-score = -0.48, R) >droVir3.scaffold_12875 13481031 114 - 20611582 UGCUGCAGCGCUGCAUUGGACUGCUGCUCAUUCAGCUCGAAGCCAACUUCGACUGCAUUAUGCAGCAGCAAAAGUUUCUCGACUGCAUCUGCCGCUCGGAUGUGGGCGACAUUU ((((((.((((.(((......))).)).....(((.((((((....)))))))))......)).))))))........(((.(..((((((.....))))))..).)))..... ( -39.40, z-score = -0.46, R) >droMoj3.scaffold_6496 6918712 114 - 26866924 UGAUGCAGCGCUGCAUUGGGCUGCUGCUGCUGCAGCUGCAGACGAACUUCGACUGCGUUCUGCAGCAGCAGAAGUUUCUGGAGUGCAUCUGCCGCUCGGAUGUGGGCGACAUUU ((.(((.(((((.((..(((((.((((((((((((.(((((.((.....)).)))))..)))))))))))).))))).)).))))).....((((......))))))).))... ( -54.60, z-score = -2.37, R) >droGri2.scaffold_15245 572832 114 + 18325388 UGAUGCAACGCUGCAUUGGACUACUCCUGAUGCAGCUGCAAUCCAAUUUCGAUUGCAUUAUGCAGCAACAGAAAUUUCUCGAGUGCAUUUGUCGCUCGGAUGUGGGCGACAUUU ...((((..(((((((((((....))).))))))))(((((((.......)))))))...))))(((...((......))...)))...(((((((((....)))))))))... ( -43.20, z-score = -3.51, R) >anoGam1.chrU 54660854 101 - 59568033 -----CGAUGCAUCAUCAG-CUGCUGCUUCUGCAAUCGUACGUCCGCUGUGAC----UUUCGCUGCUUC-CUGGUGGCUGGGAUGGAUUU--CGAGCGGAUGUUAAAGAUGUGC -----....(((.((((((-(....)))(((((..(((...((((((.(((..----...))).)).((-(..(...)..))).))))..--)))))))).......))))))) ( -31.00, z-score = -0.00, R) >consensus UGAUGCAGCGCUCCAUUGGCCUGCUGCUCAUUCAAUUGCAGUCGAAUUUUGACUGCAUUCUGCAGCAGCAGAGCUUCCUGGAGUGCAUCUGUCGCUCGGAUGUGGGCGACAUUU ....((((((...........)))))).......................(.((((.....)))))..................(((((((.....)))))))........... (-14.11 = -14.28 + 0.17)


| Location | 18,738,270 – 18,738,384 |
|---|---|
| Length | 114 |
| Sequences | 13 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 76.59 |
| Shannon entropy | 0.54116 |
| G+C content | 0.52080 |
| Mean single sequence MFE | -43.49 |
| Consensus MFE | -14.11 |
| Energy contribution | -14.28 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.39 |
| Mean z-score | -2.06 |
| Structure conservation index | 0.32 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.566149 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
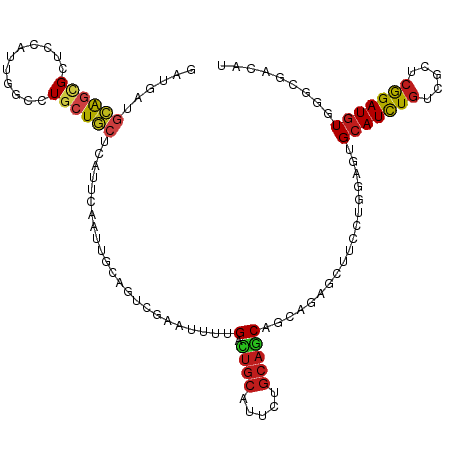
>dm3.chr2R 18738270 114 - 21146708 GUUGAUGCAGCGCUCCAUUGGCCUGCUGCUUAUCCAAUUGCAGUCGAAUUUUGACUGCAUACUGCAGCAGCAGCGCUUCCUUGAGUGCAUUUGUCGUUCGGAUGUGGGCGAUAU .........((((((....(((((((((((........(((((((((...)))))))))......)))))))).))).....))))))...(((((((((....))))))))). ( -47.44, z-score = -3.09, R) >droPer1.super_2 4406905 114 - 9036312 GAUGAUGCAGCGGUCCAUUGGGCUGCUGCUCGUUCAGUUGCAGUCGAACUUUGACUGCAUUCUGCAGCAACAGAGAUUUCUGGACUGCAUCUGCCGCUCAGAUGUCGGCGACAU (((((.((((((((((...))))))))))))))).((.(((((((.....(((.((((.....)))))))(((......)))))))))).))(((((......).))))..... ( -49.80, z-score = -3.14, R) >dp4.chr3 4219957 114 - 19779522 GAUGAUGCAGCGGUCCAUUGGGCUGCUGCUCGUUCAGUUGCAGUCGAACUUUGACUGCAUUCUGCAGCAACAGAGAUUUCUGGACUGCAUCUGCCGCUCAGAUGUAGGCGACAU (((((.((((((((((...)))))))))))))))(((.(((((((((...)))))))))..)))..((..(((......)))..(((((((((.....)))))))))))..... ( -51.20, z-score = -3.67, R) >droAna3.scaffold_13266 15077367 114 - 19884421 CGUCAUUCAGCGCUCCAUAGCACUGCUGCUUAUUCAACUCCAGAGCAACUUCGACUGCAUCCUGCAACAGCAACGCUUCCUCGAUUGCAUCUGCCGCUCGGAUGUGGGCGAUAU ..........(((.((((((((....))).........(((.((((.........(((.....)))...((((((......)).)))).......))))))))))))))).... ( -28.00, z-score = 0.69, R) >droEre2.scaffold_4845 20102273 114 - 22589142 GGUGAUGCAGCGCUCCAUUGGCCUGCUGCUUAUCCAACUGCAGUCGAAUUUUGACUGCAUUCUGCAGCAGCAGCGCUUCCUUGAGUGCAUCUGUCGUUCGGAUGUGGGCGACAU .........((((((....(((((((((((........(((((((((...)))))))))......)))))))).))).....))))))...(((((((((....))))))))). ( -48.54, z-score = -2.64, R) >droYak2.chr2R 18702743 114 - 21139217 GGUCAUGCAGCGGUCCAUUGGCCUGCUGCUCAUUCAACUGCAGUCGAAUUUUGACUGCAUUCUGCAGCAACAGCGCUUCCUCGAGUGCAUCUGUCGUUCGGAUGUGGGCGACAU .(((....(((((((....)))).)))((((((.....(((((((((...))))))))).((((.(((.(((((((((....))))))...))).))))))).))))))))).. ( -41.80, z-score = -1.23, R) >droSec1.super_9 2037164 114 - 3197100 GAUGAUGCAGCGCUCCAUUGGCCUGCUGCUCAUCCAAUUGCAGUCGAAUUUUGACUGCAUACUGCAGCAGCAGCGCUUCCUUGAGUGCAUCUGUCGUUCGGAUGUGGGCGAUAU .........((((((....(((((((((((((......(((((((((...)))))))))...)).)))))))).))).....))))))...(((((((((....))))))))). ( -48.00, z-score = -2.74, R) >droSim1.chr2R 17332435 114 - 19596830 GAUGAUGCAGCGCUCCAUUGGCCUGCUGCUCAUCCAAUUGCAGUCGAAUUUUGACUGCAUACUGCAGCAGCAGCGCUUCCUGGAGUGCAUCUGUCGUUCGGAUGUCGGCGAUAU .....(((.((((((((..(((((((((((((......(((((((((...)))))))))...)).)))))))).)))...))))))))(((((.....)))))....))).... ( -51.20, z-score = -3.57, R) >droWil1.scaffold_180700 2890758 114 + 6630534 GCUGAUGAAGAGUUCCAUUAACCUGCUUCUCACACAAUUGGAAAAGAAUUUCGAUUGCAUUCUGCAACAAGAAAGAUUUCUUAAUUGCAUUUGUCGCUCAGAUGUGGGUGAUAU ..(((.((((.(((.....)))...)))))))..(((((((((.....))))))))).....(((((.(((((....)))))..)))))..(((((((((....))))))))). ( -25.50, z-score = -0.26, R) >droVir3.scaffold_12875 13481033 114 - 20611582 GCUGCUGCAGCGCUGCAUUGGACUGCUGCUCAUUCAGCUCGAAGCCAACUUCGACUGCAUUAUGCAGCAGCAAAAGUUUCUCGACUGCAUCUGCCGCUCGGAUGUGGGCGACAU ((((((((((.((.(((......))).)).)...(((.((((((....))))))))).....))))))))).........(((.(..((((((.....))))))..).)))... ( -45.40, z-score = -1.80, R) >droMoj3.scaffold_6496 6918714 114 - 26866924 GCUGAUGCAGCGCUGCAUUGGGCUGCUGCUGCUGCAGCUGCAGACGAACUUCGACUGCGUUCUGCAGCAGCAGAAGUUUCUGGAGUGCAUCUGCCGCUCGGAUGUGGGCGACAU ((.(((((...(((.((..(((((.((((((((((((.(((((.((.....)).)))))..)))))))))))).))))).)).)))))))).))((((((....)))))).... ( -55.60, z-score = -2.11, R) >droGri2.scaffold_15245 572834 114 + 18325388 UUUGAUGCAACGCUGCAUUGGACUACUCCUGAUGCAGCUGCAAUCCAAUUUCGAUUGCAUUAUGCAGCAACAGAAAUUUCUCGAGUGCAUUUGUCGCUCGGAUGUGGGCGACAU .....((((..(((((((((((....))).))))))))(((((((.......)))))))...))))(((...((......))...)))...(((((((((....))))))))). ( -43.20, z-score = -3.51, R) >anoGam1.chrU 54660856 101 - 59568033 -----GUCGAUGCAUCAUCAG-CUGCUGCUUCUGCAAUCGUACGUCCGCUGUGAC----UUUCGCUGCUUC-CUGGUGGCUGGGAUGGAUUU--CGAGCGGAUGUUAAAGAUGU -----((.((.(((.((....-.)).))).)).)).(((..(((((((((.(((.----.(((((..((..-..))..))......)))..)--)))))))))))....))).. ( -29.70, z-score = 0.33, R) >consensus GAUGAUGCAGCGCUCCAUUGGCCUGCUGCUCAUUCAAUUGCAGUCGAAUUUUGACUGCAUUCUGCAGCAGCAGAGCUUCCUGGAGUGCAUCUGUCGCUCGGAUGUGGGCGACAU ......((((((...........)))))).......................(.((((.....)))))..................(((((((.....)))))))......... (-14.11 = -14.28 + 0.17)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:44:21 2011