| Sequence ID | dm3.chr2R |
|---|---|
| Location | 18,678,607 – 18,678,719 |
| Length | 112 |
| Max. P | 0.938390 |

| Location | 18,678,607 – 18,678,719 |
|---|---|
| Length | 112 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.23 |
| Shannon entropy | 0.09993 |
| G+C content | 0.34639 |
| Mean single sequence MFE | -33.02 |
| Consensus MFE | -27.61 |
| Energy contribution | -27.69 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.51 |
| Structure conservation index | 0.84 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.45 |
| SVM RNA-class probability | 0.938390 |
| Prediction | RNA |
| WARNING | Out of training range. z-scores are NOT reliable. |
Download alignment: ClustalW | MAF
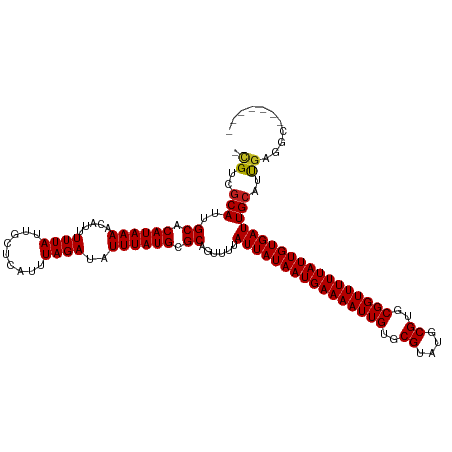
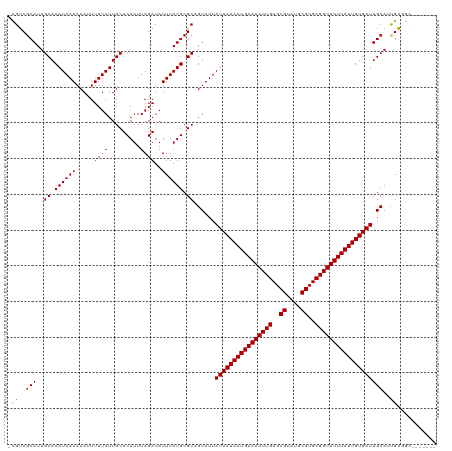
>dm3.chr2R 18678607 112 + 21146708 -CGUCGCAUUGCACAUAAAACACUUUUAUUGCUCAUUUAGAUAUUUAUGCGCAGUUUUAUUAUAAUGAAAAUUGUGCGUAUGCGUGCGGUUUUUAUUGUGAUUGCAUCGAGGC------- -((..(((((((.((((((.....((((.........))))..)))))).)))))...((((((((((((((((..((....))..)))))))))))))))).))..))....------- ( -31.40, z-score = -1.77, R) >droAna3.scaffold_13266 15024252 112 + 19884421 -CGUCGCAUUGCACAUAAAACAUUUUUAUUGCUCAUUUAGAUAUUUAUGCGCAGUUUUAUUAUAAUGAAAAUUGUGCGUAUGCGUGCGGUUUUUAUUGUGAUUGCAUUGAGGC------- -.(((.((.((((..............(((((.(((..(....)..))).)))))...((((((((((((((((..((....))..)))))))))))))))))))).)).)))------- ( -33.20, z-score = -2.48, R) >droEre2.scaffold_4845 20041748 112 + 22589142 -CGUCGCAUUGCACAUAAAACACUUUUAUUGCUCAUUUAGAUAUUUAUGCGCAGUUUUAUUAUAAUGAAAAUUGUGCGUAUGCGUGCGGUUUUUAUUGUGAUUGCAUCGAGGC------- -((..(((((((.((((((.....((((.........))))..)))))).)))))...((((((((((((((((..((....))..)))))))))))))))).))..))....------- ( -31.40, z-score = -1.77, R) >droYak2.chr2R 16145137 112 + 21139217 -CGUCGCAUUGCACAUAAAACACUUUUAUUGCUCAUUUAGAUAUUUAUGCGCAGUUUUAUUAUAAUGAAAAUUGUGCGUAUGCGUGCGGUUUUUAUUGUGAUUGCAUCGAGGC------- -((..(((((((.((((((.....((((.........))))..)))))).)))))...((((((((((((((((..((....))..)))))))))))))))).))..))....------- ( -31.40, z-score = -1.77, R) >droSec1.super_9 1980076 112 + 3197100 -CGUCGCAUUGCACAUAAAACACUUUUAUUGCUCAUUUAGAUAUUUAUGCGCAGUUUUAUUAUAAUGAAAAUUGUGCGUAUGCGUGCGGUUUUUAUUGUGAUUGCAUCGAGGC------- -((..(((((((.((((((.....((((.........))))..)))))).)))))...((((((((((((((((..((....))..)))))))))))))))).))..))....------- ( -31.40, z-score = -1.77, R) >droSim1.chr2R 17274113 112 + 19596830 -CGUCGCAUUGCACAUAAAACACUUUUAUUGCUCAUUUAGAUAUUUAUGCGCAGUUUUAUUAUAAUGAAAAUUGUGCGUAUGCGUGCGGUUUUUAUUGUGAUUGCAUCGAGGC------- -((..(((((((.((((((.....((((.........))))..)))))).)))))...((((((((((((((((..((....))..)))))))))))))))).))..))....------- ( -31.40, z-score = -1.77, R) >droWil1.scaffold_180700 3861622 113 + 6630534 CUGUCUCAUUGCACAUAAAACAUUUUUAUUGCUCAUUUAGAUAUUUAUGCGCAGUUUUAUUAUAAUGAAAAUUGUGCGUAUGCGUGCGGUUUUUAUUGUGAUUGCAUUGAGAC------- ..((((((.((((..............(((((.(((..(....)..))).)))))...((((((((((((((((..((....))..)))))))))))))))))))).))))))------- ( -37.90, z-score = -5.01, R) >dp4.chr3 4159118 112 + 19779522 -CGUCGCAUUGCACAUAAAACAUUUUUAUUGCUCAUUUAGAUAUUUAUGCGCAGUUUUAUUAUAAUGAAAAUUGUGCGUAUGCGUGCGGUUUUUAUUGUGAUUGCAUUGAGGC------- -.(((.((.((((..............(((((.(((..(....)..))).)))))...((((((((((((((((..((....))..)))))))))))))))))))).)).)))------- ( -33.20, z-score = -2.48, R) >droPer1.super_2 4346463 112 + 9036312 -CGUCGCAUUGCACAUAAAACAUUUUUAUUGCUCAUUUAGAUAUUUAUGCGCAGUUUUAUUAUAAUGAAAAUUGUGCGUAUGCGUGCGGUUUUUAUUGUGAUUGCAUUGAGGC------- -.(((.((.((((..............(((((.(((..(....)..))).)))))...((((((((((((((((..((....))..)))))))))))))))))))).)).)))------- ( -33.20, z-score = -2.48, R) >droVir3.scaffold_12875 13924909 111 + 20611582 --GUCGCAUUGCACAUAAAACAUUUUUAUUGCUCAUUUAGAUAUUUAUGCGCUGUUUUAUUAUAAUGAAAAUUGUGCGUAUGCGAGCGGUUUUUAUUGUGAUUGCAGUGGACU------- --(((.(((((((..(((((((........((.(((..(....)..))).)))))))))((((((((((((((((.((....)).)))))))))))))))).)))))))))).------- ( -32.90, z-score = -2.84, R) >droMoj3.scaffold_6496 9354296 119 + 26866924 -CGUCGCAUUGCACAUAAAACAUUUUUAUUGCUCAUUUAGAUAUUUAUGCGCUGUUUUAUUAUAAUGAAAAUUGUGCGUAUGCGUGCGGUUUUUAUUGUGAUUGCACUGGACCACCAACA -.(((.((.((((..(((((((........((.(((..(....)..))).)))))))))(((((((((((((((..((....))..))))))))))))))).)))).)))))........ ( -32.60, z-score = -2.17, R) >droGri2.scaffold_15245 1009321 112 - 18325388 -CGUCGCAUUGCACAUAAAACAUUUUUAUUGCUCAUUUAGAUAUUUAUGCGCUGUUUUAUUAUAAUGAAAAUUGUGCGUAUGCGUGCGGUUUUUAUUGUGAUUGCAGUGGACC------- -.(((.(((((((..(((((((........((.(((..(....)..))).)))))))))(((((((((((((((..((....))..))))))))))))))).)))))))))).------- ( -36.30, z-score = -3.78, R) >consensus _CGUCGCAUUGCACAUAAAACAUUUUUAUUGCUCAUUUAGAUAUUUAUGCGCAGUUUUAUUAUAAUGAAAAUUGUGCGUAUGCGUGCGGUUUUUAUUGUGAUUGCAUUGAGGC_______ ..(.(((((.(((.(((((.....))))))))((.....)).....)))))).((...((((((((((((((((..((....))..)))))))))))))))).))............... (-27.61 = -27.69 + 0.08)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:44:05 2011