
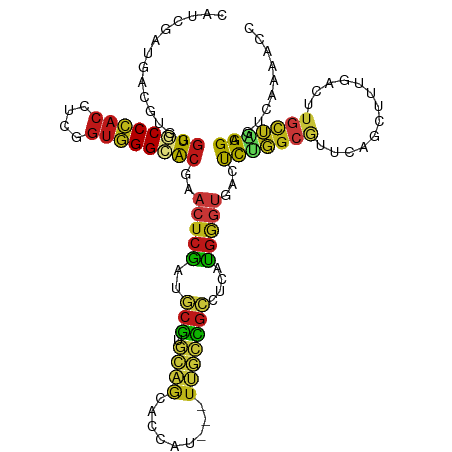
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 18,612,786 – 18,612,899 |
| Length | 113 |
| Max. P | 0.945410 |

| Location | 18,612,786 – 18,612,899 |
|---|---|
| Length | 113 |
| Sequences | 15 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 78.72 |
| Shannon entropy | 0.50625 |
| G+C content | 0.55906 |
| Mean single sequence MFE | -42.01 |
| Consensus MFE | -23.15 |
| Energy contribution | -23.44 |
| Covariance contribution | 0.29 |
| Combinations/Pair | 1.74 |
| Mean z-score | -1.78 |
| Structure conservation index | 0.55 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.51 |
| SVM RNA-class probability | 0.945410 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
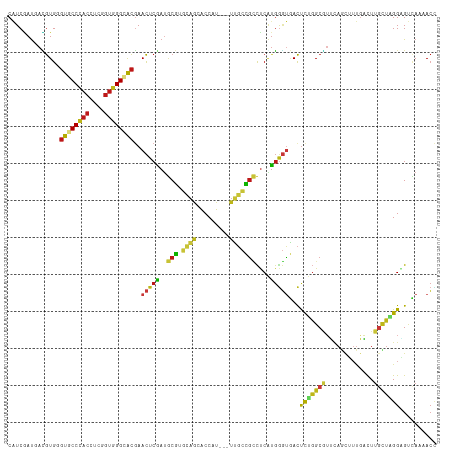
>dm3.chr2R 18612786 113 + 21146708 CAUCGAUCACGUGGGUGCCCACCUCGGUGGGCACGAACUCGAUGCGAGCAGCACCAU---UUGCCGCCUCGUGGGUGACUCUGGCGUUCAGCUUUGACUUGCUAGGAGUCAAAACU ......((((((..((((((((....))))))))..))..((.(((.((((......---))))))).)))))).(((((((((((.((......))..))))).))))))..... ( -46.10, z-score = -2.18, R) >droSim1.chr2R 17208239 113 + 19596830 CAUCGAUCACGUGGGUGCCCACCUCGGUGGGCACGAACUCGAUGCGAGCAGCACCAU---UUGCCGCCUCGUGGGUGACUCUGGCGUUCAGCUUUGACUUGCUAGGAGUCAAAACC .(((((....((..((((((((....))))))))..)))))))((((((.(((....---.))).).)))))((.(((((((((((.((......))..))))).))))))...)) ( -46.70, z-score = -2.33, R) >droSec1.super_9 1914850 113 + 3197100 CAUCGAUCACGUGGGUGCCCACCUCGGUGGGCACGAACUCGAUGCGAGCAGCACCAU---UUGCCGCCUCGUGGGUGACUCUGGCGUUCAGCUUUGACUUGCUAGGAGUCAAAACC .(((((....((..((((((((....))))))))..)))))))((((((.(((....---.))).).)))))((.(((((((((((.((......))..))))).))))))...)) ( -46.70, z-score = -2.33, R) >droYak2.chr2R 16078462 113 + 21139217 CAUCGAUCACGUGGGUGCCCACCUCGGUGGGCACGAACUCGAUGCGAGCAGCACCAU---UUGCCGCCUCAUGGGUGACUCUGGCGUUCAGCUUUGACUUGCUAGGAGUCAAAACC ((((((....((..((((((((....))))))))..)))))))).((((.(((....---.))).).)))..((.(((((((((((.((......))..))))).))))))...)) ( -45.40, z-score = -2.27, R) >droEre2.scaffold_4845 19974604 113 + 22589142 CAUCGAUCACGUGGGUGCCCACCUCGGUGGGCACGAACUCGAUGCGAGCAGCACCAU---UUGCCGCCUCAUGGGUGACUCUGGCGUUCAGCUUUGACUUGCUAGGAGUCAAAACC ((((((....((..((((((((....))))))))..)))))))).((((.(((....---.))).).)))..((.(((((((((((.((......))..))))).))))))...)) ( -45.40, z-score = -2.27, R) >droAna3.scaffold_13266 14953782 113 + 19884421 CAUCGAUGACGUGGGUGCCCACCUCUGUGGGCACGAACUCGAUGCGUGCAGCACCAU---UUGCCGCCUCGUGGGUGACUCUGGCGUUGAGCUUUGACUUACUGGGAGUCAAAACC ((((((....((..((((((((....))))))))..)))))))).((.((((.(((.---..(.((((.....)))).)..))).)))).))((((((((.....))))))))... ( -43.90, z-score = -1.81, R) >dp4.chr3 4092486 113 + 19779522 CAUCGAUGACGUGCGUUCCCACUUCCGUGGGGACGAACUCGAUGCGUGCAGCACCAU---UUGCCGCCUCAUGGGUGACUCUGGCGUUCAGCUUUGACUUGUUGGGAGUCAAAACC ((((.((((((..(((((((((....)))))))))....))..(((.((((......---))))))).)))).))))....((((.((((((........)))))).))))..... ( -41.10, z-score = -1.37, R) >droPer1.super_2 4279544 113 + 9036312 CAUCGAUGACGUGCGUUCCCACUUCCGUGGGGACGAACUCGAUGCGUGCAGCACCAU---UUGCCGCCUCAUGGGUGACUCUGGCGUUCAGCUUUGACUUGUUGGGAGUCAAAACC ((((.((((((..(((((((((....)))))))))....))..(((.((((......---))))))).)))).))))....((((.((((((........)))))).))))..... ( -41.10, z-score = -1.37, R) >droWil1.scaffold_180700 2652387 113 - 6630534 CAUCGAUAAUGUGGGUACCCACCUCGGUGGGGACAAACUCAAUGCGUGCAGCACCAU---UUGCCGCCUCAUGGGUGACUCUGGCAUUCAGUUUUGACUUGUUUGGCGUCAAUACC ..........((((((.(((((....))))).))........(((.....)))))))---(((.((((..(..(((.(..(((.....)))...).)))..)..)))).))).... ( -32.70, z-score = 0.32, R) >droMoj3.scaffold_6496 9245244 113 + 26866924 CAUCAAUGAUGUGCGUGCCCACCUCGGUGGGCACGAACUCAAUGCGUGCAGCACCAU---UUGCCGCCUCAUGGGUGACUCUGGCAUUCAGCUUUGACUUGUUUGGGGUCAAGACC ......(((.((.(((((((((....))))))))).)))))..(((.((((......---))))))).....((.((((((..(((.((......))..)).)..))))))...)) ( -42.50, z-score = -1.41, R) >droVir3.scaffold_12875 13832415 113 + 20611582 CAUCAAUGAUGUGCGUGCCCACUUCGGUGGGCACGAACUCAAUGCGUGCAGCACCAU---UUGCCGCCUCAUGGGUGACUCUGGCAUUCAGCUUUGACUUGCUUGGAGUCAAGACC ......(((.((.(((((((((....))))))))).)))))..(((.((((......---))))))).....((.(((((((((((.((......))..))).))))))))...)) ( -46.60, z-score = -2.95, R) >droGri2.scaffold_15245 922884 113 - 18325388 CAUCAAUGAUGUGCGUGCCCACUUCGGUGGGCACGAACUCAAUGCGUGCAGCACCAU---UUGCCGCCUCAUGGGUGACUCUGGCAUUCAGCUUUGACUUGUUGGGUGUCAAAACC ((((.((((.((.(((((((((....))))))))).)).....(((.((((......---))))))).)))).))))....(((((((((((........)))))))))))..... ( -49.30, z-score = -3.77, R) >anoGam1.chr2L 40172616 113 + 48795086 CUUCGAUAAUGUGCGUGCCGACCUCCGACGGGACGAACUCGAUCCGGAGCGAACCGU---UGGCCGACUCGUGCGUCACCCGGGCGGACACUUUGGUCUUGCUCGGGGAGAGCACG ..........((.((.((((((.(((..((((.((....)).))))..).))...))---)))))))).(((((.((.(((((((((((......))).))))))))))..))))) ( -47.50, z-score = -1.12, R) >apiMel3.Group10 2588888 116 + 11440700 CUUCUACAAGAUGACUACCUACUUCUAUUGGAGUGAAUUCAACACGAAAUUCUCCAGAUCUUCCUGAUACUUCAUCAAUUUUUGCUUGUAUAUAUCUUUUAGAAGGUGCUAAAAAU ((((((.((((((..(((.........((((((.((.(((.....))).)))))))).......((((.....))))..........)))..)))))).))))))........... ( -20.80, z-score = -1.27, R) >triCas2.ChLG3 14294167 112 + 32080666 CUUCAAUGAUGUGAGUCCCGACCUCCGUCGGCACAAACUCCACACUUUGAACUCCGG---UGCGAUUUCCGGGGAGCACUCUCGCCGGCACCGACCUUUUGCUGGGCGCAAUCAC- .(((((.(.(((((((.(((((....))))).....))).))))).)))))...(((---((((....)(((.(((....))).))))))))).....((((.....))))....- ( -34.30, z-score = -0.64, R) >consensus CAUCGAUGACGUGGGUGCCCACCUCGGUGGGCACGAACUCGAUGCGUGCAGCACCAU___UUGCCGCCUCAUGGGUGACUCUGGCGUUCAGCUUUGACUUGCUAGGAGUCAAAACC ..............((((((((....))))))))..(((((..(((.((((.........)))))))....)))))...(((((((.............))))))).......... (-23.15 = -23.44 + 0.29)

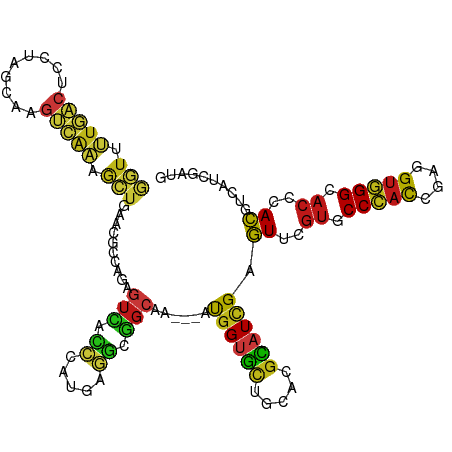
| Location | 18,612,786 – 18,612,899 |
|---|---|
| Length | 113 |
| Sequences | 15 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 78.72 |
| Shannon entropy | 0.50625 |
| G+C content | 0.55906 |
| Mean single sequence MFE | -41.17 |
| Consensus MFE | -20.46 |
| Energy contribution | -20.15 |
| Covariance contribution | -0.31 |
| Combinations/Pair | 1.71 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.50 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.10 |
| SVM RNA-class probability | 0.890717 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
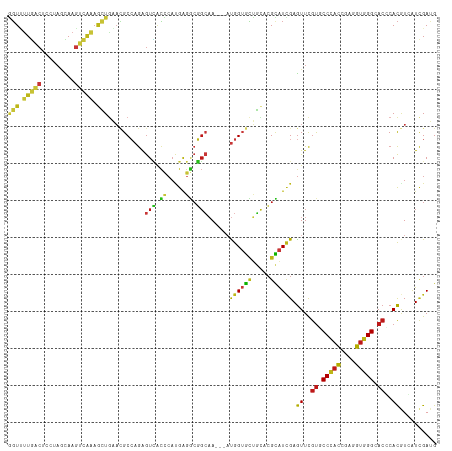
>dm3.chr2R 18612786 113 - 21146708 AGUUUUGACUCCUAGCAAGUCAAAGCUGAACGCCAGAGUCACCCACGAGGCGGCAA---AUGGUGCUGCUCGCAUCGAGUUCGUGCCCACCGAGGUGGGCACCCACGUGAUCGAUG ((((((((((.......))))))))))...(((((..(((.((.....)).)))..---.)))))....(((.((((.((..((((((((....))))))))..)).))))))).. ( -45.00, z-score = -2.49, R) >droSim1.chr2R 17208239 113 - 19596830 GGUUUUGACUCCUAGCAAGUCAAAGCUGAACGCCAGAGUCACCCACGAGGCGGCAA---AUGGUGCUGCUCGCAUCGAGUUCGUGCCCACCGAGGUGGGCACCCACGUGAUCGAUG ((((((((((.......))))))))))...(((((..(((.((.....)).)))..---.)))))....(((.((((.((..((((((((....))))))))..)).))))))).. ( -45.00, z-score = -2.23, R) >droSec1.super_9 1914850 113 - 3197100 GGUUUUGACUCCUAGCAAGUCAAAGCUGAACGCCAGAGUCACCCACGAGGCGGCAA---AUGGUGCUGCUCGCAUCGAGUUCGUGCCCACCGAGGUGGGCACCCACGUGAUCGAUG ((((((((((.......))))))))))...(((((..(((.((.....)).)))..---.)))))....(((.((((.((..((((((((....))))))))..)).))))))).. ( -45.00, z-score = -2.23, R) >droYak2.chr2R 16078462 113 - 21139217 GGUUUUGACUCCUAGCAAGUCAAAGCUGAACGCCAGAGUCACCCAUGAGGCGGCAA---AUGGUGCUGCUCGCAUCGAGUUCGUGCCCACCGAGGUGGGCACCCACGUGAUCGAUG ((((((((((.......))))))))))...(((((..(((.((.....)).)))..---.)))))....(((.((((.((..((((((((....))))))))..)).))))))).. ( -45.00, z-score = -2.33, R) >droEre2.scaffold_4845 19974604 113 - 22589142 GGUUUUGACUCCUAGCAAGUCAAAGCUGAACGCCAGAGUCACCCAUGAGGCGGCAA---AUGGUGCUGCUCGCAUCGAGUUCGUGCCCACCGAGGUGGGCACCCACGUGAUCGAUG ((((((((((.......))))))))))...(((((..(((.((.....)).)))..---.)))))....(((.((((.((..((((((((....))))))))..)).))))))).. ( -45.00, z-score = -2.33, R) >droAna3.scaffold_13266 14953782 113 - 19884421 GGUUUUGACUCCCAGUAAGUCAAAGCUCAACGCCAGAGUCACCCACGAGGCGGCAA---AUGGUGCUGCACGCAUCGAGUUCGUGCCCACAGAGGUGGGCACCCACGUCAUCGAUG ((((((((((.......))))))))))((.(((((..(((.((.....)).)))..---.))))).))....((((((((..((((((((....))))))))..))....)))))) ( -42.60, z-score = -2.33, R) >dp4.chr3 4092486 113 - 19779522 GGUUUUGACUCCCAACAAGUCAAAGCUGAACGCCAGAGUCACCCAUGAGGCGGCAA---AUGGUGCUGCACGCAUCGAGUUCGUCCCCACGGAAGUGGGAACGCACGUCAUCGAUG ((((((((((.......))))))))))...(((((..(((.((.....)).)))..---.))))).......((((((((.(((.(((((....))))).))).))....)))))) ( -43.30, z-score = -2.55, R) >droPer1.super_2 4279544 113 - 9036312 GGUUUUGACUCCCAACAAGUCAAAGCUGAACGCCAGAGUCACCCAUGAGGCGGCAA---AUGGUGCUGCACGCAUCGAGUUCGUCCCCACGGAAGUGGGAACGCACGUCAUCGAUG ((((((((((.......))))))))))...(((((..(((.((.....)).)))..---.))))).......((((((((.(((.(((((....))))).))).))....)))))) ( -43.30, z-score = -2.55, R) >droWil1.scaffold_180700 2652387 113 + 6630534 GGUAUUGACGCCAAACAAGUCAAAACUGAAUGCCAGAGUCACCCAUGAGGCGGCAA---AUGGUGCUGCACGCAUUGAGUUUGUCCCCACCGAGGUGGGUACCCACAUUAUCGAUG ((((.((((.........))))(((((.(((((.....(((....))).((((((.---....))))))..))))).)))))...(((((....)))))))))............. ( -34.30, z-score = -0.79, R) >droMoj3.scaffold_6496 9245244 113 - 26866924 GGUCUUGACCCCAAACAAGUCAAAGCUGAAUGCCAGAGUCACCCAUGAGGCGGCAA---AUGGUGCUGCACGCAUUGAGUUCGUGCCCACCGAGGUGGGCACGCACAUCAUUGAUG (((....)))........(((((((((.(((((.....(((....))).((((((.---....))))))..))))).))))(((((((((....))))))))).......))))). ( -41.80, z-score = -1.69, R) >droVir3.scaffold_12875 13832415 113 - 20611582 GGUCUUGACUCCAAGCAAGUCAAAGCUGAAUGCCAGAGUCACCCAUGAGGCGGCAA---AUGGUGCUGCACGCAUUGAGUUCGUGCCCACCGAAGUGGGCACGCACAUCAUUGAUG ((...((((((...(((..(((....))).)))..)))))).)).....((((((.---....))))))...((.(((((.(((((((((....))))))))).)).))).))... ( -43.60, z-score = -2.20, R) >droGri2.scaffold_15245 922884 113 + 18325388 GGUUUUGACACCCAACAAGUCAAAGCUGAAUGCCAGAGUCACCCAUGAGGCGGCAA---AUGGUGCUGCACGCAUUGAGUUCGUGCCCACCGAAGUGGGCACGCACAUCAUUGAUG (((((((((.........))))))))).(((((.....(((....))).((((((.---....))))))..)))))((((.(((((((((....))))))))).)).))....... ( -41.80, z-score = -2.09, R) >anoGam1.chr2L 40172616 113 - 48795086 CGUGCUCUCCCCGAGCAAGACCAAAGUGUCCGCCCGGGUGACGCACGAGUCGGCCA---ACGGUUCGCUCCGGAUCGAGUUCGUCCCGUCGGAGGUCGGCACGCACAUUAUCGAAG (((((..((((((.((..(((......))).)).)))).)).))))).(((((((.---.(((((((...)))))))..((((......)))))))))))................ ( -41.30, z-score = -0.14, R) >apiMel3.Group10 2588888 116 - 11440700 AUUUUUAGCACCUUCUAAAAGAUAUAUACAAGCAAAAAUUGAUGAAGUAUCAGGAAGAUCUGGAGAAUUUCGUGUUGAAUUCACUCCAAUAGAAGUAGGUAGUCAUCUUGUAGAAG ...........((((((.(((((...(((..((.....((((((...)))))).......(((((...(((.....)))....)))))......))..)))...))))).)))))) ( -21.90, z-score = -0.38, R) >triCas2.ChLG3 14294167 112 - 32080666 -GUGAUUGCGCCCAGCAAAAGGUCGGUGCCGGCGAGAGUGCUCCCCGGAAAUCGCA---CCGGAGUUCAAAGUGUGGAGUUUGUGCCGACGGAGGUCGGGACUCACAUCAUUGAAG -....((((.....))))..(((((((.((((.(((....))).))))..)))).)---))....(((((.((((((.....((.(((((....))))).))))))))..))))). ( -38.60, z-score = -0.27, R) >consensus GGUUUUGACUCCUAGCAAGUCAAAGCUGAACGCCAGAGUCACCCAUGAGGCGGCAA___AUGGUGCUGCACGCAUCGAGUUCGUGCCCACCGAGGUGGGCACCCACGUCAUCGAUG (((.(((((.........))))).)))..........(((.((.....)).)))......((((((.....)))))).((..((.(((((....))))).))..)).......... (-20.46 = -20.15 + -0.31)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:44:02 2011