
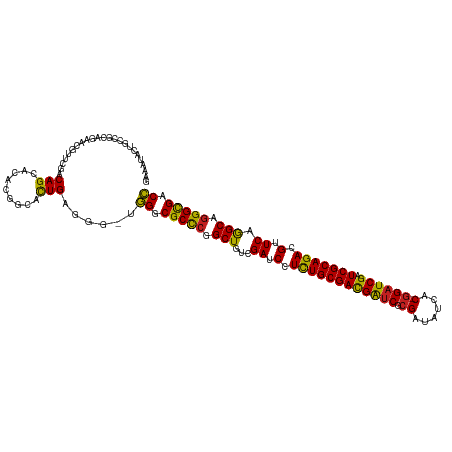
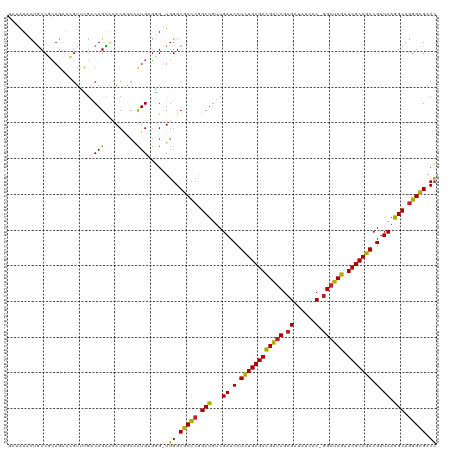
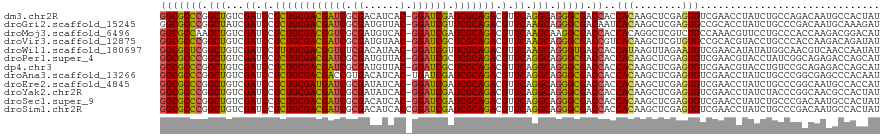
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 18,278,917 – 18,279,081 |
| Length | 164 |
| Max. P | 0.983504 |

| Location | 18,278,917 – 18,279,035 |
|---|---|
| Length | 118 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.05 |
| Shannon entropy | 0.35056 |
| G+C content | 0.62449 |
| Mean single sequence MFE | -51.41 |
| Consensus MFE | -33.82 |
| Energy contribution | -33.09 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.10 |
| Structure conservation index | 0.66 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.34 |
| SVM RNA-class probability | 0.929208 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
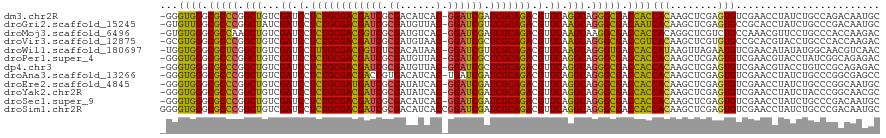
>dm3.chr2R 18278917 118 + 21146708 GAAAUCCUGCCCCAGAGGGUUCGACAGCACACCGCACUGCGGG-UGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGACAUCAC-GGAUCGAUCGCAGACGUUCAGGCAGGGCGACC ....(((((((..(((((((.(((((((((.((((...)))))-)((....)).))))))))))))))(((((((((.((......)-)))))).)))).........)))))))..... ( -58.10, z-score = -3.37, R) >droGri2.scaffold_15245 6816407 118 + 18325388 CAGAUAUUGCCGGACAAAGUGGAGCAGUAUACAACGCUGAGUG-UUGGCGCCCGGCUAUCGAUCCUCUGCGACGAUCGCGAUGUUAC-GGAUCGUUCGCAGACGUUCAAGCAGGGCGACA ...((((((((.(......).).))))))).((((((...)))-))).(((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).)))))... ( -45.90, z-score = -2.05, R) >droMoj3.scaffold_6496 13012540 118 + 26866924 GAGGUGUUGCCGCAGGGCGUGAGGCAGCACACAGCUCUGAGUG-UGGGCGCCAAGCUGUCGAUCCUCUGCGACGGUCGCGAUGUCAC-GGAUCGAUCGCAGACGUUCAAGCAAGGCGACC ...((((((((.((.....)).))))))))...((((......-.))))(((..(((...((.(.((((((((((((.((......)-)))))).))))))).).)).)))..))).... ( -49.10, z-score = -0.42, R) >droVir3.scaffold_12875 19316131 118 + 20611582 GUGGUGUUGCCGCAGGACGUGAGGCAGCAUACAAAACUGAGCG-UGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUGUAAC-GGAUCGCUCGCAGACGUUCAAGCAGGGCGACC (((.(((((((.((.....)).))))))))))...........-..(((((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).))))).)) ( -50.80, z-score = -1.40, R) >droWil1.scaffold_180697 4079280 118 - 4168966 UUGGUAUUGCCAGAACAAAUUCGACAGCAUACAUCGUUGAUGG-UGGGCGCUCGGCUGUCGAUCCUUUGCGACGGUCUCGACAUAAC-GGAUCGUUCGCAGACGUUCAAGCAGGGUGACC .......(((..((((....((((((((...(((((....)))-))........))))))))...((((((((((((.((......)-)))))).))))))).))))..)))((....)) ( -42.50, z-score = -1.79, R) >droPer1.super_4 4198003 117 + 7162766 -CCAUACUUCCGCAGAACGUUCGACAGAUAACGACACUGAGGG-UGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUGUUAC-GGAUCGCUCGCAGACGUUCAGGCAGGGCGACC -((((....((.(((..((((........))))...))).)))-))).(((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).)))))... ( -45.10, z-score = -1.08, R) >dp4.chr3 4925338 118 - 19779522 GCCAUACUUCCGCAGAACGUUCGACAGAUAACGACACUGAGGG-UGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUGUUAC-GGAUCGCUCGCAGACGUUCAGGCAGGGCGACC (((.....(((.(((..((((........))))...))).)))-..)))((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).)))).... ( -46.70, z-score = -1.26, R) >droAna3.scaffold_13266 12407665 118 - 19884421 GUUAUCCUGCCGCAGGACGUUCGACAGCGCACAACUCUGAGGG-UGGGCGCCCGGCUGUCGAUCCUCUGCGACGACCGUGACAUCAC-UGAUCGAUCGCAGACGUUCAGGCAGGGCGACC (((..((((((...(((((.((((((((...((....)).(((-(....)))).))))))))...(((((((((((.(((....)))-.).))).)))))))))))).))))))..))). ( -57.90, z-score = -3.68, R) >droEre2.scaffold_4845 12419287 118 + 22589142 GAAAUCCUGCCCCAGAGGGUUCGACAGCACACGGCACUGCGGG-UGGGCGCCCGGCUGUCGAUCCUCUGCGAUGAUCGCGAUAUCAC-GGAUCGAUCGCAGACGUUCAGGCAGGGCGACC ....(((((((..(((((((.(((((((...((....))((((-(....)))))))))))))))))))(((((((((.((......)-))))).))))).........)))))))..... ( -55.20, z-score = -2.55, R) >droYak2.chr2R 18028236 118 + 21139217 GAAAUACUGCCCCAGAUGGUUCGACAGCACACGGCACUGCGGG-UGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUAUCAC-GGAUCGAUCGCAGACGUUCAGGCAGGGCGACC ......(((((......((.((((((((...((....))((((-(....))))))))))))).))((((((((((((.((......)-)))))).)))))))......)))))....... ( -52.30, z-score = -2.33, R) >droSec1.super_9 1587078 118 + 3197100 GAAAUCCUGCCCCAGAGGGUUCGACAGCACACGGCACUGCGGG-UGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGACAUCAC-GGAUCGAUCGCAGACGUUCAGGCAGGGCGACC ....(((((((..(((((((.(((((((...((....))((((-(....)))))))))))))))))))(((((((((.((......)-)))))).)))).........)))))))..... ( -56.60, z-score = -2.86, R) >droSim1.chr2R 16876962 120 + 19596830 GAAAUCCUGCCCCAGAGGGUUCGACAGCACACGGCACUGCGGGGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGACAUCACCGGAUCGAUCGCAGACGUUCAGGCAGGGCGACC ....(((((((..(((((((.(((((((...((......))((((....)))).))))))))))))))(((((((((.((.......))))))).)))).........)))))))..... ( -56.70, z-score = -2.42, R) >consensus GAAAUACUGCCGCAGAACGUUCGACAGCACACGGCACUGAGGG_UGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUAUCAC_GGAUCGAUCGCAGACGUUCAGGCAGGGCGACC ........................(((.........)))......((.(((((.(((...((.(.((((((((((((............))))).))))))).).)).))).))))).)) (-33.82 = -33.09 + -0.72)

| Location | 18,278,917 – 18,279,035 |
|---|---|
| Length | 118 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 84.05 |
| Shannon entropy | 0.35056 |
| G+C content | 0.62449 |
| Mean single sequence MFE | -51.40 |
| Consensus MFE | -34.58 |
| Energy contribution | -34.85 |
| Covariance contribution | 0.27 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.41 |
| Structure conservation index | 0.67 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.93 |
| SVM RNA-class probability | 0.975669 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 18278917 118 - 21146708 GGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC-GUGAUGUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCA-CCCGCAGUGCGGUGUGCUGUCGAACCCUCUGGGGCAGGAUUUC ......(((((((.(...(((((((.(((((.-(((....)))))))))))))))((.((((((((...(((((((-(.....))).))))))))))))).))...).)))))))..... ( -60.30, z-score = -3.14, R) >droGri2.scaffold_15245 6816407 118 - 18325388 UGUCGCCCUGCUUGAACGUCUGCGAACGAUCC-GUAACAUCGCGAUCGUCGCAGAGGAUCGAUAGCCGGGCGCCAA-CACUCAGCGUUGUAUACUGCUCCACUUUGUCCGGCAAUAUCUG ....((((.((((((.(.(((((((.((((((-(......)).)))))))))))).).)))..))).))))(((.(-((...((((........))))......)))..)))........ ( -43.30, z-score = -2.55, R) >droMoj3.scaffold_6496 13012540 118 - 26866924 GGUCGCCUUGCUUGAACGUCUGCGAUCGAUCC-GUGACAUCGCGACCGUCGCAGAGGAUCGACAGCUUGGCGCCCA-CACUCAGAGCUGUGUGCUGCCUCACGCCCUGCGGCAACACCUC ((.((((..((((((.(.(((((((.((...(-(((....))))..))))))))).).)))..)))..)))).)).-......(((.(((.((((((..........))))))))).))) ( -47.30, z-score = -1.67, R) >droVir3.scaffold_12875 19316131 118 - 20611582 GGUCGCCCUGCUUGAACGUCUGCGAGCGAUCC-GUUACAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCA-CGCUCAGUUUUGUAUGCUGCCUCACGUCCUGCGGCAACACCAC ((.(((((.((((((.(.(((((((.((((((-(......)).)))))))))))).).)))..))).))))).)).-..........(((.((((((..........))))))))).... ( -49.80, z-score = -2.49, R) >droWil1.scaffold_180697 4079280 118 + 4168966 GGUCACCCUGCUUGAACGUCUGCGAACGAUCC-GUUAUGUCGAGACCGUCGCAAAGGAUCGACAGCCGAGCGCCCA-CCAUCAACGAUGUAUGCUGUCGAAUUUGUUCUGGCAAUACCAA (((.....((((.(((((..(((((.((.(((-(......)).)).))))))).....((((((((.(......).-.((((...))))...))))))))...))))).))))..))).. ( -34.70, z-score = -0.88, R) >droPer1.super_4 4198003 117 - 7162766 GGUCGCCCUGCCUGAACGUCUGCGAGCGAUCC-GUAACAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCA-CCCUCAGUGUCGUUAUCUGUCGAACGUUCUGCGGAAGUAUGG- ((.(((((.((.(((.(.(((((((.((((((-(......)).)))))))))))).).)))...)).))))).)).-.((.(((...((((........))))..))).))........- ( -48.30, z-score = -1.98, R) >dp4.chr3 4925338 118 + 19779522 GGUCGCCCUGCCUGAACGUCUGCGAGCGAUCC-GUAACAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCA-CCCUCAGUGUCGUUAUCUGUCGAACGUUCUGCGGAAGUAUGGC ((.(((((.((.(((.(.(((((((.((((((-(......)).)))))))))))).).)))...)).))))).)).-.((.(((...((((........))))..))).))......... ( -48.30, z-score = -1.67, R) >droAna3.scaffold_13266 12407665 118 + 19884421 GGUCGCCCUGCCUGAACGUCUGCGAUCGAUCA-GUGAUGUCACGGUCGUCGCAGAGGAUCGACAGCCGGGCGCCCA-CCCUCAGAGUUGUGCGCUGUCGAACGUCCUGCGGCAGGAUAAC ......((((((......(((((((.(((((.-(((....)))))))))))))))(((((((((((.(((......-))).....(.....)))))))))...)))...))))))..... ( -52.00, z-score = -1.88, R) >droEre2.scaffold_4845 12419287 118 - 22589142 GGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC-GUGAUAUCGCGAUCAUCGCAGAGGAUCGACAGCCGGGCGCCCA-CCCGCAGUGCCGUGUGCUGUCGAACCCUCUGGGGCAGGAUUUC ......(((((((.(...((((((((.((((.-(((....)))))))))))))))((.(((((((((((((((...-......))))).)).)))))))).))...).)))))))..... ( -58.10, z-score = -3.50, R) >droYak2.chr2R 18028236 118 - 21139217 GGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC-GUGAUAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCA-CCCGCAGUGCCGUGUGCUGUCGAACCAUCUGGGGCAGUAUUUC .......((((((.(...(((((((.(((((.-(((....)))))))))))))))((.(((((((((((((((...-......))))).)).)))))))).))...).))))))...... ( -56.60, z-score = -3.28, R) >droSec1.super_9 1587078 118 - 3197100 GGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC-GUGAUGUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCA-CCCGCAGUGCCGUGUGCUGUCGAACCCUCUGGGGCAGGAUUUC ......(((((((.(...(((((((.(((((.-(((....)))))))))))))))((.(((((((((((((((...-......))))).)).)))))))).))...).)))))))..... ( -59.50, z-score = -3.31, R) >droSim1.chr2R 16876962 120 - 19596830 GGUCGCCCUGCCUGAACGUCUGCGAUCGAUCCGGUGAUGUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCCCGCAGUGCCGUGUGCUGUCGAACCCUCUGGGGCAGGAUUUC ......(((((((.(...(((((((.((((((((.....))).))))))))))))((.(((((((((((((((..........))))).)).)))))))).))...).)))))))..... ( -58.60, z-score = -2.56, R) >consensus GGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC_GUGAUAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCA_CCCUCAGUGCCGUGUGCUGUCGAACCUUCUGCGGCAGGAUUUC ((.(((((.((.(((.(.(((((((.(((((..((......)))))))))))))).).)))...)).))))).))...................((((...........))))....... (-34.58 = -34.85 + 0.27)

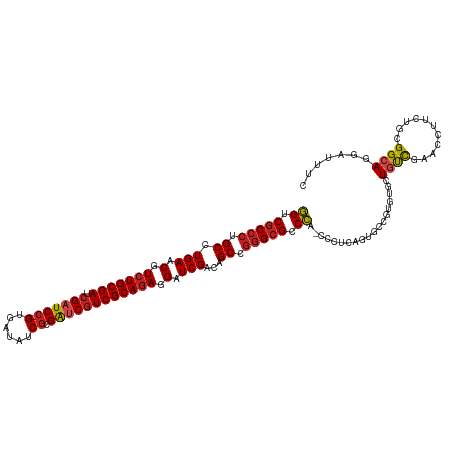
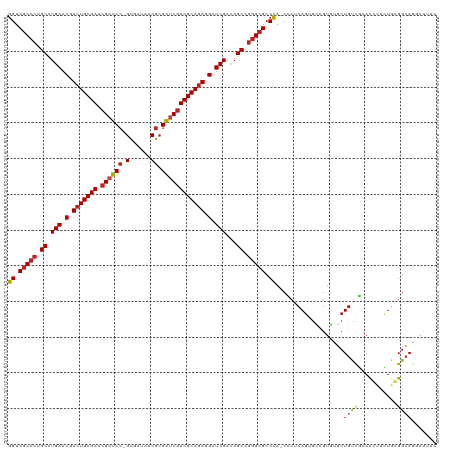
| Location | 18,278,957 – 18,279,075 |
|---|---|
| Length | 118 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 87.18 |
| Shannon entropy | 0.28480 |
| G+C content | 0.62409 |
| Mean single sequence MFE | -49.82 |
| Consensus MFE | -38.99 |
| Energy contribution | -38.07 |
| Covariance contribution | -0.92 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.89 |
| Structure conservation index | 0.78 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.17 |
| SVM RNA-class probability | 0.903466 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 18278957 118 + 21146708 -GGGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGACAUCAC-GGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUGCCAGACAAUGC -.(((((.(((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).))))).)))))....((.....((((..............))))..)) ( -49.84, z-score = -1.94, R) >droGri2.scaffold_15245 6816447 118 + 18325388 -GUGUUGGCGCCCGGCUAUCGAUCCUCUGCGACGAUCGCGAUGUUAC-GGAUCGUUCGCAGACGUUCAAGCAGGGCGACAAUCACAAGCUCGAGUGCCGCACCUAUCUGCCCGACAAUGC -.(((((((((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).)))))..........((......)).(((......))))))))).... ( -43.70, z-score = -1.18, R) >droMoj3.scaffold_6496 13012580 118 + 26866924 -GUGUGGGCGCCAAGCUGUCGAUCCUCUGCGACGGUCGCGAUGUCAC-GGAUCGAUCGCAGACGUUCAAGCAAGGCGACCACCACAGGCUCGUCUGCCAAACGUUCCUGCCCACCAAGAC -(.((((((.....((.((((((((...(((.....)))(....)..-)))))))).)).((((((...(((.(((((((......)).))))))))..))))))...)))))))..... ( -47.90, z-score = -2.20, R) >droVir3.scaffold_12875 19316171 118 + 20611582 -GCGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUGUAAC-GGAUCGCUCGCAGACGUUCAAGCAGGGCGACCGUCACAAGCUCGUGUGCCGCACGUACCUGCCCACCAAGAC -(.((((((((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).))))..........((..((((.....))))...)))))))))..... ( -51.10, z-score = -1.68, R) >droWil1.scaffold_180697 4079320 118 - 4168966 -UGGUGGGCGCUCGGCUGUCGAUCCUUUGCGACGGUCUCGACAUAAC-GGAUCGUUCGCAGACGUUCAAGCAGGGUGACCACCAUAAGUUAGAAUGUCGAACAUAUAUGGCAACGUCAAC -((((((.(((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).))))).)))))).......((.(((((........)))))...))... ( -42.20, z-score = -2.17, R) >droPer1.super_4 4198042 118 + 7162766 -GGGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUGUUAC-GGAUCGCUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACGUACCUAUCGGCAGAGAC -.(((((.(((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).))))).))))).....(((...((((((.........))))))))).. ( -53.70, z-score = -2.28, R) >dp4.chr3 4925378 118 - 19779522 -GGGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUGUUAC-GGAUCGCUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACGUACCUGUCCGCAGAGAC -.(((((.(((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).))))).)))))....(.(((.....))).)((..(((....)))..)) ( -51.40, z-score = -1.30, R) >droAna3.scaffold_13266 12407705 118 - 19884421 -GGGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGACCGUGACAUCAC-UGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUGCCCGGCGAGCC -.(((((.(((((.(((...((.(.(((((((((((.(((....)))-.).))).))))))).).)).))).))))).)))))....(((((.(.((.((.....)).)).)..))))). ( -53.40, z-score = -2.18, R) >droEre2.scaffold_4845 12419327 118 + 22589142 -GGGUGGGCGCCCGGCUGUCGAUCCUCUGCGAUGAUCGCGAUAUCAC-GGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUGCCCGGCAAUGC -.(((((.(((((.(((...((.(.((((((((((((.((......)-))))).)))))))).).)).))).))))).)))))....((.((.(.((.((.....)).)))))))..... ( -50.10, z-score = -1.53, R) >droYak2.chr2R 18028276 118 + 21139217 -GGGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUAUCAC-GGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUACCCGGCAACGC -.(((((.(((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).))))).)))))....((.((.(.((.((.....)).)))))))..... ( -51.20, z-score = -2.23, R) >droSec1.super_9 1587118 118 + 3197100 -GGGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGACAUCAC-GGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUGCCCGACAAUGC -.(((((.(((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).))))).)))))....((.....(((((............)))))..)) ( -51.70, z-score = -2.26, R) >droSim1.chr2R 16877002 120 + 19596830 GGGGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGACAUCACCGGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUGCCCGACAAUGC ..(((((.(((((.(((...((.(.((((((((((((.((.......))))))).))))))).).)).))).))))).)))))....((.....(((((............)))))..)) ( -51.60, z-score = -1.77, R) >consensus _GGGUGGGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUAUCAC_GGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUGCCCGCCAAGGC ...((((.(((((.(((...((.(.((((((((((((............))))).))))))).).)).))).))))).))))(((........)))........................ (-38.99 = -38.07 + -0.92)
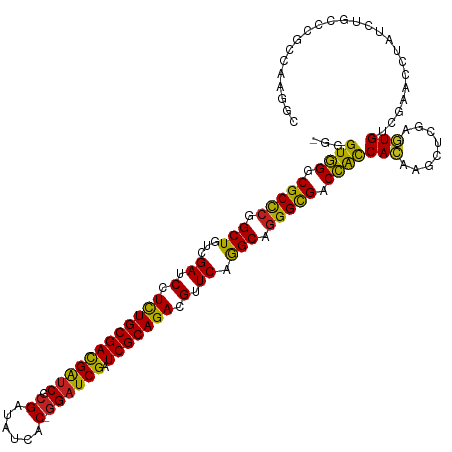
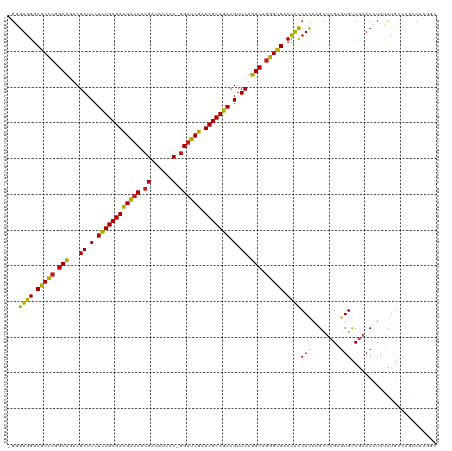
| Location | 18,278,957 – 18,279,075 |
|---|---|
| Length | 118 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.18 |
| Shannon entropy | 0.28480 |
| G+C content | 0.62409 |
| Mean single sequence MFE | -54.38 |
| Consensus MFE | -40.22 |
| Energy contribution | -40.94 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.63 |
| Structure conservation index | 0.74 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.14 |
| SVM RNA-class probability | 0.983504 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 18278957 118 - 21146708 GCAUUGUCUGGCAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC-GUGAUGUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCC- ((........))..(((((((((.....)))))))))(((((.(((((.((.(((.(.(((((((.(((((.-(((....))))))))))))))).).)))...)).))))).))))).- ( -58.50, z-score = -3.61, R) >droGri2.scaffold_15245 6816447 118 - 18325388 GCAUUGUCGGGCAGAUAGGUGCGGCACUCGAGCUUGUGAUUGUCGCCCUGCUUGAACGUCUGCGAACGAUCC-GUAACAUCGCGAUCGUCGCAGAGGAUCGAUAGCCGGGCGCCAACAC- .....(((.(((...((((.((((((.(((......))).)))))))))).((((.(.(((((((.((((((-(......)).)))))))))))).).))))..))).)))........- ( -50.90, z-score = -2.37, R) >droMoj3.scaffold_6496 13012580 118 - 26866924 GUCUUGGUGGGCAGGAACGUUUGGCAGACGAGCCUGUGGUGGUCGCCUUGCUUGAACGUCUGCGAUCGAUCC-GUGACAUCGCGACCGUCGCAGAGGAUCGACAGCUUGGCGCCCACAC- ..........(((((..((((.....))))..))))).((((.((((..((((((.(.(((((((.((...(-(((....))))..))))))))).).)))..)))..)))).))))..- ( -51.30, z-score = -1.58, R) >droVir3.scaffold_12875 19316171 118 - 20611582 GUCUUGGUGGGCAGGUACGUGCGGCACACGAGCUUGUGACGGUCGCCCUGCUUGAACGUCUGCGAGCGAUCC-GUUACAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACGC- ......((((((((((.((((.....)))).)))).........((((.((((((.(.(((((((.((((((-(......)).)))))))))))).).)))..))).))))))))))..- ( -56.70, z-score = -2.03, R) >droWil1.scaffold_180697 4079320 118 + 4168966 GUUGACGUUGCCAUAUAUGUUCGACAUUCUAACUUAUGGUGGUCACCCUGCUUGAACGUCUGCGAACGAUCC-GUUAUGUCGAGACCGUCGCAAAGGAUCGACAGCCGAGCGCCCACCA- ((((((((........))).)))))...........((((((.(.(.(.((((((.(.(.(((((.((.(((-(......)).)).))))))).).).)))..))).).).).))))))- ( -31.60, z-score = -0.03, R) >droPer1.super_4 4198042 118 - 7162766 GUCUCUGCCGAUAGGUACGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAGCGAUCC-GUAACAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCC- .....((((....)))).(((((.....)))))....(((((.(((((.((.(((.(.(((((((.((((((-(......)).)))))))))))).).)))...)).))))).))))).- ( -55.90, z-score = -3.39, R) >dp4.chr3 4925378 118 + 19779522 GUCUCUGCGGACAGGUACGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAGCGAUCC-GUAACAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCC- ((..(((....)))..))(((((.....)))))....(((((.(((((.((.(((.(.(((((((.((((((-(......)).)))))))))))).).)))...)).))))).))))).- ( -55.90, z-score = -2.77, R) >droAna3.scaffold_13266 12407705 118 + 19884421 GGCUCGCCGGGCAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCA-GUGAUGUCACGGUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCC- .(((.....)))..(((((((((.....)))))))))(((((.(((((.((.(((.(.(((((((.(((((.-(((....))))))))))))))).).)))...)).))))).))))).- ( -60.70, z-score = -3.31, R) >droEre2.scaffold_4845 12419327 118 - 22589142 GCAUUGCCGGGCAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC-GUGAUAUCGCGAUCAUCGCAGAGGAUCGACAGCCGGGCGCCCACCC- ...((((...))))(((((((((.....)))))))))(((((.(((((.((.(((.(.((((((((.((((.-(((....))))))))))))))).).)))...)).))))).))))).- ( -57.20, z-score = -3.48, R) >droYak2.chr2R 18028276 118 - 21139217 GCGUUGCCGGGUAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC-GUGAUAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCC- ..............(((((((((.....)))))))))(((((.(((((.((.(((.(.(((((((.(((((.-(((....))))))))))))))).).)))...)).))))).))))).- ( -57.60, z-score = -3.21, R) >droSec1.super_9 1587118 118 - 3197100 GCAUUGUCGGGCAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC-GUGAUGUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCC- ((........))..(((((((((.....)))))))))(((((.(((((.((.(((.(.(((((((.(((((.-(((....))))))))))))))).).)))...)).))))).))))).- ( -58.50, z-score = -3.25, R) >droSim1.chr2R 16877002 120 - 19596830 GCAUUGUCGGGCAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCCGGUGAUGUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCCC ((........))..(((((((((.....)))))))))(((((.(((((.((.(((.(.(((((((.((((((((.....))).)))))))))))).).)))...)).))))).))))).. ( -57.70, z-score = -2.55, R) >consensus GCAUUGGCGGGCAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC_GUGAUAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCCCACCC_ ..................(((((.....))))).....((((.(((((.((.(((.(.(((((((.(((((..((......)))))))))))))).).)))...)).))))).))))... (-40.22 = -40.94 + 0.72)


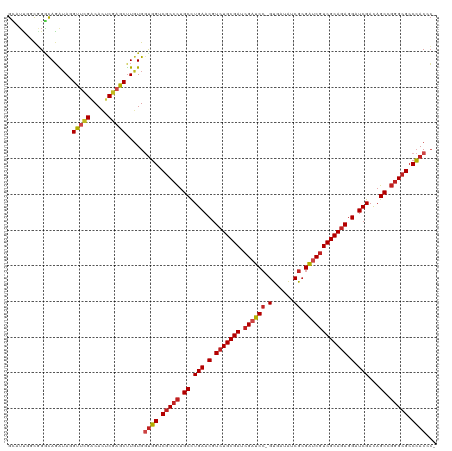
| Location | 18,278,962 – 18,279,081 |
|---|---|
| Length | 119 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 86.31 |
| Shannon entropy | 0.30558 |
| G+C content | 0.60531 |
| Mean single sequence MFE | -44.11 |
| Consensus MFE | -35.45 |
| Energy contribution | -34.98 |
| Covariance contribution | -0.47 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.40 |
| Structure conservation index | 0.80 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.58 |
| SVM RNA-class probability | 0.749588 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
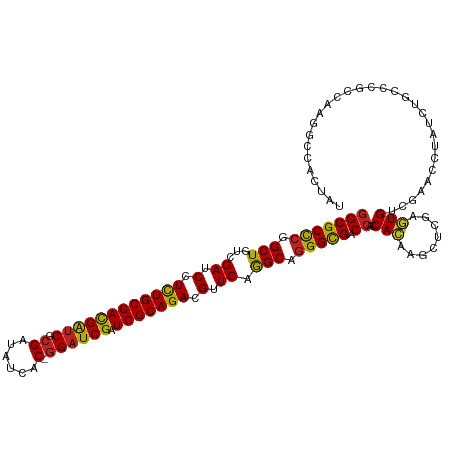
>dm3.chr2R 18278962 119 + 21146708 GGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGACAUCAC-GGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUGCCAGACAAUGCCACUAU .......(((((((((.(.((((((((((((.((......)-)))))).))))))).).)).((((((.((((.(((........).))))))......)))))).))))..)))..... ( -44.80, z-score = -1.88, R) >droGri2.scaffold_15245 6816452 119 + 18325388 GGCGCCCGGCUAUCGAUCCUCUGCGACGAUCGCGAUGUUAC-GGAUCGUUCGCAGACGUUCAAGCAGGGCGACAAUCACAAGCUCGAGUGCCGCACCUAUCUGCCCGACAAUGCAAAGAU (((((.(((((...(((..((((((((((((.((......)-)))))).)))))))(((((.....)))))...)))...))).)).)))))(((....((.....))...)))...... ( -40.70, z-score = -0.84, R) >droMoj3.scaffold_6496 13012585 119 + 26866924 GGCGCCAAGCUGUCGAUCCUCUGCGACGGUCGCGAUGUCAC-GGAUCGAUCGCAGACGUUCAAGCAAGGCGACCACCACAGGCUCGUCUGCCAAACGUUCCUGCCCACCAAGACGGACAU ((((((..(((...((.(.((((((((((((.((......)-)))))).))))))).).)).)))..)))).))....((((..(((.......)))..))))....((.....)).... ( -42.20, z-score = -1.07, R) >droVir3.scaffold_12875 19316176 119 + 20611582 GGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUGUAAC-GGAUCGCUCGCAGACGUUCAAGCAGGGCGACCGUCACAAGCUCGUGUGCCGCACGUACCUGCCCACCAAGACAGAUAU (((((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).))))).))(((....((.((((.....)))).....)).......)))...... ( -45.80, z-score = -1.32, R) >droWil1.scaffold_180697 4079325 119 - 4168966 GGCGCUCGGCUGUCGAUCCUUUGCGACGGUCUCGACAUAAC-GGAUCGUUCGCAGACGUUCAAGCAGGGUGACCACCAUAAGUUAGAAUGUCGAACAUAUAUGGCAACGUCAACCAAUAU ((((....(((((........((((((((((.((......)-)))))).)))))(((((((.(((..(((....)))....))).)))))))........)))))..))))......... ( -36.30, z-score = -1.31, R) >droPer1.super_4 4198047 119 + 7162766 GGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUGUUAC-GGAUCGCUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACGUACCUAUCGGCAGAGACCAGCAU (((((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).))))).)).......(((.(.((.((...((.......))...)).)))))).. ( -47.60, z-score = -1.34, R) >dp4.chr3 4925383 119 - 19779522 GGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUGUUAC-GGAUCGCUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACGUACCUGUCCGCAGAGACCAGCAU (((((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).))))).)).......(((((.....)))..((..(((....)))..))..)).. ( -46.30, z-score = -0.75, R) >droAna3.scaffold_13266 12407710 119 - 19884421 GGCGCCCGGCUGUCGAUCCUCUGCGACGACCGUGACAUCAC-UGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUGCCCGGCGAGCCCACAAU (((((((.(((...((.(.(((((((((((.(((....)))-.).))).))))))).).)).))).))))).)).......(((((.(.((.((.....)).)).)..)))))....... ( -45.80, z-score = -1.63, R) >droEre2.scaffold_4845 12419332 119 + 22589142 GGCGCCCGGCUGUCGAUCCUCUGCGAUGAUCGCGAUAUCAC-GGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUGCCCGGCAAUGCCACCAU ((((((.(((....((.(.((((((((((((.((......)-))))).)))))))).).))((..(((.((((.(((........).))))))..)))..))))).)))...)))..... ( -44.60, z-score = -1.07, R) >droYak2.chr2R 18028281 119 + 21139217 GGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUAUCAC-GGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUACCCGGCAACGCCACUAU ((((.(((((((((((.(.((((((((((((.((......)-)))))).))))))).).)).)))))((((((.(((........).))))))..)).......))))...))))..... ( -45.50, z-score = -1.99, R) >droSec1.super_9 1587123 119 + 3197100 GGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGACAUCAC-GGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUGCCCGACAAUGCCACUAU (((((((.(((...((.(.((((((((((((.((......)-)))))).))))))).).)).))).))))).)).......((.....(((((............)))))..))...... ( -44.90, z-score = -1.79, R) >droSim1.chr2R 16877008 120 + 19596830 GGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGACAUCACCGGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUGCCCGACAAUGCCACUAU (((((((.(((...((.(.((((((((((((.((.......))))))).))))))).).)).))).))))).)).......((.....(((((............)))))..))...... ( -44.80, z-score = -1.76, R) >consensus GGCGCCCGGCUGUCGAUCCUCUGCGACGAUCGCGAUAUCAC_GGAUCGAUCGCAGACGUUCAGGCAGGGCGACCACCACAAGCUCGAGUGUCGAACCUAUCUGCCCGCCAAGGCCACUAU (((((((.(((...((.(.((((((((((((............))))).))))))).).)).))).))))).))..(((........))).............................. (-35.45 = -34.98 + -0.47)
| Location | 18,278,962 – 18,279,081 |
|---|---|
| Length | 119 |
| Sequences | 12 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 86.31 |
| Shannon entropy | 0.30558 |
| G+C content | 0.60531 |
| Mean single sequence MFE | -51.32 |
| Consensus MFE | -35.73 |
| Energy contribution | -36.43 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.62 |
| Structure conservation index | 0.70 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.635302 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
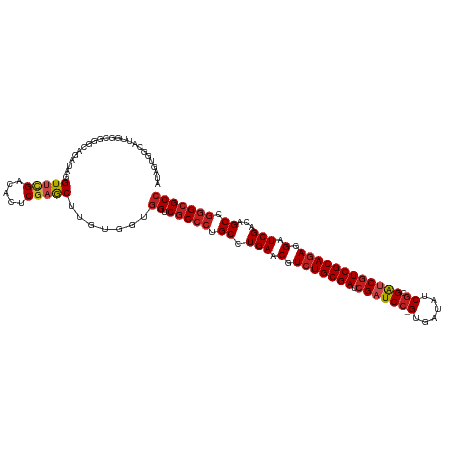
>dm3.chr2R 18278962 119 - 21146708 AUAGUGGCAUUGUCUGGCAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC-GUGAUGUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCC ....((((..((((.((((((((((((((.....)))))))))(((....)))))))).((.(.(((((((.(((((.-(((....))))))))))))))).).))))))))))...... ( -54.60, z-score = -2.39, R) >droGri2.scaffold_15245 6816452 119 - 18325388 AUCUUUGCAUUGUCGGGCAGAUAGGUGCGGCACUCGAGCUUGUGAUUGUCGCCCUGCUUGAACGUCUGCGAACGAUCC-GUAACAUCGCGAUCGUCGCAGAGGAUCGAUAGCCGGGCGCC ...........(((.(((...((((.((((((.(((......))).)))))))))).((((.(.(((((((.((((((-(......)).)))))))))))).).))))..))).)))... ( -50.90, z-score = -2.20, R) >droMoj3.scaffold_6496 13012585 119 - 26866924 AUGUCCGUCUUGGUGGGCAGGAACGUUUGGCAGACGAGCCUGUGGUGGUCGCCUUGCUUGAACGUCUGCGAUCGAUCC-GUGACAUCGCGACCGUCGCAGAGGAUCGACAGCUUGGCGCC ....(((......)))(((((..((((.....))))..)))))...((.((((..((((((.(.(((((((.((...(-(((....))))..))))))))).).)))..)))..)))))) ( -46.40, z-score = -0.05, R) >droVir3.scaffold_12875 19316176 119 - 20611582 AUAUCUGUCUUGGUGGGCAGGUACGUGCGGCACACGAGCUUGUGACGGUCGCCCUGCUUGAACGUCUGCGAGCGAUCC-GUUACAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCC .(((((((((....)))))))))(((.((((...(((((..(((.....)))...)))))..(.(((((((.((((((-(......)).)))))))))))).).......)))).))).. ( -53.90, z-score = -1.81, R) >droWil1.scaffold_180697 4079325 119 + 4168966 AUAUUGGUUGACGUUGCCAUAUAUGUUCGACAUUCUAACUUAUGGUGGUCACCCUGCUUGAACGUCUGCGAACGAUCC-GUUAUGUCGAGACCGUCGCAAAGGAUCGACAGCCGAGCGCC .....((.((((...((((((...(((.(.....).))).)))))).)))).)).(((((..((....))..((((((-.((.((.((....)).)).)).)))))).....)))))... ( -32.50, z-score = 0.01, R) >droPer1.super_4 4198047 119 - 7162766 AUGCUGGUCUCUGCCGAUAGGUACGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAGCGAUCC-GUAACAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCC ..((.(..(.(((((....)))..(((((.....)))))....)).)..)((((.((.(((.(.(((((((.((((((-(......)).)))))))))))).).)))...)).)))))). ( -48.60, z-score = -0.72, R) >dp4.chr3 4925383 119 + 19779522 AUGCUGGUCUCUGCGGACAGGUACGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAGCGAUCC-GUAACAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCC .((((.((((....)))).)))).(((((.....))))).......((.(((((.((.(((.(.(((((((.((((((-(......)).)))))))))))).).)))...)).))))))) ( -50.00, z-score = -0.66, R) >droAna3.scaffold_13266 12407710 119 + 19884421 AUUGUGGGCUCGCCGGGCAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCA-GUGAUGUCACGGUCGUCGCAGAGGAUCGACAGCCGGGCGCC .....(((((((.((((((((((((((((.....)))))))))(((....))))))))))..(.(((((((.(((((.-(((....))))))))))))))).).........))))).)) ( -56.10, z-score = -2.02, R) >droEre2.scaffold_4845 12419332 119 - 22589142 AUGGUGGCAUUGCCGGGCAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC-GUGAUAUCGCGAUCAUCGCAGAGGAUCGACAGCCGGGCGCC ..((((.(...((((((((((((((((((.....)))))))))(((....))))))))))..(.((((((((.((((.-(((....))))))))))))))).).......))..).)))) ( -54.50, z-score = -2.10, R) >droYak2.chr2R 18028281 119 - 21139217 AUAGUGGCGUUGCCGGGUAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC-GUGAUAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCC .....(((((..(((((((((((((((((.....)))))))))(((....))))))))(((.(.(((((((.(((((.-(((....))))))))))))))).).))).....)))))))) ( -54.80, z-score = -2.30, R) >droSec1.super_9 1587123 119 - 3197100 AUAGUGGCAUUGUCGGGCAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC-GUGAUGUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCC ....((((..(((((((((((((((((((.....)))))))))(((....)))))))))((.(.(((((((.(((((.-(((....))))))))))))))).).))))))))))...... ( -56.50, z-score = -2.64, R) >droSim1.chr2R 16877008 120 - 19596830 AUAGUGGCAUUGUCGGGCAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCCGGUGAUGUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCC .....(((....(((((((((((((((((.....)))))))))(((....)))))))))))..((((((..(((((((.((((((.......))))))...)))))))..).)))))))) ( -57.00, z-score = -2.56, R) >consensus AUAGUGGCAUUGGCGGGCAGAUAGGUUCGACACUCGAGCUUGUGGUGGUCGCCCUGCCUGAACGUCUGCGAUCGAUCC_GUGAUAUCGCGAUCGUCGCAGAGGAUCGACAGCCGGGCGCC ........................(((((.....))))).......((.(((((.((.(((.(.(((((((.(((((..((......)))))))))))))).).)))...)).))))))) (-35.73 = -36.43 + 0.70)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:43:12 2011