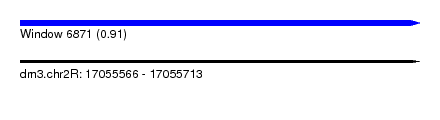
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 17,055,566 – 17,055,713 |
| Length | 147 |
| Max. P | 0.914741 |

| Location | 17,055,566 – 17,055,713 |
|---|---|
| Length | 147 |
| Sequences | 14 |
| Columns | 161 |
| Reading direction | forward |
| Mean pairwise identity | 75.47 |
| Shannon entropy | 0.53072 |
| G+C content | 0.49854 |
| Mean single sequence MFE | -50.08 |
| Consensus MFE | -21.97 |
| Energy contribution | -21.16 |
| Covariance contribution | -0.81 |
| Combinations/Pair | 1.48 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.44 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.24 |
| SVM RNA-class probability | 0.914741 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 17055566 147 + 21146708 AUGAACUUCUCCACACUCUGGACGACGUGCUUAUACAGCUUGAUGGCCCGCAUCGCCGACUUGGUGAUGGCGGCCAUCUUCGCUUUGGAAAUGGGCGGUCGGCUGUCAUAGAGUCCGGAGAGCUAAGGAGAAAUAUAG--AUGUUACAG------------ ......((((((.(.((((((((...(((.....((((((.((((((((.(((((((.....)))))))).))))))).((((((.......))))))..)))))))))...)))))))).)....))))))......--.........------------ ( -57.50, z-score = -3.18, R) >droSim1.chr2R 15704396 145 + 19596830 AUGAACUUCUCCACACUCUGGACGACGUGCUUAUACAGCUUGAUGGCCCGCAUCGCCGACUUGGUGAUGGCGGCCAUCUUCGCCUUGGAAAUGGGCGGUCGGCUGUCAUAGAGUCCGGAGAGCUAAGGA--AACAUAG--AUGUUAAAG------------ ...(((.(((.....((((((((...(((.....((((((.((((((((.(((((((.....)))))))).))))))).((((((.......))))))..)))))))))...))))))))......(..--..)..))--).)))....------------ ( -58.10, z-score = -3.48, R) >droSec1.super_9 381935 145 + 3197100 AUGAACUUCUCCACACUCUGGACGACGUGCUUAUACAGCUUGAUGGCCCGCAUCGCCGACUUGGUGAUGGCGGCCAUCUUCGCCUUGGAAAUGGGCGGUCGGCUGUCAUAGAGUCCGGAGAGCUAAGGA--AACAUAG--AUGUUAAAG------------ ...(((.(((.....((((((((...(((.....((((((.((((((((.(((((((.....)))))))).))))))).((((((.......))))))..)))))))))...))))))))......(..--..)..))--).)))....------------ ( -58.10, z-score = -3.48, R) >droYak2.chr2R 8998204 143 - 21139217 AUGAACUUCUCCACACUCUGGACGACGUGCUUGUACAGCUUAAUGGCCCGCAUCGCCGACUUGGUGAUGGCGGCCAUCUUCGCCUUGGAAAUGGGCGGUCGGCUGUCAUAGAGUCCGGAGAGCUACGAG----AAAGG--AUGAUUUAG------------ ......(((((..(.((((((((...(((.....((((((..(((((((.(((((((.....)))))))).))))))..((((((.......))))))..)))))))))...)))))))).)....)))----))...--.........------------ ( -53.60, z-score = -1.96, R) >droEre2.scaffold_4845 11197937 143 + 22589142 AUGAACUUCUCCACACUCUGGACGACGUGCUUGUACAGCUUGAUGGCCCGCAUCGCCGACUUGGUGAUGGCGGCCAUCUUCGCCUUGGAAAUGGGCGGUCGGCUGUCAUAGAGUCCGGAGAGCUAAGAG----AAAAG--AUGAUUUAG------------ ......(((((..(.((((((((...(((.....((((((.((((((((.(((((((.....)))))))).))))))).((((((.......))))))..)))))))))...)))))))).)....)))----))...--.........------------ ( -57.30, z-score = -3.08, R) >droAna3.scaffold_13266 12975012 148 - 19884421 AUGAACUUCUCCACGCUCUGCACGACAUGCUUGUAGAACUUGAUGGCGCGCAUCGCCGACUUGGUGAUUGCAGCCAUCUUGGCCUUGGAUAUGGGCGGUCGGCUGUCAUAGAGUCCGGAGAGCUGCAAAGGAAAAUAGC-AUGUAACAU------------ .......(((((..((((((...((((.(((.......(..((((((..((((((((.....))))).))).))))))..)((((.......))))....))))))).))))))..)))))((((..........))))-.........------------ ( -50.00, z-score = -0.67, R) >dp4.chr3 17692620 150 + 19779522 AUGAACUUCUCCACGCUCUGGACGACAUGCUUAUAGAACUUGAUGGCUCGCAUUGCCGACUUCGUAAUUGCGGCCAUUUUGGCCUUGGAUAUGGGCGGUCGGCUGUCAUAGAGUCCGGAGAGCUGCAAAGGACAGAAUGGGAAGAACAAG----------- .....((((.(((..((((((((..((((((...((..(..((((((.((((((((.(....))))).))))))))))..)..)).)).))))(((((....))))).....))))))))..((((....).)))..)))))))......----------- ( -50.90, z-score = -1.62, R) >droPer1.super_31 837642 150 - 935084 AUGAACUUCUCCACGCUCUGGACGACAUGCUUAUAGAACUUGAUGGCUCGCAUUGCCGACUUCGUAAUUGCGGCCAUUUUGGCCUUGGAUAUGGGCGGUCGGCUGUCAUAGAGUCCGGAGAGCUGCAAAGGACAGAAUGGGAAGAACAAG----------- .....((((.(((..((((((((..((((((...((..(..((((((.((((((((.(....))))).))))))))))..)..)).)).))))(((((....))))).....))))))))..((((....).)))..)))))))......----------- ( -50.90, z-score = -1.62, R) >droWil1.scaffold_180699 1855949 161 + 2593675 AUGAAUUUCUCCACACUCUGGACGACAUGUUUAUAAAAUUUGAUGGCACGCAUCGCCGAUUUGGUGAUGGCGGCCAUUUUGGCCUUGGAUAUGGGUGGACGGCUGUCAUAGAGCCCGGAAAGCUGUAAUCAAGAAAGAAAGAAAAAGAGAGCACGAUCAGG .(((.....(((((..((.....))((((((((........((((((.(((((((((.....)))))).))))))))).......)))))))).)))))(((((.((.((.(((.......))).)).((......))........)).))).)).))).. ( -45.46, z-score = -0.65, R) >droVir3.scaffold_12875 9707824 146 - 20611582 AUAAACUUUUCGACACUCUGCACAACAUGCUUGUAGAAUUUGAUUGCACGCAUUGCCGACUUUGUGAUGGCGGCCAUUUUAGCCUUUGAUAUGGGCGGCCGGCUAUCAUAGAGCCCGGAGAGCUGAAAAAUAA-AUACAUGUACAUG-------------- ......((((((...((((.......((((.(((((.......))))).))))..(((.((((((((((((((((......((((.......)))))))).))))))))))))..))))))).))))))....-.............-------------- ( -43.30, z-score = -2.19, R) >droMoj3.scaffold_6496 16033924 149 + 26866924 AUAAACUUUUCAACACUCUGCACAACAUGCUUGUAGAAUUUAAUUGCACGCAUUGCCGACUUUGUGAUGGCGGCCAUUUUAGCCUUCGAUAUGGGCGGUCGGCUAUCAUAGAGCCCGGAGAGCUGAAAGAAAACAUACACAUGUAGAAA------------ ......((((((...((((.......((((.(((((.......))))).))))..(((.((((((((((((((((......((((.......)))))))).))))))))))))..))))))).))))))...((((....)))).....------------ ( -41.60, z-score = -1.72, R) >droGri2.scaffold_15245 14318074 151 - 18325388 AUGAACUUUUCGACACUCUGCACAACAUGCUUAUAGAAUUUGAUUGCACGCAUUGCCGACUUUGUGAUGGCCGCCAUUUUAGCCUUCGAUAUGGGCGGCCGGCUAUCAUAGAGUCCGGAGAGCUGUGGAAACAAAUGGUGAUCAGUAAAAA---------- ...........((((((..(((.....))).......(((((.((.(((((.(..((((((((((((((((((((......((((.......)))))).))))))))))))))).)))..))).))).)).))))))))).))........---------- ( -49.70, z-score = -2.67, R) >anoGam1.chr2R 24759951 147 - 62725911 AUAAAUUUUUCCACACUCUGCACCACAUGCUUGUAGAACUUGAUCGCCUUCAUUGCACUGCGGGUAAUCGAGGCCAUUUUGGCCUUCGAAAUGGGCGGCUUCGACUCAUAGAGCCCGGACAGCUGUAACGGCAUACAUUUGGGGCAG-------------- .................((((.(((.(((..(((......(((......)))(((((((((((((..((((((((.....)))).)))).(((((((....)).)))))...)))))..))).)))))..)))..))).))).))))-------------- ( -45.70, z-score = -1.52, R) >triCas2.chrUn_66 155257 145 + 261922 AUGAAUUUUUCCACACUUUGCACUACAUGUUUGUAGAAUUUGAUAGCUUUGAUGGCCCCCCGCGUUAUCGCAGUCAUCUUGGCUUUGGAAAUCGGAGGCUUGACUUCGUACAGGGACGAAAGCUACAAGGG-AAAUGGAGCCGUUA--------------- ...(((..(((((..(((((..(((((....))))).......(((((((..((.(((...(((....)))(((((....((((((.(....).))))))))))).......))).)))))))))))))).-...)))))..))).--------------- ( -39.00, z-score = -0.20, R) >consensus AUGAACUUCUCCACACUCUGGACGACAUGCUUAUAGAACUUGAUGGCCCGCAUCGCCGACUUGGUGAUGGCGGCCAUCUUGGCCUUGGAAAUGGGCGGUCGGCUGUCAUAGAGUCCGGAGAGCUGAAAAGGAAAAUAG__AUGUAAAAG____________ ......(((((....(((((.....................(((.((.((((((((.......))))).))))).)))...((((.......))))............)))))....)))))....................................... (-21.97 = -21.16 + -0.81)

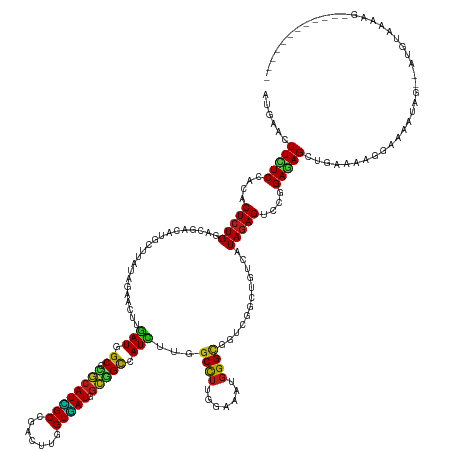
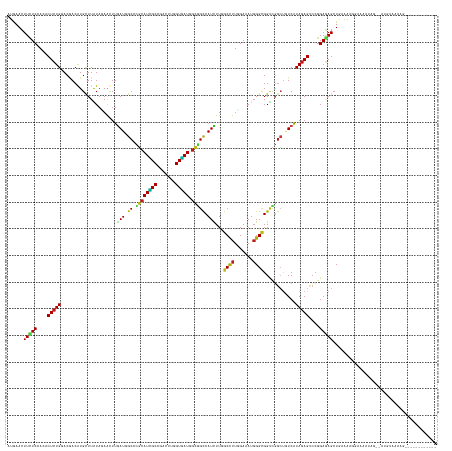
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:40:02 2011