
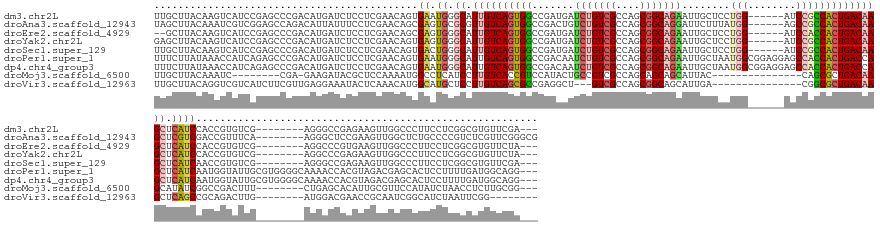
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 3,463,261 – 3,463,428 |
| Length | 167 |
| Max. P | 0.995343 |

| Location | 3,463,261 – 3,463,428 |
|---|---|
| Length | 167 |
| Sequences | 9 |
| Columns | 184 |
| Reading direction | forward |
| Mean pairwise identity | 67.07 |
| Shannon entropy | 0.66856 |
| G+C content | 0.55717 |
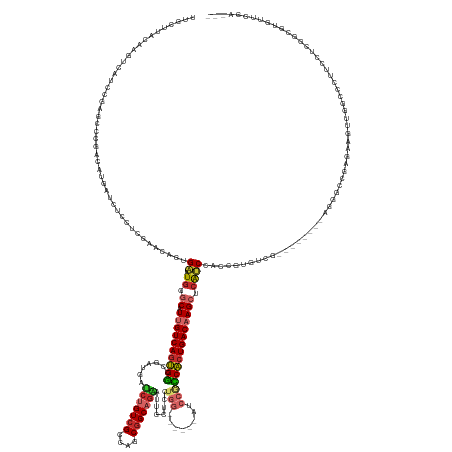
| Mean single sequence MFE | -63.48 |
| Consensus MFE | -24.67 |
| Energy contribution | -25.13 |
| Covariance contribution | 0.47 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.48 |
| Structure conservation index | 0.39 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.79 |
| SVM RNA-class probability | 0.995343 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 3463261 167 + 23011544 UUGCUUACAAGUCAUCCGAGCCCGACAUGAUCUCCUCGAACAGUGAAUGGGCAUUGUCAGUGGCCGAUGAUCUGUCGCCAGCGGCAGAAUUGCUCCUGG------AUCCGCCACUGACAAGCUCAUCCACCGUGUCG--------AGGGCCGAGAAGUUGGCCCUUCCUCGGCGUGUUCGA--- ................(((((.(..........((((((((.(((.((((((.(((((((((((((((.....))))((((.(((......))).))))------....))))))))))))))))).))).)).)))--------)))(((((((((......))).))))))).))))).--- ( -74.20, z-score = -4.10, R) >droAna3.scaffold_12943 2541833 170 - 5039921 UAGCUUACAAAUCGUCGGAGCCAGACAUUAUUUCCUCGAACAGCGAGUGCGCGUUGUCAGUGGCCGACUGUCUGUCGCCAGCGGCAGGAUUCUUUAUGG------AGCCGCCACUGACAAGCUCGUCGACCGUUUCA--------AGGGCUCCGAAGUUGGCUCUGCCCCGUCUCGUUCGGGCG .((((.((......((((((((.(((........((((.....)))).(.((.(((((((((((......(((((((....))))))).((((....))------))..))))))))))))).))))((.....)).--------..)))))))).)).))))..((((.(......).)))). ( -65.40, z-score = -1.39, R) >droEre2.scaffold_4929 3498101 165 + 26641161 --GCUUACAAGUCAUCCGAGCCCGACAUGAUCUCCUCGAACAGCGAGUGGGCAUUGUCAGUGGCCGAUGAUCUGUCGCCAGCGGCAGAAUUGCUCCUGG------AUCCACCACUGACAAGCUCAUCCACCGUGUCG--------AGGCCCGUGAAGUUGGCCCUUCCUCGGCGUGUUCUA--- --.......((.((((((((..(((((((.....((((.....))))(((((.((((((((((.((((.....))))((((.(((......))).))))------.....))))))))))))))).....)))))))--------.((((.........))))....))))).)))..)).--- ( -62.90, z-score = -1.99, R) >droYak2.chr2L 3450830 167 + 22324452 GAGCUUACAAGUCAUCCGAGCCCGACAUGAUCUCCUCGAACAGUGAGUGGGCAUUGUCAGUGGCCGAUGAUCUGUCGCCAGCGGCAGAAUUGCUCCUGG------AUCCGCCACUGACAAGCUCAUCCACCGUGUCG--------AGGCCCGAGAAGUUGGCCCUUCCUCGGCGUGUUCUA--- ((((...............((((((((((.((.....))...(((..(((((.(((((((((((((((.....))))((((.(((......))).))))------....))))))))))))))))..))))))))))--------.)))((((((((......))).)))))...))))..--- ( -70.30, z-score = -3.36, R) >droSec1.super_129 25358 167 + 51580 UUGCUUACAAGUCAUCCGAGCCCGACAUGAUCUCCUCGAACAGUGACUGGGCAUUGUCAGUGGCCGAUGAUCUGUCGCCAGCGGCAGAAUUGCUCCUGG------AUCCGCCACUGACAAGCUCAUCAACCGUGUCG--------AGGGCCGAGAAGUUGGCCCUUCCUCGGCGUGUUCGA--- ............((((((((..(((((((.((.....))....(((.(((((.(((((((((((((((.....))))((((.(((......))).))))------....)))))))))))))))))))..)))))))--------((((((((....))))))))..))))).))).....--- ( -70.40, z-score = -3.09, R) >droPer1.super_1 2415155 181 + 10282868 UUUCUUAUAAACCAUCAGAGCCCGACAUGAUCUCCUCGAACAGUGAAUGGGCAUUGUCAGUGGCCGACAAUCUGUCGCCAGCGGCAGAAUUGCUAAUGGCGGAGGAGCCACCACUGACCAGCUCAUCAAUGGUAUUGCGUGGGGCAAAACCACGUAGACGAGCACUCCUUUUGAUGGCAGG--- ...........(((((((((((((.(((..((.....))...)))..)))))...((((((((..(.((((((((((....)))))).)))))...((((......))))))))))))..((((((.....)).((((((((.......))))))))..))))......))))))))....--- ( -65.50, z-score = -2.23, R) >dp4.chr4_group3 936686 181 + 11692001 UUUCUUAUAAACCAUCAGAGCCCGACAUGAUCUCCUCGAACAGUGAAUGGGCAUUGUCAGUGGCCGACAAUCUGUCGCCAGCGGCAGAAUUGCUAAUGGCGGAGGAGCCACCACUGACCAGCUCAUCAAUGGUAUUGCGUGGGGCAAAACCACGUAGACGAGCACUCCUUUUGAUGGCAGG--- ...........(((((((((((((.(((..((.....))...)))..)))))...((((((((..(.((((((((((....)))))).)))))...((((......))))))))))))..((((((.....)).((((((((.......))))))))..))))......))))))))....--- ( -65.50, z-score = -2.23, R) >droMoj3.scaffold_6500 5396183 149 + 32352404 UUGCUUACAAAUC--------CGA-GAAGAUACGCUCCAAAAUGGCCUCAUCCUUGUCACCGUCCAUACUGCCGUCGCCAGCAGCAGCAUUAC---------------CAGCGCUGACAAGCAUAUCGGCCGACUUU--------CUGAGCACAUUGCGUUCCAUAUCUAACCUCUUGCGG--- ............(--------(((-(((((((.((((..((((((((..((.(((((((.(((...((.(((.((.(....).)).))).)).---------------..))).))))))).))...)))))..)))--------..))))((.....))....)))))....))))).).--- ( -33.90, z-score = -0.15, R) >droVir3.scaffold_12963 15952014 150 + 20206255 UUGCUUACAGGUCGUCAUCUUCGUUGAGGAAAUACUCAAACAUGGCAUGCUCCUUGUCAGCGCCGAGGCU---GUCGCCAGCGGCAGCAUUGA---------------CGGCGCUGACAAGCUCAGCCGCAGACUUG--------AUGGACGAACCGCAAUCGGCAUCUAAUUCGG-------- .((((....((((((((((.((.(((((......)))))....(((.((...(((((((((((((..(((---((((....))))))).....---------------)))))))))))))..)))))...))...)--------)).)))).)))......))))..........-------- ( -63.20, z-score = -3.74, R) >consensus UUGCUUACAAGUCAUCCGAGCCCGACAUGAUCUCCUCGAACAGUGAAUGGGCAUUGUCAGUGGCCGAUGAUCUGUCGCCAGCGGCAGAAUUGCUCCUGG______AUCCGCCACUGACAAGCUCAUCCACCGUGUCG________AGGGCCGAGAAGUUGGCCCUUCCUCGGCGUGUUCGA___ ............................................((.((.((.((((((((((.......(((((((....)))))))........(((........))))))))))))))).))))......................................................... (-24.67 = -25.13 + 0.47)
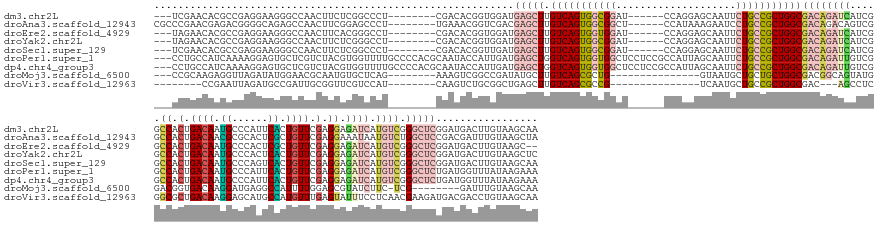
| Location | 3,463,261 – 3,463,428 |
|---|---|
| Length | 167 |
| Sequences | 9 |
| Columns | 184 |
| Reading direction | reverse |
| Mean pairwise identity | 67.07 |
| Shannon entropy | 0.66856 |
| G+C content | 0.55717 |
| Mean single sequence MFE | -66.97 |
| Consensus MFE | -25.77 |
| Energy contribution | -26.94 |
| Covariance contribution | 1.17 |
| Combinations/Pair | 1.36 |
| Mean z-score | -2.47 |
| Structure conservation index | 0.38 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.67 |
| SVM RNA-class probability | 0.994152 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 3463261 167 - 23011544 ---UCGAACACGCCGAGGAAGGGCCAACUUCUCGGCCCU--------CGACACGGUGGAUGAGCUUGUCAGUGGCGGAU------CCAGGAGCAAUUCUGCCGCUGGCGACAGAUCAUCGGCCACUGACAAUGCCCAUUCACUGUUCGAGGAGAUCAUGUCGGGCUCGGAUGACUUGUAAGCAA ---((((.((.((((((.(((......)))))))))(((--------(((.((((((((((.(((((((((((((.(((------.(((((....)))))...(((....)))...))).))))))))))).)).))))))))))))))))......))))))(((.(.(.....).).))).. ( -77.50, z-score = -4.23, R) >droAna3.scaffold_12943 2541833 170 + 5039921 CGCCCGAACGAGACGGGGCAGAGCCAACUUCGGAGCCCU--------UGAAACGGUCGACGAGCUUGUCAGUGGCGGCU------CCAUAAAGAAUCCUGCCGCUGGCGACAGACAGUCGGCCACUGACAACGCGCACUCGCUGUUCGAGGAAAUAAUGUCUGGCUCCGACGAUUUGUAAGCUA .((((..........))))..(((..((.((((((((((--------((((.(((.(((...((((((((((((((((.------..............))).....((((.....))))))))))))))).))....))))))))))))((.......)).))))))))......))..))). ( -65.76, z-score = -1.63, R) >droEre2.scaffold_4929 3498101 165 - 26641161 ---UAGAACACGCCGAGGAAGGGCCAACUUCACGGGCCU--------CGACACGGUGGAUGAGCUUGUCAGUGGUGGAU------CCAGGAGCAAUUCUGCCGCUGGCGACAGAUCAUCGGCCACUGACAAUGCCCACUCGCUGUUCGAGGAGAUCAUGUCGGGCUCGGAUGACUUGUAAGC-- ---.......(((((((((((......))))..((.(((--------(((.((((((..((.(((((((((((((.(((------.(((((....)))))...(((....)))...))).))))))))))).)).))..))))))))))))...))........))))).))..........-- ( -69.10, z-score = -2.34, R) >droYak2.chr2L 3450830 167 - 22324452 ---UAGAACACGCCGAGGAAGGGCCAACUUCUCGGGCCU--------CGACACGGUGGAUGAGCUUGUCAGUGGCGGAU------CCAGGAGCAAUUCUGCCGCUGGCGACAGAUCAUCGGCCACUGACAAUGCCCACUCACUGUUCGAGGAGAUCAUGUCGGGCUCGGAUGACUUGUAAGCUC ---..((.(...(((((.(((......)))))))).(((--------(((.((((((..((.(((((((((((((.(((------.(((((....)))))...(((....)))...))).))))))))))).)).))..)))))))))))).).)).....(((((.(.(.....).).))))) ( -71.30, z-score = -2.69, R) >droSec1.super_129 25358 167 - 51580 ---UCGAACACGCCGAGGAAGGGCCAACUUCUCGGCCCU--------CGACACGGUUGAUGAGCUUGUCAGUGGCGGAU------CCAGGAGCAAUUCUGCCGCUGGCGACAGAUCAUCGGCCACUGACAAUGCCCAGUCACUGUUCGAGGAGAUCAUGUCGGGCUCGGAUGACUUGUAAGCAA ---((((.((.((((((.(((......)))))))))(((--------(((.(((((.(((..(((((((((((((.(((------.(((((....)))))...(((....)))...))).))))))))))).))...))))))))))))))......))))))(((.(.(.....).).))).. ( -69.90, z-score = -2.36, R) >droPer1.super_1 2415155 181 - 10282868 ---CCUGCCAUCAAAAGGAGUGCUCGUCUACGUGGUUUUGCCCCACGCAAUACCAUUGAUGAGCUGGUCAGUGGUGGCUCCUCCGCCAUUAGCAAUUCUGCCGCUGGCGACAGAUUGUCGGCCACUGACAAUGCCCAUUCACUGUUCGAGGAGAUCAUGUCGGGCUCUGAUGGUUUAUAAGAAA ---...(((((((........(((((((...(((((.((((.....)))).))))).)))))))..((((((((((((......)))....(((((.(((.((....)).))))))))..)))))))))...((((...((.((.((.....)).))))..))))..))))))).......... ( -67.70, z-score = -2.33, R) >dp4.chr4_group3 936686 181 - 11692001 ---CCUGCCAUCAAAAGGAGUGCUCGUCUACGUGGUUUUGCCCCACGCAAUACCAUUGAUGAGCUGGUCAGUGGUGGCUCCUCCGCCAUUAGCAAUUCUGCCGCUGGCGACAGAUUGUCGGCCACUGACAAUGCCCAUUCACUGUUCGAGGAGAUCAUGUCGGGCUCUGAUGGUUUAUAAGAAA ---...(((((((........(((((((...(((((.((((.....)))).))))).)))))))..((((((((((((......)))....(((((.(((.((....)).))))))))..)))))))))...((((...((.((.((.....)).))))..))))..))))))).......... ( -67.70, z-score = -2.33, R) >droMoj3.scaffold_6500 5396183 149 - 32352404 ---CCGCAAGAGGUUAGAUAUGGAACGCAAUGUGCUCAG--------AAAGUCGGCCGAUAUGCUUGUCAGCGCUG---------------GUAAUGCUGCUGCUGGCGACGGCAGUAUGGACGGUGACAAGGAUGAGGCCAUUUUGGAGCGUAUCUUC-UCG--------GAUUUGUAAGCAA ---(((..(((((.((.((((........))))((((..--------(((((.((((..(((.(((((((.(((..---------------.....))((((((((....)))))))).....).))))))).))).))))))))).)))).)).))))-)))--------)............ ( -52.50, z-score = -1.29, R) >droVir3.scaffold_12963 15952014 150 - 20206255 --------CCGAAUUAGAUGCCGAUUGCGGUUCGUCCAU--------CAAGUCUGCGGCUGAGCUUGUCAGCGCCG---------------UCAAUGCUGCCGCUGGCGAC---AGCCUCGGCGCUGACAAGGAGCAUGCCAUGUUUGAGUAUUUCCUCAACGAAGAUGACGACCUGUAAGCAA --------..........(((...((((((.(((((.((--------(........(((((..(((((((((((((---------------.....((((.((....)).)---)))..)))))))))))))...)).))).((((.(((......)))))))..)))))))).))))))))). ( -61.30, z-score = -3.00, R) >consensus ___CCGAACACGACGAGGAAGGGCCAACUUCGCGGCCCU________CGACACGGUGGAUGAGCUUGUCAGUGGCGGAU______CCAGGAGCAAUUCUGCCGCUGGCGACAGAUCAUCGGCCACUGACAAUGCCCAUUCACUGUUCGAGGAGAUCAUGUCGGGCUCGGAUGACUUGUAAGCAA ............................................................(((((.(((((((((((....................)))))))))))((((((((.....((.(.((((.((......)).)))).).)).)))).)))).)))))................. (-25.77 = -26.94 + 1.17)
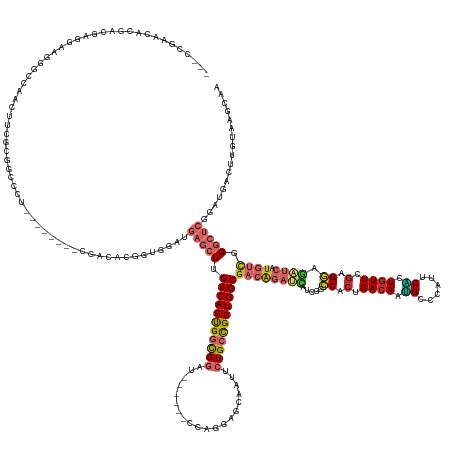
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:13:57 2011