| Sequence ID | dm3.chr2R |
|---|---|
| Location | 15,548,643 – 15,548,735 |
| Length | 92 |
| Max. P | 0.999758 |

| Location | 15,548,643 – 15,548,735 |
|---|---|
| Length | 92 |
| Sequences | 13 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 75.61 |
| Shannon entropy | 0.47853 |
| G+C content | 0.39834 |
| Mean single sequence MFE | -27.71 |
| Consensus MFE | -21.50 |
| Energy contribution | -21.59 |
| Covariance contribution | 0.10 |
| Combinations/Pair | 1.17 |
| Mean z-score | -3.28 |
| Structure conservation index | 0.78 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.93 |
| SVM RNA-class probability | 0.999484 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
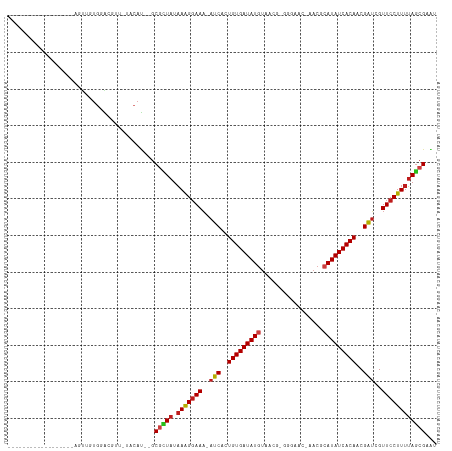
>dm3.chr2R 15548643 92 + 21146708 ------------------AGUUGUGGACUUUAAACAU--GCGUUAUAAAGGAAA-AUCACUGUGAUAUGUUA---GGAAAC-AACUCAUAUCACAACGAUCGUUCCUUUUAGCGAAU ------------------...................--.(((((.(((((((.-(((..(((((((((...---(....)-....)))))))))..)))..))))))))))))... ( -26.10, z-score = -3.78, R) >droSim1.chr2R 14254078 91 + 19596830 ------------------AAUUGUGGACUUUUAACAU--GCGAUAUAAAGGAAA-AUCACUGUGAUAUGUU----GGGAAC-AACUCAUAUCACAACGAUCGUUCCUUUUAGCGAAU ------------------..................(--((.....(((((((.-(((..(((((((((((----(....)-)))..))))))))..)))..)))))))..)))... ( -24.00, z-score = -3.24, R) >droSec1.super_1 13063372 92 + 14215200 ------------------AAUUGUGGACUUUAAACAU--GCGUUAUAAAGGAAA-AUCACUGUGAUAUGUUC---GGGAAC-AACUCAUAUCACAACGAUCGUUCCUUUUAGCGAAU ------------------...................--.(((((.(((((((.-(((..(((((((((...---(....)-....)))))))))..)))..))))))))))))... ( -24.60, z-score = -3.20, R) >droYak2.chr2R 7515070 92 - 21139217 ------------------AGUUGUGGACUUUGUACAU--GCGUUAUAAAGGAAA-AUCACUGUGAUAUGUUA---GGGAAU-AACGCAUAUCACAACGAUCGUUCCUUUUAGCGAAC ------------------...................--.(((((.(((((((.-(((..((((((((((..---......-...))))))))))..)))..))))))))))))... ( -24.30, z-score = -2.28, R) >droEre2.scaffold_4845 9748894 92 + 22589142 ------------------AGUUUUGGACUUUUUACAU--GCGUUAUAAAGGAAA-AUCACUGUGAUAUGUUA---GGGAAC-AACGCAUAUCACAACGAUCGUUCCUUUUAGCGAAC ------------------...................--.(((((.(((((((.-(((..((((((((((..---(....)-...))))))))))..)))..))))))))))))... ( -26.10, z-score = -3.76, R) >droAna3.scaffold_13266 10152652 94 - 19884421 ------------------CAUUUUGGACAAC-UGCAU--GCGUUAUAAAGGAAA-AUCACUGUGAUAUGUAACGAGGGAUA-AACGCAUAUCACAACGAUCGUUCCUUUUAGCGAAU ------------------.............-.....--.(((((.(((((((.-(((..((((((((((...........-...))))))))))..)))..))))))))))))... ( -24.24, z-score = -2.68, R) >dp4.chr3 12677221 94 + 19779522 ------------------GGCUGUGGGCGCA-GACAU--GCGCUAUAAAGGAAA-AUCACUGUGAUAUGUAACG-GUGAAUUAACGCAUAUCACAACGAUCGUUCCUUUUAGCGUAU ------------------..((((....)))-)..((--((((((.(((((((.-(((..((((((((((....-((......))))))))))))..)))..))))))))))))))) ( -34.00, z-score = -4.20, R) >droPer1.super_2 129862 94 - 9036312 ------------------GGCUGUGGGCGCA-GACAU--GCGCUAUAAAGGAAA-AUCACUGUGAUAUGUAACG-GUGAAUUAACGCAUAUCACAACGAUCGUUCCUUUUAGCGUAU ------------------..((((....)))-)..((--((((((.(((((((.-(((..((((((((((....-((......))))))))))))..)))..))))))))))))))) ( -34.00, z-score = -4.20, R) >droWil1.scaffold_180701 1651448 96 + 3904529 ------------------UAUUGGACAGAACAUGCAU--GCGCUAUAAAGGAAA-AUCACUGUGAUAUAUAACGGUGAAUCUAACGCAUAUCACAACGAUCGUUCCUUUUAGCGAAU ------------------...................--.(((((.(((((((.-(((..((((((((....((..........)).))))))))..)))..))))))))))))... ( -22.90, z-score = -2.30, R) >droVir3.scaffold_12875 16499029 91 - 20611582 --------------------CUUUACGGGCU-G-CAC--GCGCUAUAAAGGAAA-AUCACUGUGAUAUGUAACG-GUUAAUGAACGCAUAUCACAACGAUCGUUCCUUUUAGAGCAU --------------------........((.-.-...--))(((..(((((((.-(((..((((((((((....-(((....)))))))))))))..)))..)))))))...))).. ( -26.10, z-score = -3.02, R) >droMoj3.scaffold_6496 20936448 91 + 26866924 ---------------------UCUGGGGGAC-UGCAC--GCGCUAUAAAGGAAA-AUCACUGUGAUAUGUAACG-GUCGAUUAACGCAUAUCACAACGAUCGUUCCUUUUAGCGCAU ---------------------...((....)-)....--((((((.(((((((.-(((..((((((((((....-..........))))))))))..)))..))))))))))))).. ( -33.04, z-score = -4.71, R) >droGri2.scaffold_15112 2108515 92 + 5172618 --------------------UUUUGGGCAGA--GCAC--GCGCUAUAAAGGAAA-AUCACUGUGAUAUGUAACGAUUUAAUGAACGCAUAUCACAACGAUCGUUCCUUUUAGCGAGU --------------------...........--..((--.(((((.(((((((.-(((..((((((((((...............))))))))))..)))..)))))))))))).)) ( -27.36, z-score = -3.23, R) >anoGam1.chr3R 43698023 115 + 53272125 GUACAAACAUCUUGAGCACGCGUUGAAUGAGGAACAGCAGCAAUAUCAGGUAACUACCGCUGUGAUAUGGUACGGAUUUUCCAAC--AUAUCACACCGGGAGUUCCUUCGAUCGCCA ((.(((.....))).))..(((((((...((((((............((....)).(((.(((((((((....((.....))..)--)))))))).)))..)))))))))).))).. ( -33.50, z-score = -2.10, R) >consensus __________________AGUUGUGGACGUU_UACAU__GCGCUAUAAAGGAAA_AUCACUGUGAUAUGUAACG_GGGAAC_AACGCAUAUCACAACGAUCGUUCCUUUUAGCGAAU ........................................(((((.(((((((...((..(((((((((.................)))))))))..))...))))))))))))... (-21.50 = -21.59 + 0.10)


| Location | 15,548,643 – 15,548,735 |
|---|---|
| Length | 92 |
| Sequences | 13 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 75.61 |
| Shannon entropy | 0.47853 |
| G+C content | 0.39834 |
| Mean single sequence MFE | -27.63 |
| Consensus MFE | -18.94 |
| Energy contribution | -19.12 |
| Covariance contribution | 0.18 |
| Combinations/Pair | 1.14 |
| Mean z-score | -3.72 |
| Structure conservation index | 0.69 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 4.32 |
| SVM RNA-class probability | 0.999758 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
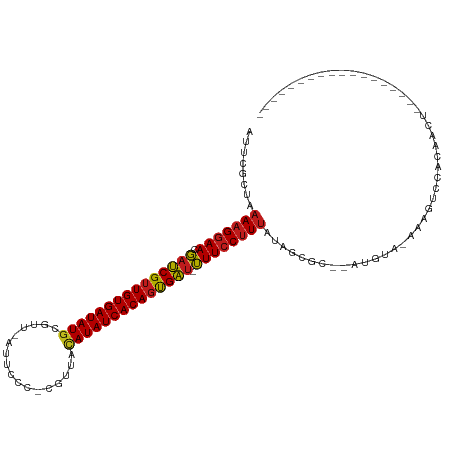
>dm3.chr2R 15548643 92 - 21146708 AUUCGCUAAAAGGAACGAUCGUUGUGAUAUGAGUU-GUUUCC---UAACAUAUCACAGUGAU-UUUCCUUUAUAACGC--AUGUUUAAAGUCCACAACU------------------ ....((..(((((((.(((((((((((((((....-......---...))))))))))))))-)))))))).....))--...................------------------ ( -22.52, z-score = -3.03, R) >droSim1.chr2R 14254078 91 - 19596830 AUUCGCUAAAAGGAACGAUCGUUGUGAUAUGAGUU-GUUCCC----AACAUAUCACAGUGAU-UUUCCUUUAUAUCGC--AUGUUAAAAGUCCACAAUU------------------ ....((..(((((((.((((((((((((((..(((-(....)----))))))))))))))))-)))))))).....))--...................------------------ ( -23.40, z-score = -3.60, R) >droSec1.super_1 13063372 92 - 14215200 AUUCGCUAAAAGGAACGAUCGUUGUGAUAUGAGUU-GUUCCC---GAACAUAUCACAGUGAU-UUUCCUUUAUAACGC--AUGUUUAAAGUCCACAAUU------------------ ....((..(((((((.(((((((((((((((..((-(....)---)).))))))))))))))-)))))))).....))--...................------------------ ( -23.90, z-score = -3.34, R) >droYak2.chr2R 7515070 92 + 21139217 GUUCGCUAAAAGGAACGAUCGUUGUGAUAUGCGUU-AUUCCC---UAACAUAUCACAGUGAU-UUUCCUUUAUAACGC--AUGUACAAAGUCCACAACU------------------ ....((..(((((((.(((((((((((((((....-......---...))))))))))))))-)))))))).....))--...................------------------ ( -22.52, z-score = -3.21, R) >droEre2.scaffold_4845 9748894 92 - 22589142 GUUCGCUAAAAGGAACGAUCGUUGUGAUAUGCGUU-GUUCCC---UAACAUAUCACAGUGAU-UUUCCUUUAUAACGC--AUGUAAAAAGUCCAAAACU------------------ ....((..(((((((.(((((((((((((((....-......---...))))))))))))))-)))))))).....))--...................------------------ ( -22.52, z-score = -3.28, R) >droAna3.scaffold_13266 10152652 94 + 19884421 AUUCGCUAAAAGGAACGAUCGUUGUGAUAUGCGUU-UAUCCCUCGUUACAUAUCACAGUGAU-UUUCCUUUAUAACGC--AUGCA-GUUGUCCAAAAUG------------------ ........(((((((.(((((((((((((((((..-.......))...))))))))))))))-))))))))(((((..--.....-)))))........------------------ ( -23.60, z-score = -2.91, R) >dp4.chr3 12677221 94 - 19779522 AUACGCUAAAAGGAACGAUCGUUGUGAUAUGCGUUAAUUCAC-CGUUACAUAUCACAGUGAU-UUUCCUUUAUAGCGC--AUGUC-UGCGCCCACAGCC------------------ ....(((.(((((((.(((((((((((((((...((((....-.))))))))))))))))))-))))))))...((((--(....-)))))....))).------------------ ( -31.60, z-score = -4.65, R) >droPer1.super_2 129862 94 + 9036312 AUACGCUAAAAGGAACGAUCGUUGUGAUAUGCGUUAAUUCAC-CGUUACAUAUCACAGUGAU-UUUCCUUUAUAGCGC--AUGUC-UGCGCCCACAGCC------------------ ....(((.(((((((.(((((((((((((((...((((....-.))))))))))))))))))-))))))))...((((--(....-)))))....))).------------------ ( -31.60, z-score = -4.65, R) >droWil1.scaffold_180701 1651448 96 - 3904529 AUUCGCUAAAAGGAACGAUCGUUGUGAUAUGCGUUAGAUUCACCGUUAUAUAUCACAGUGAU-UUUCCUUUAUAGCGC--AUGCAUGUUCUGUCCAAUA------------------ ...((((((((((((.(((((((((((((((((..........)))...)))))))))))))-)))))))).))))).--...................------------------ ( -27.10, z-score = -3.11, R) >droVir3.scaffold_12875 16499029 91 + 20611582 AUGCUCUAAAAGGAACGAUCGUUGUGAUAUGCGUUCAUUAAC-CGUUACAUAUCACAGUGAU-UUUCCUUUAUAGCGC--GUG-C-AGCCCGUAAAG-------------------- ((((.((((((((((.(((((((((((((((...........-.....))))))))))))))-)))))))).))).))--)).-.-...........-------------------- ( -28.19, z-score = -3.80, R) >droMoj3.scaffold_6496 20936448 91 - 26866924 AUGCGCUAAAAGGAACGAUCGUUGUGAUAUGCGUUAAUCGAC-CGUUACAUAUCACAGUGAU-UUUCCUUUAUAGCGC--GUGCA-GUCCCCCAGA--------------------- (((((((((((((((.(((((((((((((((...((((....-.))))))))))))))))))-)))))))).))))))--))...-..........--------------------- ( -34.30, z-score = -5.48, R) >droGri2.scaffold_15112 2108515 92 - 5172618 ACUCGCUAAAAGGAACGAUCGUUGUGAUAUGCGUUCAUUAAAUCGUUACAUAUCACAGUGAU-UUUCCUUUAUAGCGC--GUGC--UCUGCCCAAAA-------------------- ((.((((((((((((.(((((((((((((((((..........))...))))))))))))))-)))))))).))))).--))..--...........-------------------- ( -30.10, z-score = -4.69, R) >anoGam1.chr3R 43698023 115 - 53272125 UGGCGAUCGAAGGAACUCCCGGUGUGAUAU--GUUGGAAAAUCCGUACCAUAUCACAGCGGUAGUUACCUGAUAUUGCUGCUGUUCCUCAUUCAACGCGUGCUCAAGAUGUUUGUAC .((((((...(((((((.(((.((((((((--(.(((.....)))...))))))))).))).)))).)))...))))))((.(((........)))))((((...........)))) ( -37.80, z-score = -2.66, R) >consensus AUUCGCUAAAAGGAACGAUCGUUGUGAUAUGCGUU_AUUCCC_CGUUACAUAUCACAGUGAU_UUUCCUUUAUAGCGC__AUGUA_AAAGUCCACAACU__________________ ........(((((((..((((((((((((((.................))))))))))))))..))))))).............................................. (-18.94 = -19.12 + 0.18)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:35:31 2011