
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 14,735,814 – 14,735,934 |
| Length | 120 |
| Max. P | 0.649218 |

| Location | 14,735,814 – 14,735,934 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.65 |
| Shannon entropy | 0.49188 |
| G+C content | 0.55000 |
| Mean single sequence MFE | -45.95 |
| Consensus MFE | -16.75 |
| Energy contribution | -16.46 |
| Covariance contribution | -0.29 |
| Combinations/Pair | 1.59 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.36 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.33 |
| SVM RNA-class probability | 0.649218 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
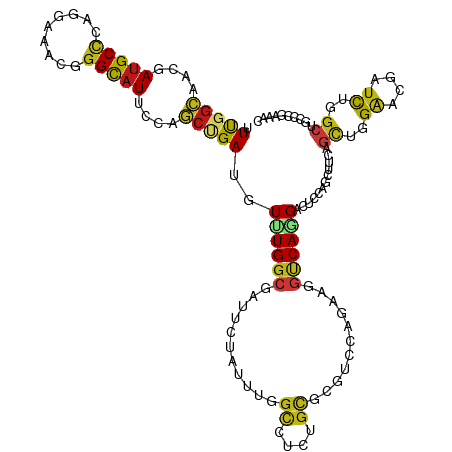
>dm3.chr2R 14735814 120 - 21146708 UUCGGCAACGAUGCCCAGGAAACUGGCAUUCCAGCUGAUGUUUGGCGAUUCUAUUUGGCAUCUGCGCGACCAGAAGGUCAGGAUUCUAGCUUCAGCUGGAAUGAUCUGGCUGCCCGAAAU (((((((.((((..((((....))))((((((((((((.(((.((.((((((.....((....))..((((....))))))))))))))).))))))))))))))).)..)).))))).. ( -50.30, z-score = -3.52, R) >droSim1.chr2R 13435696 120 - 19596830 UUCGGCAACGAUGCCCAGGAAACUGGCAUUCCAGCUGAUGUUUGGCGAUUCUACUUGGCCUCUGCGCGUCCUGAAGGUCAGGACUCCAGCUUCAGCUGGAAUGAUCUGGCUGCCCGAAAU (((((((.((((..((((....))))((((((((((((.....(((.(.......).))).(((.(.((((((.....)))))).))))..))))))))))))))).)..)).))))).. ( -53.60, z-score = -4.09, R) >droSec1.super_1 12244103 120 - 14215200 UUCGGCAACGAUGCCCAGGAAACUGGCAUUCCAGCUGAUGUUUGGCGAUUCUACUUGGCCUCUGCGCGUCCUGAAGGUCAGGACUCCAGCUUCAGCUGGAAUGAUCUCGCUGUCCGAAAU (((((((.(((...((((....))))((((((((((((.....(((.(.......).))).(((.(.((((((.....)))))).))))..))))))))))))...))).)).))))).. ( -56.20, z-score = -5.21, R) >droYak2.chr2R 6679353 120 + 21139217 UUUGGCAACGAUGCCCAGGAAACGGGCAUUCCAGCUGAUGUGUGGCGAUUCUACUUGGCCUCUGCGCGACCUGAAGGCCAGGACUCCAGCUUCAGCUGGAAUGAUCUGGCUGCCCGAAAC ...((((.((((((((.(....)))))(((((((((((.((.(((.(......(((((((((..........).)))))))).).))))).))))))))))).))).)..))))...... ( -48.70, z-score = -2.60, R) >droEre2.scaffold_4845 8928001 120 - 22589142 UUUGGCAACGAUGCCCAGGAAACUGGCAUUCCAGCUGAUGUGUGGCGUUUCUACUUGGCCUCUGCGCGGCCUGAAGGUCAGGACUCCAGCUUCAGCUGGAAUGAUUUGGCUGCCCGAAAC ...((((.(((...((((....))))((((((((((((.((.(((.((((..((((((((.......))))...))))..)))).))))).))))))))))))..)))..))))...... ( -52.20, z-score = -3.37, R) >droAna3.scaffold_13266 2273065 120 + 19884421 UUCGGCAACGACGCUCAAGAAACAGGCAUUCCGGCUGACGUCUGGCGUUUCUAUCUUGCCUCCGCCCGUCCGGAAGGUCAGGACUCUAGCUUCAGUUGGAACGAUCUGGCUGCCCGUAAC ..((....))(((....((...(((((.((((((((((.(.((((.(((.....((.(((((((......))).)))).))))).))))).)))))))))).).)))).))...)))... ( -38.90, z-score = -0.22, R) >dp4.chr3 2531879 120 - 19779522 UUUGGCAAUGAUGCCCAGGCAACGGGCAUUCCGGCAGAUGUCUGGCGCUUCUAUUUGGCCUCGGCACGACCAGAGGGCCAGGACUCGAGCUUCAGUUGGAACGAUUUGGCGGCACGCAAC ....((...(((((((.(....))))))))(((.(((((.((((((.(((((..(((((....)).)))..)))))))))))..((.(((....))).))...))))).)))...))... ( -47.10, z-score = -1.69, R) >droPer1.super_2 2722706 120 - 9036312 UUUGGCAAUGAUGCCCAGGCAACGGGCAUUCCGGCAGAUGUCUGGCGCUUCUAUUUGGCCUCGGCACGACCAGAGGGCCAGGACUCGAGCUUCAGUUGGAACGAUCUGGCGGCACGCAAC ....((...(((((((.(....))))))))(((.(((((.((((((.(((((..(((((....)).)))..)))))))))))..((.(((....))).))...))))).)))...))... ( -49.70, z-score = -2.17, R) >droWil1.scaffold_180697 2264629 120 - 4168966 UUUGGCAAUGAUGCCCAAGAGACUGGCAUACCAGCAGAUGUCUGGCGUUUUUAUCUCGCCUCGGCUCGUCCAGAGGGUCAGGAUUCGAGUUUCAGUUGGAACGAUUUGGCAGCGCGCAAC ...((((....)))).......((((....))))(((....)))(((((.(((..((.(((.(((((.......))))))))......(((((....)))))))..))).)))))..... ( -32.10, z-score = 1.47, R) >droVir3.scaffold_12875 831382 120 + 20611582 UUUGGCAAUGAUGCCCAGGAGACGGGCAUACCCGCUGACGUUUGGCGUUUCUAUUUGGCAUCGGCACGUCCCGAGGGUCAGGAUUCCAGCUUCAGCUGGAACGAUUUGGCCGCACGCAAC .(..((....((((((.(....)))))))....))..)(((.((((.......((((((.((((......))))..)))))).(((((((....))))))).......)))).))).... ( -46.40, z-score = -1.83, R) >droMoj3.scaffold_6496 19994984 120 + 26866924 UUUGGCAACGAUGCCCAGGAGACGGGCAUACCCGCCGACGUCUGGCGUUUCUAUCUGGCCUCCGCCCGUCCCGAGGGCCAGGACUCUAGCUUCAGCUGGACUGAUUUGGCCGCACGCAAC .(((((....((((((.(....)))))))....)))))(((.((((.......(((((((..((.......))..)))))))..((((((....))))))........)))).))).... ( -50.60, z-score = -2.09, R) >droGri2.scaffold_15245 17792260 120 - 18325388 UUUGGCAACGAUGCCCAAGAGACGGGCAUUCCCGCUGAUGUUUGGCGCUUCUAUUUGGCGUCAGCACGGCCUGAAGGCCAGGACUCUAGCUUUAGCUGGAACGAUCUGGCUGCACGCAAC .(..((...(((((((.......)))))))...))..)(((((((((((.......)))))))).)))((.((..(((((((.(((((((....))))))..).))))))).)).))... ( -46.60, z-score = -1.97, R) >anoGam1.chr3R 50739573 120 + 53272125 UUUGGCAACGAUGCGCAGGACACGGGCAUUCCGGCCGACGUGUGGCGCUUCUAUCUCGGUGCAGCGCGGCCCGAGGGCCAGGAUUCAUCGUUCAGCUGGAGCGAUCUGCAGGCGCGCAAC .(((....)))(((((.(((..(.(((.(((.(((((.(((...(((((........))))).)))))))).))).))).)..)))(((((((.....)))))))........))))).. ( -50.90, z-score = -0.50, R) >apiMel3.Group6 193035 120 - 14581788 UUUGGAACUGAUGCUAGAGAUACAGGUAUACCAUCUGAUGUAUGGCGAUUUUAUCUAGCAUAUGUUAGACCCGAAACUCAAGAUUCAAAUUUUAAUUGGGUAGAUUUAGCAACUAAAAAU (((((.....((((((((......(.(((((........))))).).......)))))))).((((((((((((.....(((((....)))))..))))))....)))))).)))))... ( -25.42, z-score = -1.29, R) >triCas2.ChLG7 11503140 120 + 17478683 UUUGGAAACGAUGCCCGAGAUACGGGCAUUCCGAGCGACGUUUGGCGAUUUUAUUUAUUGUAUGUGAGGCCAGAAUCACAAGAUUCCAGCUUUAGUUGGUCGGAUUUGGCCACGAAAAAU .(((....)))((((((.....)))))).(((((.((((.....(((((.......)))))...((((((..(((((....)))))..)))))))))).)))))((((....)))).... ( -40.50, z-score = -3.22, R) >consensus UUUGGCAACGAUGCCCAGGAAACGGGCAUUCCAGCUGAUGUUUGGCGAUUCUAUUUGGCCUCUGCGCGUCCAGAAGGUCAGGACUCCAGCUUCAGCUGGAACGAUCUGGCUGCCCGAAAC .(((((....(((((.........)))))....)))))..((((((...........((....))...........))))))............((..((....))..)).......... (-16.75 = -16.46 + -0.29)


Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:32:53 2011