
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 14,520,993 – 14,521,126 |
| Length | 133 |
| Max. P | 0.999313 |

| Location | 14,520,993 – 14,521,104 |
|---|---|
| Length | 111 |
| Sequences | 14 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 75.65 |
| Shannon entropy | 0.56623 |
| G+C content | 0.57007 |
| Mean single sequence MFE | -42.16 |
| Consensus MFE | -24.86 |
| Energy contribution | -25.61 |
| Covariance contribution | 0.75 |
| Combinations/Pair | 1.52 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.59 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.06 |
| SVM RNA-class probability | 0.997226 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
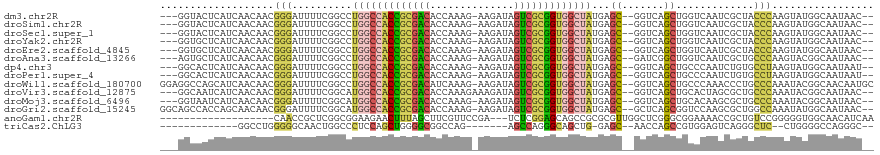
>dm3.chr2R 14520993 111 + 21146708 --GUUAUUGCCAUACUUGGGUAGCGAUUGACCAGCUGACC--GCUCAUAGCCACCGCGACUAUCUU-CUUUGGUGUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAGUACC--- --.(((((..((....((((((((.........)))).(.--(((....(((((((((((.(((..-....))))))))))))))..))).).....)))).))..))))).....--- ( -38.40, z-score = -1.99, R) >droSim1.chr2R 13225385 111 + 19596830 --GUUAUUGCCAUACUUGGGUAGCGAUUGACCAGCUGACC--GCUCAUAGCCACCGCGACUAUCUU-CUUUGGUGUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAGUACC--- --.(((((..((....((((((((.........)))).(.--(((....(((((((((((.(((..-....))))))))))))))..))).).....)))).))..))))).....--- ( -38.40, z-score = -1.99, R) >droSec1.super_1 12028711 111 + 14215200 --GUUAUUGCCAUACUUGGGUAGCGAUUGACCAGCUGACC--GCUCAUAGCCACCGCGACUAUCUU-CUUUGGUGUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAGUACC--- --.(((((..((....((((((((.........)))).(.--(((....(((((((((((.(((..-....))))))))))))))..))).).....)))).))..))))).....--- ( -38.40, z-score = -1.99, R) >droYak2.chr2R 6456488 111 - 21139217 --GUUAUUGCCAUACUUGGGUAGCGAUUGACCAGCUGACC--GCUCAUAGCCACCGCGACUAUCUU-CUUUGGUGUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAGCACC--- --...............((.((((.........)))).))--((((((.(((((((((((.(((..-....))))))))))))))..((.........)).......))))))...--- ( -40.70, z-score = -2.45, R) >droEre2.scaffold_4845 8708801 111 + 22589142 --GUUAUUGCCAUACUUGGGUAGCGAUUGACCAGCUGACC--GCUCAUAGCCACCGCGACUAUCUU-CUUUGGUGUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAGCACC--- --...............((.((((.........)))).))--((((((.(((((((((((.(((..-....))))))))))))))..((.........)).......))))))...--- ( -40.70, z-score = -2.45, R) >droAna3.scaffold_13266 6887933 111 + 19884421 --GUUAUUGCCGUACUUGGGCAGCGAUUGACCAGCCGAUC--GCUCAUAGCCACCGCGACUAUCUU-CUUUGGUGUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAGCACU--- --.....((((((...((((.((((((((......)))))--)))....(((((((((((.(((..-....))))))))))))))............))))......))).)))..--- ( -44.40, z-score = -2.90, R) >dp4.chr3 10581863 111 + 19779522 --AUUAUUGCCAUACUUAGGCACAGAUUGGGCAGCUGACC--GCUCAUAGCCACCGCGACUAUCUU-CUUUGGUGUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAGUGCC--- --................(((((.((((.((((((.....--)))....(((((((((((.(((..-....))))))))))))))...)))...)))).((((...)))).)))))--- ( -41.80, z-score = -2.31, R) >droPer1.super_4 5910374 111 + 7162766 --AUUAUUGCCAUACUUAGGCACAGAUUGGGCAGCUGACC--GCUCAUAGCCACCGCGACUAUCUU-CUUUGGUGUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAGUGCC--- --................(((((.((((.((((((.....--)))....(((((((((((.(((..-....))))))))))))))...)))...)))).((((...)))).)))))--- ( -41.80, z-score = -2.31, R) >droWil1.scaffold_180700 1809394 116 - 6630534 GCAUUGUUGCCGUAUUUGGGCAGGGUUUGGGCAGCUGACC--GCUCAUAGCCACCGCGACUAUCUU-CUUUGAUGUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGCUGGCCUCC ........((((((((..(((.((((((.((((((.....--)))....(((((((((((.(((..-....))))))))))))))...)))..)))))).)))...))))).))).... ( -49.50, z-score = -2.61, R) >droVir3.scaffold_12875 9466490 112 - 20611582 --GUUAUUGCCGUAUUUGGGCAGCGCUAGUGCAGCUGACC--GCUCAUAGCCACCGCGACUAUCUUUCUUUGGUGUCGCGGUGGCCAUGCCGAAAAUCCCGUUGUUGAUGAUUGCC--- --((((((..((....((((((((((....)).))))..(--(..(((.(((((((((((.(((.......)))))))))))))).))).)).....)))).))..))))))....--- ( -42.70, z-score = -2.65, R) >droMoj3.scaffold_6496 15781964 111 + 26866924 --GUUAUUGCCGUAUUUGGGCAGCGCUUGUGCAGCUGACC--GCUCAUAGCCACCGCGACUAUCUU-CUUUGGUGUCGCGGUGGCCAUGCCGAAAAUCCCGUUGUUGAUGAUUACC--- --((((((..((....((((((((((....)).))))..(--(..(((.(((((((((((.(((..-....)))))))))))))).))).)).....)))).))..))))))....--- ( -43.20, z-score = -3.24, R) >droGri2.scaffold_15245 13589624 114 + 18325388 --GUUAUUGCCAUAUUUGGCCAGCGCUUGGACCGCUGAGC--GCUCAUAGCCACCGCGACUAUCUU-CUUUGGUGUCGCGGUGGCCAUGCCGAAAAUCCCGUUGUUGCUGGUGGCUGCC --......((((..((((((.((((((..(....)..)))--)))....(((((((((((.(((..-....))))))))))))))...))))))...((.((....)).)))))).... ( -52.50, z-score = -4.18, R) >anoGam1.chr2R 24316465 97 + 62725911 UUGAUGUUGCCACCCCCGGACAGCGGUUUUCCGCCCGAGCCAACGCGCGGCUGCUCCGAGA---UCGGAACGAAGCUAAAGUUCUUCCGCCGAGCGGUUG------------------- .....((((((......)).))))(((((.......)))))..(((.((((.(.((((...---.)))).)((((........)))).)))).)))....------------------- ( -30.10, z-score = 0.61, R) >triCas2.ChLG3 5084278 92 + 32080666 --GCCCUGGCCCCAG--GAGCCCUGACUCCACGGCUGGUU--GCUC-CAGCUGCCCUGGCU-------CUGGCCGCCCCAGCUGGAGGGCCAGUUGCCCCCAGGCC------------- --..(((((.....(--(((......)))).(((((((((--.(((-((((((...((((.-------...))))...))))))))))))))))))...)))))..------------- ( -47.70, z-score = -0.93, R) >consensus __GUUAUUGCCAUACUUGGGCAGCGAUUGACCAGCUGACC__GCUCAUAGCCACCGCGACUAUCUU_CUUUGGUGUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAGUACC___ .......((((.......)))).........(((((((......)))..(((((((((((.(((.......))))))))))))))..................))))............ (-24.86 = -25.61 + 0.75)


| Location | 14,520,993 – 14,521,104 |
|---|---|
| Length | 111 |
| Sequences | 14 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 75.65 |
| Shannon entropy | 0.56623 |
| G+C content | 0.57007 |
| Mean single sequence MFE | -42.92 |
| Consensus MFE | -25.60 |
| Energy contribution | -25.65 |
| Covariance contribution | 0.05 |
| Combinations/Pair | 1.65 |
| Mean z-score | -2.54 |
| Structure conservation index | 0.60 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.54 |
| SVM RNA-class probability | 0.998889 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 14520993 111 - 21146708 ---GGUACUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACACCAAAG-AAGAUAGUCGCGGUGGCUAUGAGC--GGUCAGCUGGUCAAUCGCUACCCAAGUAUGGCAAUAAC-- ---(((.(((.........)))......(((((((((((((((((.......-......)))))))))))))..(((--.....))))))).)))((((........))))......-- ( -37.52, z-score = -1.88, R) >droSim1.chr2R 13225385 111 - 19596830 ---GGUACUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACACCAAAG-AAGAUAGUCGCGGUGGCUAUGAGC--GGUCAGCUGGUCAAUCGCUACCCAAGUAUGGCAAUAAC-- ---(((.(((.........)))......(((((((((((((((((.......-......)))))))))))))..(((--.....))))))).)))((((........))))......-- ( -37.52, z-score = -1.88, R) >droSec1.super_1 12028711 111 - 14215200 ---GGUACUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACACCAAAG-AAGAUAGUCGCGGUGGCUAUGAGC--GGUCAGCUGGUCAAUCGCUACCCAAGUAUGGCAAUAAC-- ---(((.(((.........)))......(((((((((((((((((.......-......)))))))))))))..(((--.....))))))).)))((((........))))......-- ( -37.52, z-score = -1.88, R) >droYak2.chr2R 6456488 111 + 21139217 ---GGUGCUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACACCAAAG-AAGAUAGUCGCGGUGGCUAUGAGC--GGUCAGCUGGUCAAUCGCUACCCAAGUAUGGCAAUAAC-- ---..(((((((......((((........))))(((((((((((.......-......))))))))))).))))))--)((((((((((........)))..))).))))......-- ( -41.32, z-score = -2.48, R) >droEre2.scaffold_4845 8708801 111 - 22589142 ---GGUGCUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACACCAAAG-AAGAUAGUCGCGGUGGCUAUGAGC--GGUCAGCUGGUCAAUCGCUACCCAAGUAUGGCAAUAAC-- ---..(((((((......((((........))))(((((((((((.......-......))))))))))).))))))--)((((((((((........)))..))).))))......-- ( -41.32, z-score = -2.48, R) >droAna3.scaffold_13266 6887933 111 - 19884421 ---AGUGCUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACACCAAAG-AAGAUAGUCGCGGUGGCUAUGAGC--GAUCGGCUGGUCAAUCGCUGCCCAAGUACGGCAAUAAC-- ---...((((((......((((........))))(((((((((((.......-......))))))))))).))))))--((((....))))....((((........))))......-- ( -42.52, z-score = -2.51, R) >dp4.chr3 10581863 111 - 19779522 ---GGCACUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACACCAAAG-AAGAUAGUCGCGGUGGCUAUGAGC--GGUCAGCUGCCCAAUCUGUGCCUAAGUAUGGCAAUAAU-- ---((((((.(((.....(((.....)))((((((((((((((((.......-......))))))))))))).).))--))).)).)))).......((((.......)))).....-- ( -42.02, z-score = -2.53, R) >droPer1.super_4 5910374 111 - 7162766 ---GGCACUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACACCAAAG-AAGAUAGUCGCGGUGGCUAUGAGC--GGUCAGCUGCCCAAUCUGUGCCUAAGUAUGGCAAUAAU-- ---((((((.(((.....(((.....)))((((((((((((((((.......-......))))))))))))).).))--))).)).)))).......((((.......)))).....-- ( -42.02, z-score = -2.53, R) >droWil1.scaffold_180700 1809394 116 + 6630534 GGAGGCCAGCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACAUCAAAG-AAGAUAGUCGCGGUGGCUAUGAGC--GGUCAGCUGCCCAAACCCUGCCCAAAUACGGCAACAAUGC (((((((((.(((........))).)).)))))(((((((((((((((....-..))).)))))))))))).((.((--((....)))).))...))((((.......))))....... ( -47.70, z-score = -3.37, R) >droVir3.scaffold_12875 9466490 112 + 20611582 ---GGCAAUCAUCAACAACGGGAUUUUCGGCAUGGCCACCGCGACACCAAAGAAAGAUAGUCGCGGUGGCUAUGAGC--GGUCAGCUGCACUAGCGCUGCCCAAAUACGGCAAUAAC-- ---.((((((.((......))))))..((.(((((((((((((((..............)))))))))))))))...--((.((((.((....)))))).)).....))))......-- ( -43.14, z-score = -3.62, R) >droMoj3.scaffold_6496 15781964 111 - 26866924 ---GGUAAUCAUCAACAACGGGAUUUUCGGCAUGGCCACCGCGACACCAAAG-AAGAUAGUCGCGGUGGCUAUGAGC--GGUCAGCUGCACAAGCGCUGCCCAAAUACGGCAAUAAC-- ---................(((....(((.(((((((((((((((.......-......)))))))))))))))..)--)).((((.((....)))))))))...............-- ( -42.82, z-score = -3.74, R) >droGri2.scaffold_15245 13589624 114 - 18325388 GGCAGCCACCAGCAACAACGGGAUUUUCGGCAUGGCCACCGCGACACCAAAG-AAGAUAGUCGCGGUGGCUAUGAGC--GCUCAGCGGUCCAAGCGCUGGCCAAAUAUGGCAAUAAC-- ....((((...((.....(((.....))).(((((((((((((((.......-......))))))))))))))).))--((.(((((.......)))))))......))))......-- ( -48.02, z-score = -3.61, R) >anoGam1.chr2R 24316465 97 - 62725911 -------------------CAACCGCUCGGCGGAAGAACUUUAGCUUCGUUCCGA---UCUCGGAGCAGCCGCGCGUUGGCUCGGGCGGAAAACCGCUGUCCGGGGGUGGCAACAUCAA -------------------((((.((.((((.((((........))))((((((.---...)))))).)))).)))))).(((((((((.......)))))))))((((....)))).. ( -47.10, z-score = -3.05, R) >triCas2.ChLG3 5084278 92 - 32080666 -------------GGCCUGGGGGCAACUGGCCCUCCAGCUGGGGCGGCCAG-------AGCCAGGGCAGCUG-GAGC--AACCAGCCGUGGAGUCAGGGCUC--CUGGGGCCAGGGC-- -------------(((((((..((..((((((((((....)))).))))))-------..((((.....)))-).))--..))).(((.(((((....))))--)))))))).....-- ( -50.30, z-score = -0.04, R) >consensus ___GGUACUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGACACCAAAG_AAGAUAGUCGCGGUGGCUAUGAGC__GGUCAGCUGGUCAAUCGCUGCCCAAGUAUGGCAAUAAC__ ...................(((.....((((.(((((((((((((..............)))))))))))))(((......)))))))...........)))................. (-25.60 = -25.65 + 0.05)
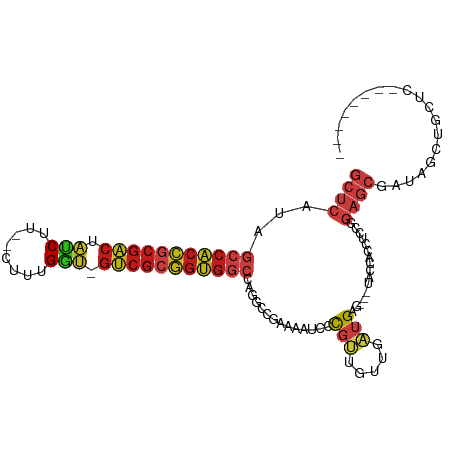
| Location | 14,521,031 – 14,521,126 |
|---|---|
| Length | 95 |
| Sequences | 14 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 76.33 |
| Shannon entropy | 0.51831 |
| G+C content | 0.59416 |
| Mean single sequence MFE | -37.71 |
| Consensus MFE | -25.35 |
| Energy contribution | -26.56 |
| Covariance contribution | 1.21 |
| Combinations/Pair | 1.38 |
| Mean z-score | -2.84 |
| Structure conservation index | 0.67 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.75 |
| SVM RNA-class probability | 0.999272 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
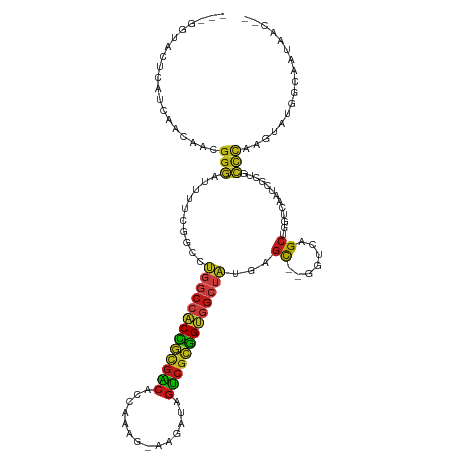
>dm3.chr2R 14521031 95 + 21146708 GCUCAUAGCCACCGCGACUAUCUU--CUUUGGU-GUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAG---UACCACCUCCCGAGCGAUAGCUACUC-------- (((....(((((((((((.(((..--....)))-)))))))))))..)))..........(((((((..(((---......))).....))))))).....-------- ( -34.70, z-score = -2.98, R) >droSim1.chr2R 13225423 95 + 19596830 GCUCAUAGCCACCGCGACUAUCUU--CUUUGGU-GUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAG---UACCACCUCCCGAGCGAUAGCUACUC-------- (((....(((((((((((.(((..--....)))-)))))))))))..)))..........(((((((..(((---......))).....))))))).....-------- ( -34.70, z-score = -2.98, R) >droSec1.super_1 12028749 95 + 14215200 GCUCAUAGCCACCGCGACUAUCUU--CUUUGGU-GUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAG---UACCACCUCCCGAGCGAUAGCUACUC-------- (((....(((((((((((.(((..--....)))-)))))))))))..)))..........(((((((..(((---......))).....))))))).....-------- ( -34.70, z-score = -2.98, R) >droYak2.chr2R 6456526 95 - 21139217 GCUCAUAGCCACCGCGACUAUCUU--CUUUGGU-GUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAG---CACCACCUCCCGAGCGAUAGCUGCUC-------- ((((((.(((((((((((.(((..--....)))-)))))))))))..((.........)).......)))))---)..........(((((.....)))))-------- ( -38.70, z-score = -3.61, R) >droEre2.scaffold_4845 8708839 95 + 22589142 GCUCAUAGCCACCGCGACUAUCUU--CUUUGGU-GUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAG---CACCACCCCCCGAGCGAUAGCUGCUC-------- ((((((.(((((((((((.(((..--....)))-)))))))))))..((.........)).......)))))---)..........(((((.....)))))-------- ( -38.70, z-score = -3.83, R) >droAna3.scaffold_13266 6887971 95 + 19884421 GCUCAUAGCCACCGCGACUAUCUU--CUUUGGU-GUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAG---CACUGCCUCCGGAGCGAUAACUGCUC-------- ((((((.(((((((((((.(((..--....)))-)))))))))))..((.........)).......)))))---)..........(((((.....)))))-------- ( -38.40, z-score = -3.21, R) >dp4.chr3 10581901 95 + 19779522 GCUCAUAGCCACCGCGACUAUCUU--CUUUGGU-GUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAG---UGCCUCCUCCGGAGCGGUAGCUACUC-------- ((((...(((((((((((.(((..--....)))-))))))))))).(((((....(((........)))..)---.))))......))))...........-------- ( -38.00, z-score = -2.66, R) >droPer1.super_4 5910412 95 + 7162766 GCUCAUAGCCACCGCGACUAUCUU--CUUUGGU-GUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAG---UGCCUCCUCCGGAGCGGUAGCUACUC-------- ((((...(((((((((((.(((..--....)))-))))))))))).(((((....(((........)))..)---.))))......))))...........-------- ( -38.00, z-score = -2.66, R) >droWil1.scaffold_180700 1809434 98 - 6630534 GCUCAUAGCCACCGCGACUAUCUU--CUUUGAU-GUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGCUGGCCUCCACCUCCAGAGCGAUAGCUGCUU-------- ((((...(((((((((((.(((..--....)))-))))))))))).((((((...(((........)))..)))))).........))))...........-------- ( -42.90, z-score = -4.79, R) >droVir3.scaffold_12875 9466528 96 - 20611582 GCUCAUAGCCACCGCGACUAUCUUU-CUUUGGU-GUCGCGGUGGCCAUGCCGAAAAUCCCGUUGUUGAUGAU---UGCCGCCGCCCGAGCGAUAGCUGCUC-------- ((.(((.(((((((((((.(((...-....)))-))))))))))).)))...........(((((((.....---.((....)).....))))))).))..-------- ( -34.60, z-score = -2.32, R) >droMoj3.scaffold_6496 15782002 98 + 26866924 GCUCAUAGCCACCGCGACUAUCUU--CUUUGGU-GUCGCGGUGGCCAUGCCGAAAAUCCCGUUGUUGAUGAU---UACCGCCGCCGGAGCGAUAGCUGCUGCUU----- ((.(((.(((((((((((.(((..--....)))-))))))))))).))).....((((.((....))..)))---)...)).((.(.(((....))).).))..----- ( -35.80, z-score = -2.09, R) >droGri2.scaffold_15245 13589662 98 + 18325388 GCUCAUAGCCACCGCGACUAUCUU--CUUUGGU-GUCGCGGUGGCCAUGCCGAAAAUCCCGUUGUUGCUGGUGGCUGCCGCCGCCGGAGCGAUAGCUGCUC-------- ((.(((.(((((((((((.(((..--....)))-))))))))))).)))...........(((((((((((((((....))))))..))))))))).))..-------- ( -50.30, z-score = -4.92, R) >anoGam1.chr2R 24316502 90 + 62725911 ------AGCCAACGCG--CGGCUG--CUCCGAG-AUCGGAACG---AAGCUAAAGUUCUUCCGCCGAGCGGU-----UGGACUUUGCAGCGACAGUGCUGGACCACUGC ------..(((..(((--(.((((--(((((..-..))))...---.....(((((((..((((...)))).-----.))))))))))))....)))))))........ ( -33.20, z-score = -0.52, R) >triCas2.ChLG3 5084314 85 + 32080666 ----------GCUCCAGCUGCCCUGGCUCUGGCCGCCCCAGCUGGAGGGCCAGUUGCCCCCAGGCCCCCG------UCCAACUUGGUUGCGAUUGCGGUUG-------- ----------.(((((((((...((((....))))...)))))))))((((((((((..(((((......------.....)))))..))))))..)))).-------- ( -35.30, z-score = -0.26, R) >consensus GCUCAUAGCCACCGCGACUAUCUU__CUUUGGU_GUCGCGGUGGCCAGGCCGAAAAUCCCGUUGUUGAUGAG___UACCACCUCCGGAGCGAUAGCUGCUC________ ((((...(((((((((((.(((........))).)))))))))))..............(((.....)))................))))................... (-25.35 = -26.56 + 1.21)

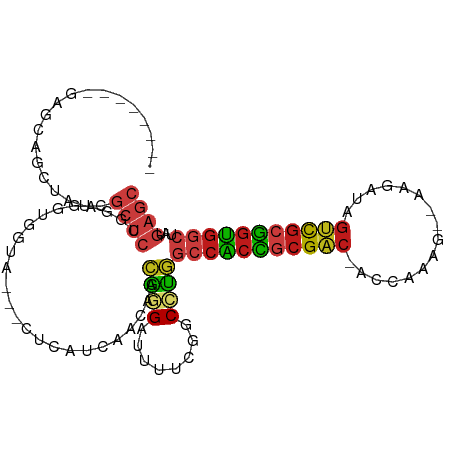
| Location | 14,521,031 – 14,521,126 |
|---|---|
| Length | 95 |
| Sequences | 14 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 76.33 |
| Shannon entropy | 0.51831 |
| G+C content | 0.59416 |
| Mean single sequence MFE | -39.98 |
| Consensus MFE | -24.57 |
| Energy contribution | -25.29 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.50 |
| Mean z-score | -2.98 |
| Structure conservation index | 0.61 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.78 |
| SVM RNA-class probability | 0.999313 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 14521031 95 - 21146708 --------GAGUAGCUAUCGCUCGGGAGGUGGUA---CUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGAC-ACCAAAG--AAGAUAGUCGCGGUGGCUAUGAGC --------(((((.(((((........)))))))---))).(((....((((........))))(((((((((((-.......--......)))))))))))..))).. ( -39.12, z-score = -3.24, R) >droSim1.chr2R 13225423 95 - 19596830 --------GAGUAGCUAUCGCUCGGGAGGUGGUA---CUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGAC-ACCAAAG--AAGAUAGUCGCGGUGGCUAUGAGC --------(((((.(((((........)))))))---))).(((....((((........))))(((((((((((-.......--......)))))))))))..))).. ( -39.12, z-score = -3.24, R) >droSec1.super_1 12028749 95 - 14215200 --------GAGUAGCUAUCGCUCGGGAGGUGGUA---CUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGAC-ACCAAAG--AAGAUAGUCGCGGUGGCUAUGAGC --------(((((.(((((........)))))))---))).(((....((((........))))(((((((((((-.......--......)))))))))))..))).. ( -39.12, z-score = -3.24, R) >droYak2.chr2R 6456526 95 + 21139217 --------GAGCAGCUAUCGCUCGGGAGGUGGUG---CUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGAC-ACCAAAG--AAGAUAGUCGCGGUGGCUAUGAGC --------(((((.(((((........)))))))---))).(((....((((........))))(((((((((((-.......--......)))))))))))..))).. ( -41.12, z-score = -3.20, R) >droEre2.scaffold_4845 8708839 95 - 22589142 --------GAGCAGCUAUCGCUCGGGGGGUGGUG---CUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGAC-ACCAAAG--AAGAUAGUCGCGGUGGCUAUGAGC --------(((((.(((((.(....).)))))))---))).(((....((((........))))(((((((((((-.......--......)))))))))))..))).. ( -42.42, z-score = -3.39, R) >droAna3.scaffold_13266 6887971 95 - 19884421 --------GAGCAGUUAUCGCUCCGGAGGCAGUG---CUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGAC-ACCAAAG--AAGAUAGUCGCGGUGGCUAUGAGC --------((((.......))))..........(---(((((......((((........))))(((((((((((-.......--......))))))))))).)))))) ( -37.92, z-score = -2.70, R) >dp4.chr3 10581901 95 - 19779522 --------GAGUAGCUACCGCUCCGGAGGAGGCA---CUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGAC-ACCAAAG--AAGAUAGUCGCGGUGGCUAUGAGC --------((((.(((.((........)).))))---))).(((....((((........))))(((((((((((-.......--......)))))))))))..))).. ( -39.42, z-score = -3.27, R) >droPer1.super_4 5910412 95 - 7162766 --------GAGUAGCUACCGCUCCGGAGGAGGCA---CUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGAC-ACCAAAG--AAGAUAGUCGCGGUGGCUAUGAGC --------((((.(((.((........)).))))---))).(((....((((........))))(((((((((((-.......--......)))))))))))..))).. ( -39.42, z-score = -3.27, R) >droWil1.scaffold_180700 1809434 98 + 6630534 --------AAGCAGCUAUCGCUCUGGAGGUGGAGGCCAGCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGAC-AUCAAAG--AAGAUAGUCGCGGUGGCUAUGAGC --------...........((((.........(((((((.(((........))).)).)))))((((((((((((-(((....--..))).))))))))))))..)))) ( -41.50, z-score = -3.27, R) >droVir3.scaffold_12875 9466528 96 + 20611582 --------GAGCAGCUAUCGCUCGGGCGGCGGCA---AUCAUCAACAACGGGAUUUUCGGCAUGGCCACCGCGAC-ACCAAAG-AAAGAUAGUCGCGGUGGCUAUGAGC --------..((.(((..(((....)))..))).---.......................(((((((((((((((-.......-.......))))))))))))))).)) ( -40.24, z-score = -3.75, R) >droMoj3.scaffold_6496 15782002 98 - 26866924 -----AAGCAGCAGCUAUCGCUCCGGCGGCGGUA---AUCAUCAACAACGGGAUUUUCGGCAUGGCCACCGCGAC-ACCAAAG--AAGAUAGUCGCGGUGGCUAUGAGC -----.(((....)))...((((....(.(((.(---(((.((......)))))).))).)((((((((((((((-.......--......)))))))))))))))))) ( -39.42, z-score = -3.15, R) >droGri2.scaffold_15245 13589662 98 - 18325388 --------GAGCAGCUAUCGCUCCGGCGGCGGCAGCCACCAGCAACAACGGGAUUUUCGGCAUGGCCACCGCGAC-ACCAAAG--AAGAUAGUCGCGGUGGCUAUGAGC --------((((.......)))).((.(((....))).)).((.....(((.....))).(((((((((((((((-.......--......))))))))))))))).)) ( -43.72, z-score = -3.49, R) >anoGam1.chr2R 24316502 90 - 62725911 GCAGUGGUCCAGCACUGUCGCUGCAAAGUCCA-----ACCGCUCGGCGGAAGAACUUUAGCUU---CGUUCCGAU-CUCGGAG--CAGCCG--CGCGUUGGCU------ (((((((..((....)))))))))..((.(((-----((.((.((((.((((........)))---)((((((..-..)))))--).))))--.)))))))))------ ( -40.00, z-score = -2.68, R) >triCas2.ChLG3 5084314 85 - 32080666 --------CAACCGCAAUCGCAACCAAGUUGGA------CGGGGGCCUGGGGGCAACUGGCCCUCCAGCUGGGGCGGCCAGAGCCAGGGCAGCUGGAGC---------- --------...((((..((.(..((.....)).------.).))((((...(((..((((((((((....)))).)))))).))).)))).)).))...---------- ( -37.20, z-score = 0.17, R) >consensus ________GAGCAGCUAUCGCUCCGGAGGUGGUA___CUCAUCAACAACGGGAUUUUCGGCCUGGCCACCGCGAC_ACCAAAG__AAGAUAGUCGCGGUGGCUAUGAGC ...................((((.........................((((........))))(((((((((((...(........)...)))))))))))...)))) (-24.57 = -25.29 + 0.72)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:32:31 2011