
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 14,489,227 – 14,489,375 |
| Length | 148 |
| Max. P | 0.542535 |

| Location | 14,489,227 – 14,489,375 |
|---|---|
| Length | 148 |
| Sequences | 7 |
| Columns | 166 |
| Reading direction | reverse |
| Mean pairwise identity | 69.56 |
| Shannon entropy | 0.54267 |
| G+C content | 0.48418 |
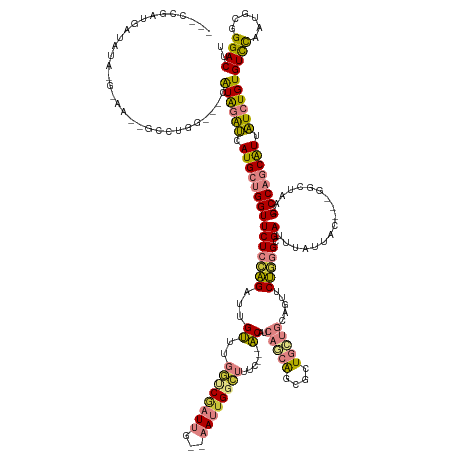
| Mean single sequence MFE | -50.08 |
| Consensus MFE | -20.73 |
| Energy contribution | -20.16 |
| Covariance contribution | -0.58 |
| Combinations/Pair | 1.46 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.41 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.542535 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
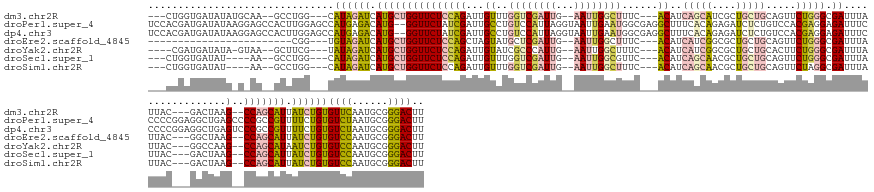
>dm3.chr2R 14489227 148 - 21146708 ---CUGGUGAUAUAUGCAA--GCCUGG---CAUAGAUCAUGCUGGUUCUCCAGAUUGUUUGGUCGAUUG--AAUUGGCUUUC---ACAUCAGCAUCGCUGCUGCAGUUCUGGGCGAUUUAUUAC---GACUAAG--CCAGCAUUAUCUGUGUUCAAUGCGGGACUU ---..............((--((((((---(((((((.((((((((((....))....(((((((..((--(((((.((...---((..(((((....)))))..))...)).)))))))...)---)))))))--))))))).))))))))......)))).))) ( -48.40, z-score = -1.41, R) >droPer1.super_4 4542078 164 + 7162766 UCCACGAUGAUAUAAGGAGCCACUUGGAGCCAUGAGACAUG--GGUUCUAUCGAUUGCCUGUCCAUUAGGUAAUUGAAUGGCGAGGCUUUCACAGAGAUCUCUGUCCACGAGGAGAUUUCCCCCGGAGGCUGAGCCCCGCCGUUUUCUGUGUCUAAUGCGGGACUU (((.((..((((((..((((....((((((((((...))).--)))))))(((((((((((.....)))))))))))..((((.(((((...(.(((((((((........)))))))))(....).)...))))).))))))))..)))))).....)))))... ( -61.00, z-score = -2.21, R) >dp4.chr3 11042412 164 + 19779522 UCCACGAUGAUAUAAGGAGCCACUUGGAGCCAUGAGACAUG--GGUUCUAUCGAUUGCCUGUCCAUUAGGUAAUUGAAUGGCGAGGCUUUCACAGAGAUCUCUGUCCACGAGGAGAUUUCCCCCGGAGGCUGAGUCCCGCCGUUUUCUGUGUCUAAUGCGGGACUU (((............)))((((..((((((((((...))).--)))))))(((((((((((.....))))))))))).))))..(((((((...(((((((((........)))))))))....)))))))(((((((((.(((..........)))))))))))) ( -61.00, z-score = -2.21, R) >droEre2.scaffold_4845 8676798 129 - 22589142 ------------------------CGG---UGUAGAUCAUGCUGGUUCUCCAGCUAGUAUGCUCGAUUG--AAUUGGCUUUC---ACAUCAUCGGCGCUGCUGCAGUUCUGGGCGAUUUAUUAC---GGCUAAG--CCAGCAUUAUCUGUGUCCAAUGCGGGACUU ------------------------...---..(((((.(((((((((((.((((.(((..((.((((((--((......)))---)....))))))))))))).))...(((.((........)---).)))))--))))))).))))).((((......)))).. ( -40.50, z-score = -0.44, R) >droYak2.chr2R 6424641 146 + 21139217 ----CGAUGAUAUA-GUAA--GCUUCG---UAUAGAUCAUGCUGGUUCUCCAGAUUGUAUCGCCCAUUG--AAUUGGCUUUC---ACAUCAUCGGCGCUGCUGCACUUCUGGGCGAUUUAUUAC---GGCCAAG--CCAGCAUAAUCUGUGUCCAAUGCGGGACUU ----..((((....-....--...)))---)((((((.(((((((((..((.......((((((((.((--((......)))---).......((.((....)).))..)))))))).......---))...))--))))))).))))))((((......)))).. ( -43.84, z-score = -0.82, R) >droSec1.super_1 11942841 144 - 14215200 ---CUGGUGAUAU----AA--GCCUGG---CAUAGAUCAUGCUGGUUCUCCAGAUUGUUUGGUCGAUUG--AAUUGGCGUUC---ACAUCAGCAACGCUGCUGCAGUUCUGGGCGAUUUAUUAC---GACUAAG--CCAGCAUUAUCUGUGUCCAAUGCGGGACUU ---..(((.....----..--)))(((---(((((((.((((((((((....))....(((((((..((--(((((.(....---((..(((((....)))))..))....).)))))))...)---)))))))--))))))).))))))).)))........... ( -46.70, z-score = -1.27, R) >droSim1.chr2R 13193419 144 - 19596830 ---CUGGUGAUAU----AA--GCCUGG---CAUAGAUCAUGCUGGUUCUCCAGAUUGUUUGGUCGAUUG--AAUUGGCUUUC---ACAUCAGCAACGCUGCUGCAGUUCUAGGCGAUUUAUUAC---GACUAAG--CCAGCAUUAUCUGUGUCCAAUGCGGGACUU ---..(((.....----..--)))(((---(((((((.((((((((((....))....(((((((..((--(((((.((...---((..(((((....)))))..))...)).)))))))...)---)))))))--))))))).))))))).)))........... ( -49.10, z-score = -2.33, R) >consensus ___CCGAUGAUAUA_G_AA__GCCUGG___CAUAGAUCAUGCUGGUUCUCCAGAUUGUUUGGUCGAUUG__AAUUGGCUUUC___ACAUCAGCAGCGCUGCUGCAGUUCUGGGCGAUUUAUUAC___GGCUAAG__CCAGCAUUAUCUGUGUCCAAUGCGGGACUU ...............................((((((.(((((((.(((((((........((((((.....))))))...........(((((....))))).....))))).))....((.....)).......))))))).))))))((((......)))).. (-20.73 = -20.16 + -0.58)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:32:22 2011