
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 13,450,116 – 13,450,209 |
| Length | 93 |
| Max. P | 0.998460 |

| Location | 13,450,116 – 13,450,209 |
|---|---|
| Length | 93 |
| Sequences | 12 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 68.08 |
| Shannon entropy | 0.62177 |
| G+C content | 0.52312 |
| Mean single sequence MFE | -41.98 |
| Consensus MFE | -8.32 |
| Energy contribution | -7.38 |
| Covariance contribution | -0.93 |
| Combinations/Pair | 1.71 |
| Mean z-score | -4.50 |
| Structure conservation index | 0.20 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.37 |
| SVM RNA-class probability | 0.998460 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
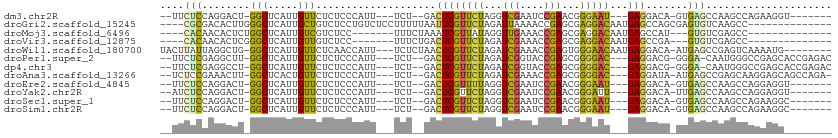
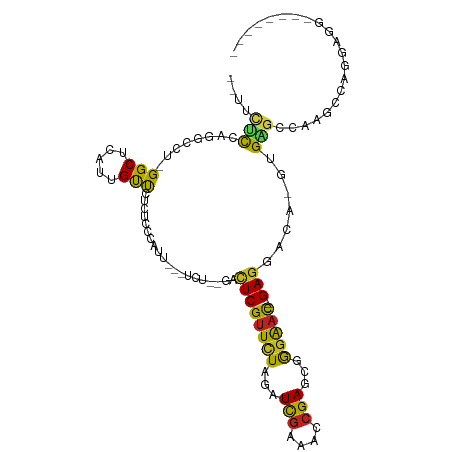
>dm3.chr2R 13450116 93 + 21146708 --UUCUCCAGGACU-GGCUCAUUGUUCUCUCCCAUU---UCU--GACUCGUUCUAGGUCGAAUCCGAACGGGAAU---GAGGACA-GUGAGCCAAGCCAGAAGGU------- --((((...((..(-((((((((((((((((((..(---((.--((.(((........))).)).))).))))..---)))))))-))))))))..))))))...------- ( -40.70, z-score = -3.78, R) >droGri2.scaffold_15245 17909391 94 - 18325388 ----CGCGACACUUGGGCUCAUUGCUGUCUCCUGUCUCCUUUUUAAUUCGUUCUAGAUUAAAACCGAGCGAGGACAAUGAGCCAGCGAGUGUCAAGCC-------------- ----...(((((((((((((((((.....((((..(((..((((((((.......))))))))..)))..)))))))))))))..)))))))).....-------------- ( -35.90, z-score = -4.04, R) >droMoj3.scaffold_6496 1337836 84 - 26866924 ----CACAACACUCUGGCUCAUUGUCGUCUCC-------UUUCUAAAUCGUUAUAGGUUGAAACCGAGCGAGGACAAUGAGCCAU---GUGUCGAGCC-------------- ----..(.((((..(((((((((((((.....-------...)....(((((...(((....))).))))).)))))))))))).---)))).)....-------------- ( -30.60, z-score = -3.93, R) >droVir3.scaffold_12875 4542500 84 + 20611582 ----CACAACACUCGGGCUCAUUGUUGUCUCC-------UUUCUGACUCGUUCUAGAUCGAAACCGAGCGAGGACAAUGAGCCGA---GUGUCGAGCC-------------- ----..(.((((((((.(((((((((.((...-------.....))(((((((............))))))))))))))))))))---)))).)....-------------- ( -34.90, z-score = -3.76, R) >droWil1.scaffold_180700 4785733 99 + 6630534 UACUUAUUAGGCUG-GGCUCAUUGUUCUCAACCAUU---UCUCUAACUCGUUCUAGAUCGAAACCGAGUGGGAACAAUGAGGACA-AUGAGCCGAGUCAAAAUG-------- .........((((.-(((((((((((((((.....(---((((..((((((((......)))..))))))))))...))))))))-))))))).))))......-------- ( -42.00, z-score = -6.57, R) >droPer1.super_2 5554254 99 + 9036312 --UUCUCGAGGCUU-GGCUCAUUGUUCUCUCCCAUU---UCU--GACUCGUUCUAGAUCGGUACCGAGCGGGGAC---GAGGACG-GGGA-CAAUGGGCCGAGCACCGAGAC --.(((((..((((-(((((((((((((((((((..---..)--)..(((((((...(((....)))...)))))---))))).)-))))-)))))))))))))..))))). ( -55.60, z-score = -6.36, R) >dp4.chr3 5349019 99 + 19779522 --UUCUCGAGGCCU-GGCUCAUUGUUCUCUCCCAUU---UCU--GACUCGUUCUAGAUCGGUACCGAGCGGGGAC---GAGGACG-GGGA-CAAUGGGCCGAGCACCGAGAC --.(((((..((.(-(((((((((((((((((((..---..)--)..(((((((...(((....)))...)))))---))))).)-))))-)))))))))).))..))))). ( -51.10, z-score = -4.70, R) >droAna3.scaffold_13266 9085599 99 - 19884421 --UCUCCGAAACUU-GGCUCACUGUUCUCUCCCAUU---UCU--GACUCGUUCUAGAUCGAAACCGAGCGGGGAC---GAGGAUA-AUGAGCCGAGCAAGGAGCAGCCAGA- --.((((....(((-((((((.(((((((((((..(---((.--(..(((........)))..).)))..)))).---)))))))-.)))))))))...))))........- ( -39.70, z-score = -2.93, R) >droEre2.scaffold_4845 7677359 93 + 22589142 --UUCUCCAGGACU-GGCUCAUUGUUCUCUCCCAUU---UCU--GACUCGUUUUAGGUCGAAUCCGAACGGGAAU---GAGGACA-GUGAGCCAAGCCAGGAGGU------- --.(((((.((..(-((((((((((((((((((..(---((.--((.(((........))).)).))).))))..---)))))))-))))))))..)).))))).------- ( -47.20, z-score = -5.57, R) >droYak2.chr2R 5402307 93 - 21139217 --AUCUCCAGGACU-GGCUCAUUGUUCUCUCCCAUU---UCU--GACUCGUUCUAGGUCGAAUCCGAACGGGAUU---GAGGACA-UUGAGCCAAGCCAGGAGGU------- --((((((.((..(-((((((.(((((((((((..(---((.--((.(((........))).)).))).))))..---)))))))-.)))))))..)).))))))------- ( -44.60, z-score = -4.81, R) >droSec1.super_1 10949536 93 + 14215200 --UUCUCCAGGACU-GGCUCAUUGUUCUCUCCCAUU---UCU--GACUCGUUCUAGGUCGAAUCCGAACGGGAAU---GAGGACA-GUGAGCCAAGCCAGAAGGC------- --((((...((..(-((((((((((((((((((..(---((.--((.(((........))).)).))).))))..---)))))))-))))))))..))))))...------- ( -40.70, z-score = -3.80, R) >droSim1.chr2R 12186170 93 + 19596830 --UUCUCCAGGACU-GGCUCAUUGUUCUCUCCCAUU---UCU--GACUCGUUCUAGGUCGAAUCCGAACGGGAAU---GAGGACA-GUGAGCCAAGCCAGAAGGC------- --((((...((..(-((((((((((((((((((..(---((.--((.(((........))).)).))).))))..---)))))))-))))))))..))))))...------- ( -40.70, z-score = -3.80, R) >consensus __UUCUCCAGGCCU_GGCUCAUUGUUCUCUCCCAUU___UCU__GACUCGUUCUAGAUCGAAACCGAGCGGGAAC___GAGGACA_GUGAGCCAAGCCAGGAGG________ ....(((........(((.....)))....................((((((((...(((....)))...)))))...))).......)))..................... ( -8.32 = -7.38 + -0.93)
| Location | 13,450,116 – 13,450,209 |
|---|---|
| Length | 93 |
| Sequences | 12 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 68.08 |
| Shannon entropy | 0.62177 |
| G+C content | 0.52312 |
| Mean single sequence MFE | -38.39 |
| Consensus MFE | -2.51 |
| Energy contribution | -2.61 |
| Covariance contribution | 0.10 |
| Combinations/Pair | 1.22 |
| Mean z-score | -3.71 |
| Structure conservation index | 0.07 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.72 |
| SVM RNA-class probability | 0.798813 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
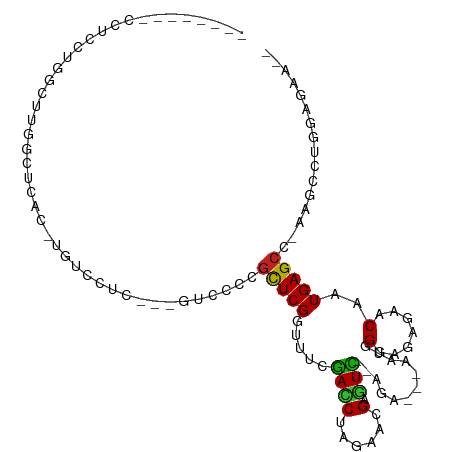
>dm3.chr2R 13450116 93 - 21146708 -------ACCUUCUGGCUUGGCUCAC-UGUCCUC---AUUCCCGUUCGGAUUCGACCUAGAACGAGUC--AGA---AAUGGGAGAGAACAAUGAGCC-AGUCCUGGAGAA-- -------..((((.((.((((((((.-(((.(((---..((((((((.((((((........))))))--.).---)))))))))).))).))))))-)).)).))))..-- ( -42.40, z-score = -4.51, R) >droGri2.scaffold_15245 17909391 94 + 18325388 --------------GGCUUGACACUCGCUGGCUCAUUGUCCUCGCUCGGUUUUAAUCUAGAACGAAUUAAAAAGGAGACAGGAGACAGCAAUGAGCCCAAGUGUCGCG---- --------------.((..((((((....(((((((((((((..(((..((((((((......).)))))))..)))..))).....))))))))))..)))))))).---- ( -33.30, z-score = -2.88, R) >droMoj3.scaffold_6496 1337836 84 + 26866924 --------------GGCUCGACAC---AUGGCUCAUUGUCCUCGCUCGGUUUCAACCUAUAACGAUUUAGAAA-------GGAGACGACAAUGAGCCAGAGUGUUGUG---- --------------....((((((---.((((((((((((.(((...(((....))).....)))........-------(....)))))))))))))..))))))..---- ( -32.30, z-score = -4.06, R) >droVir3.scaffold_12875 4542500 84 - 20611582 --------------GGCUCGACAC---UCGGCUCAUUGUCCUCGCUCGGUUUCGAUCUAGAACGAGUCAGAAA-------GGAGACAACAAUGAGCCCGAGUGUUGUG---- --------------....((((((---((((((((((((.((.(((((...((......)).))))).))...-------(....).)))))))).))))))))))..---- ( -37.70, z-score = -4.23, R) >droWil1.scaffold_180700 4785733 99 - 6630534 --------CAUUUUGACUCGGCUCAU-UGUCCUCAUUGUUCCCACUCGGUUUCGAUCUAGAACGAGUUAGAGA---AAUGGUUGAGAACAAUGAGCC-CAGCCUAAUAAGUA --------........((.(((((((-(((.((((...(((..(((((...((......)).)))))..))).---......)))).))))))))))-.))........... ( -30.00, z-score = -2.86, R) >droPer1.super_2 5554254 99 - 9036312 GUCUCGGUGCUCGGCCCAUUG-UCCC-CGUCCUC---GUCCCCGCUCGGUACCGAUCUAGAACGAGUC--AGA---AAUGGGAGAGAACAAUGAGCC-AAGCCUCGAGAA-- .((((((.(((.(((.(((((-((((-(((.(((---((.(..(.(((....))).)..).)))))..--...---.))))).))...))))).)))-.))).)))))).-- ( -37.20, z-score = -2.61, R) >dp4.chr3 5349019 99 - 19779522 GUCUCGGUGCUCGGCCCAUUG-UCCC-CGUCCUC---GUCCCCGCUCGGUACCGAUCUAGAACGAGUC--AGA---AAUGGGAGAGAACAAUGAGCC-AGGCCUCGAGAA-- .((((((.((..(((.(((((-((((-(((.(((---((.(..(.(((....))).)..).)))))..--...---.))))).))...))))).)))-..)).)))))).-- ( -37.30, z-score = -1.98, R) >droAna3.scaffold_13266 9085599 99 + 19884421 -UCUGGCUGCUCCUUGCUCGGCUCAU-UAUCCUC---GUCCCCGCUCGGUUUCGAUCUAGAACGAGUC--AGA---AAUGGGAGAGAACAGUGAGCC-AAGUUUCGGAGA-- -........((((..(((.(((((((-(...(((---.((((.(((((...((......)).))))).--(..---..))))))))...))))))))-.)))...)))).-- ( -35.10, z-score = -1.08, R) >droEre2.scaffold_4845 7677359 93 - 22589142 -------ACCUCCUGGCUUGGCUCAC-UGUCCUC---AUUCCCGUUCGGAUUCGACCUAAAACGAGUC--AGA---AAUGGGAGAGAACAAUGAGCC-AGUCCUGGAGAA-- -------..((((.((.((((((((.-(((.(((---..((((((((.((((((........))))))--.).---)))))))))).))).))))))-)).)).))))..-- ( -45.30, z-score = -5.84, R) >droYak2.chr2R 5402307 93 + 21139217 -------ACCUCCUGGCUUGGCUCAA-UGUCCUC---AAUCCCGUUCGGAUUCGACCUAGAACGAGUC--AGA---AAUGGGAGAGAACAAUGAGCC-AGUCCUGGAGAU-- -------..((((.((.((((((((.-(((.(((---..((((((((.((((((........))))))--.).---)))))))))).))).))))))-)).)).))))..-- ( -45.30, z-score = -5.71, R) >droSec1.super_1 10949536 93 - 14215200 -------GCCUUCUGGCUUGGCUCAC-UGUCCUC---AUUCCCGUUCGGAUUCGACCUAGAACGAGUC--AGA---AAUGGGAGAGAACAAUGAGCC-AGUCCUGGAGAA-- -------..((((.((.((((((((.-(((.(((---..((((((((.((((((........))))))--.).---)))))))))).))).))))))-)).)).))))..-- ( -42.40, z-score = -4.39, R) >droSim1.chr2R 12186170 93 - 19596830 -------GCCUUCUGGCUUGGCUCAC-UGUCCUC---AUUCCCGUUCGGAUUCGACCUAGAACGAGUC--AGA---AAUGGGAGAGAACAAUGAGCC-AGUCCUGGAGAA-- -------..((((.((.((((((((.-(((.(((---..((((((((.((((((........))))))--.).---)))))))))).))).))))))-)).)).))))..-- ( -42.40, z-score = -4.39, R) >consensus ________CCUCCUGGCUUGGCUCAC_UGUCCUC___GUCCCCGCUCGGUUUCGACCUAGAACGAGUC__AGA___AAUGGGAGAGAACAAUGAGCC_AAGCCUGGAGAA__ ...........................................(((((...(((........)))...............(.......)..)))))................ ( -2.51 = -2.61 + 0.10)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:30:03 2011