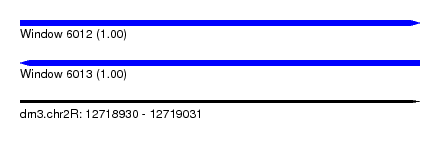
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 12,718,930 – 12,719,031 |
| Length | 101 |
| Max. P | 0.999476 |

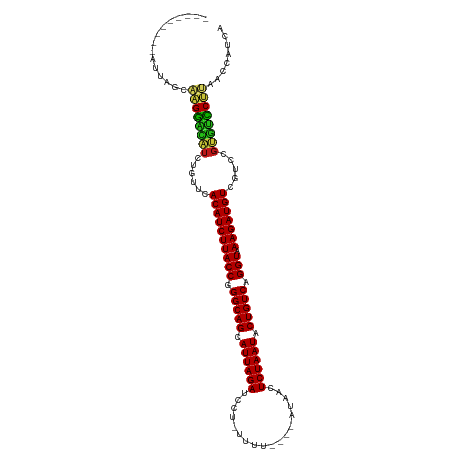
| Location | 12,718,930 – 12,719,031 |
|---|---|
| Length | 101 |
| Sequences | 15 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 82.85 |
| Shannon entropy | 0.38380 |
| G+C content | 0.41286 |
| Mean single sequence MFE | -32.91 |
| Consensus MFE | -30.59 |
| Energy contribution | -30.44 |
| Covariance contribution | -0.15 |
| Combinations/Pair | 1.31 |
| Mean z-score | -4.80 |
| Structure conservation index | 0.93 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.56 |
| SVM RNA-class probability | 0.998944 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 12718930 101 + 21146708 ---------AUUAGCAAGGACAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUCCU-UUUU----AUAACUCUAAUACUGUCAGGUAAAGAUGUCGUCCGUGUCCUUAACCUUCA ---------......((((((((..(..(((((((((((.(((((.((((((....-....----.....)))))).))))).))).))))))).)..)))))))))........ ( -32.42, z-score = -5.31, R) >apiMel3.Group11 2265690 98 + 12576330 ---------AUCGGACGGAGUAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUUGA-AGUU----GACCUUCUAAUACUGUCAGGUAAAGAUGUCGUCAGGAUUCCGAAAUC--- ---------......((((((.((((...((((((((((.(((((.((((((....-....----.....)))))).))))).))).)))))))...)))))))))).....--- ( -35.52, z-score = -4.04, R) >droSim1.chr2R 11454616 101 + 19596830 ---------AUUAGCAAGGACAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUCCU-UUUU----AUAACUCUAAUACUGUCAGGUAAAGAUGUCGUCCGUGUCCUUAACCUUCA ---------......((((((((..(..(((((((((((.(((((.((((((....-....----.....)))))).))))).))).))))))).)..)))))))))........ ( -32.42, z-score = -5.31, R) >droSec1.super_1 10223801 101 + 14215200 ---------AUUAGCAAGGACAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUCCU-UUUU----AAAACUCUAAUACUGUCAGGUAAAGAUGUCGUCCGUGUCCUUAACCUUCA ---------......((((((((..(..(((((((((((.(((((.((((((....-....----.....)))))).))))).))).))))))).)..)))))))))........ ( -32.42, z-score = -5.35, R) >droYak2.chr2R 4881892 101 - 21139217 ---------AUUAGCAAGGACAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUCCU-UUUU----AUAACUCUAAUACUGUCAGGUAAAGAUGUCGUCCGUGUCCUUAACCUUCA ---------......((((((((..(..(((((((((((.(((((.((((((....-....----.....)))))).))))).))).))))))).)..)))))))))........ ( -32.42, z-score = -5.31, R) >droEre2.scaffold_4845 9457919 101 - 22589142 ---------AUUAGCAAGGACAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUCCU-UUUU----AUAACUCUAAUACUGUCAGGUAAAGAUGUCGUCCGUGUCCUUAACCUUCA ---------......((((((((..(..(((((((((((.(((((.((((((....-....----.....)))))).))))).))).))))))).)..)))))))))........ ( -32.42, z-score = -5.31, R) >droAna3.scaffold_13266 6007682 102 - 19884421 ---------AUUUGCAAGGACAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUCCU-UUCUG---AUAACUCUAAUACUGUCAGGUAAAGAUGUCGUCCGUGUCCUUAACCAUCG ---------......((((((((..(..(((((((((((.(((((.((((((....-.....---.....)))))).))))).))).))))))).)..)))))))))........ ( -32.34, z-score = -4.50, R) >dp4.Unknown_singleton_1898 32506 101 - 80150 ---------AUUCGCAAGGACAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUCCU-UUAG----AUACCUCUAAUACUGUCAGGUAAAGAUGUCGUCCGUGUCCUUAUCCAUCA ---------......((((((((..(..(((((((((((.(((((.((((((....-....----.....)))))).))))).))).))))))).)..)))))))))........ ( -32.42, z-score = -4.74, R) >droPer1.super_4 6545758 101 + 7162766 ---------AUUCGCAAGGACAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUCCU-UUAG----AUACCUCUAAUACUGUCAGGUAAAGAUGUCGUCCGUGUCCUUAUCCAUCA ---------......((((((((..(..(((((((((((.(((((.((((((....-....----.....)))))).))))).))).))))))).)..)))))))))........ ( -32.42, z-score = -4.74, R) >droWil1.scaffold_180700 6381470 115 + 6630534 AAAGAUUCUUCUUGCAAGGACAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUUUUGUACUAUAGAUAUCUCUAAUACUGUCAGGUAAAGAUGUCGUCCGUGUCCUUAUCCAUCA .((((....))))..((((((((..(..(((((((((((.(((((.((((((.(((((...)))))....)))))).))))).))).))))))).)..)))))))))........ ( -34.40, z-score = -4.23, R) >droGri2.scaffold_15245 14542150 109 - 18325388 -----AUUUGCUUGCAAGGACAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUUCU-UGAUUUAAAUAAUUCUAAUACUGUCAGGUAAAGAUGUCGUCCGUGUCCUUAUCCAUCA -----..........((((((((..(..(((((((((((.(((((.((((((....-.............)))))).))))).))).))))))).)..)))))))))........ ( -32.93, z-score = -4.48, R) >droMoj3.scaffold_6496 16285329 105 + 26866924 ---------AUUUGCUAGGACAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUUCC-UGUUUUAAAUAACUCUAAUACUGUCAGGUAAAGAUGUCGUCCGUGUCCUUUUCCAUCA ---------.......(((((((..(..(((((((((((.(((((.((((((....-.(((......))))))))).))))).))).))))))).)..))))))))......... ( -31.30, z-score = -4.60, R) >droVir3.scaffold_12875 9939091 105 - 20611582 ---------AUUUGCAAGGACAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUUCU-UGAUUUAAAUAAUUCUAAUACUGUCAGGUAAAGAUGUCGUCCGUGUCCUUAUACAUCA ---------......((((((((..(..(((((((((((.(((((.((((((....-.............)))))).))))).))).))))))).)..)))))))))........ ( -32.93, z-score = -5.05, R) >triCas2.ChLG9 7640708 95 - 15222296 ----------------AGGAGUACUGUUAACAUCUUACCGGGCAGCAUUAGAUUACAUUUU----AUACUUCUAAUACUGUCAGGUAAAGAUGUCCUCCGGGCUCCAAUCCACCA ----------------.(((((.(((...((((((((((.(((((.((((((.........----.....)))))).))))).))).)))))))....))))))))......... ( -32.54, z-score = -4.71, R) >anoGam1.chr3L 38263618 95 + 41284009 -----------------GGGUGUCUGUUCACAUCUUACCGGGCAGCAUUAGAUAUGUUAUCG--GAUAUUUCUAAUACUGUCAGGUAAAGAUGUCGUCCGAGCCCAUGCUCAUC- -----------------((((...((..(((((((((((.(((((.((((((.(((((....--))))).)))))).))))).))).))))))).)..)).)))).........- ( -34.80, z-score = -4.27, R) >consensus _________AUUAGCAAGGACAUCUGUUCACAUCUUACCGGGCAGCAUUAGAUCCU_UUUU____AUAACUCUAAUACUGUCAGGUAAAGAUGUCGUCCGUGUCCUUAACCAUCA ...............((((((((......((((((((((.(((((.((((((..................)))))).))))).))).))))))).....))))))))........ (-30.59 = -30.44 + -0.15)

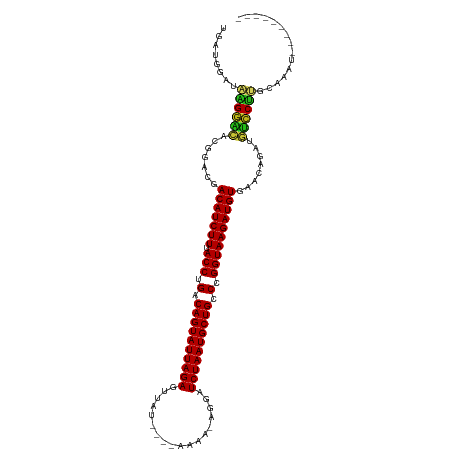
| Location | 12,718,930 – 12,719,031 |
|---|---|
| Length | 101 |
| Sequences | 15 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 82.85 |
| Shannon entropy | 0.38380 |
| G+C content | 0.41286 |
| Mean single sequence MFE | -33.02 |
| Consensus MFE | -28.03 |
| Energy contribution | -27.92 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.26 |
| Mean z-score | -4.20 |
| Structure conservation index | 0.85 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.92 |
| SVM RNA-class probability | 0.999476 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 12718930 101 - 21146708 UGAAGGUUAAGGACACGGACGACAUCUUUACCUGACAGUAUUAGAGUUAU----AAAA-AGGAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUGUCCUUGCUAAU--------- ....(((.((((((((.....(((((((.(((.(.((((((((((.....----....-....)))))))))).).)))))))))).....).))))))))))...--------- ( -34.42, z-score = -4.79, R) >apiMel3.Group11 2265690 98 - 12576330 ---GAUUUCGGAAUCCUGACGACAUCUUUACCUGACAGUAUUAGAAGGUC----AACU-UCAAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUACUCCGUCCGAU--------- ---....(((((...(((...(((((((.(((.(.(((((((((((((..----..))-)...)))))))))).).))))))))))...)))........))))).--------- ( -30.90, z-score = -3.60, R) >droSim1.chr2R 11454616 101 - 19596830 UGAAGGUUAAGGACACGGACGACAUCUUUACCUGACAGUAUUAGAGUUAU----AAAA-AGGAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUGUCCUUGCUAAU--------- ....(((.((((((((.....(((((((.(((.(.((((((((((.....----....-....)))))))))).).)))))))))).....).))))))))))...--------- ( -34.42, z-score = -4.79, R) >droSec1.super_1 10223801 101 - 14215200 UGAAGGUUAAGGACACGGACGACAUCUUUACCUGACAGUAUUAGAGUUUU----AAAA-AGGAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUGUCCUUGCUAAU--------- ....(((.((((((((.....(((((((.(((.(.((((((((((.((((----....-)))))))))))))).).)))))))))).....).))))))))))...--------- ( -34.70, z-score = -4.75, R) >droYak2.chr2R 4881892 101 + 21139217 UGAAGGUUAAGGACACGGACGACAUCUUUACCUGACAGUAUUAGAGUUAU----AAAA-AGGAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUGUCCUUGCUAAU--------- ....(((.((((((((.....(((((((.(((.(.((((((((((.....----....-....)))))))))).).)))))))))).....).))))))))))...--------- ( -34.42, z-score = -4.79, R) >droEre2.scaffold_4845 9457919 101 + 22589142 UGAAGGUUAAGGACACGGACGACAUCUUUACCUGACAGUAUUAGAGUUAU----AAAA-AGGAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUGUCCUUGCUAAU--------- ....(((.((((((((.....(((((((.(((.(.((((((((((.....----....-....)))))))))).).)))))))))).....).))))))))))...--------- ( -34.42, z-score = -4.79, R) >droAna3.scaffold_13266 6007682 102 + 19884421 CGAUGGUUAAGGACACGGACGACAUCUUUACCUGACAGUAUUAGAGUUAU---CAGAA-AGGAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUGUCCUUGCAAAU--------- .....((.((((((((.....(((((((.(((.(.((((((((((.....---.....-....)))))))))).).)))))))))).....).)))))))))....--------- ( -33.44, z-score = -3.79, R) >dp4.Unknown_singleton_1898 32506 101 + 80150 UGAUGGAUAAGGACACGGACGACAUCUUUACCUGACAGUAUUAGAGGUAU----CUAA-AGGAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUGUCCUUGCGAAU--------- .....(.(((((((((.....(((((((.(((.(.((((((((((.....----....-....)))))))))).).)))))))))).....).)))))))))....--------- ( -33.52, z-score = -3.85, R) >droPer1.super_4 6545758 101 - 7162766 UGAUGGAUAAGGACACGGACGACAUCUUUACCUGACAGUAUUAGAGGUAU----CUAA-AGGAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUGUCCUUGCGAAU--------- .....(.(((((((((.....(((((((.(((.(.((((((((((.....----....-....)))))))))).).)))))))))).....).)))))))))....--------- ( -33.52, z-score = -3.85, R) >droWil1.scaffold_180700 6381470 115 - 6630534 UGAUGGAUAAGGACACGGACGACAUCUUUACCUGACAGUAUUAGAGAUAUCUAUAGUACAAAAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUGUCCUUGCAAGAAGAAUCUUU .....(.(((((((((.....(((((((.(((.(.((((((((((..................)))))))))).).)))))))))).....).)))))))))............. ( -33.17, z-score = -3.51, R) >droGri2.scaffold_15245 14542150 109 + 18325388 UGAUGGAUAAGGACACGGACGACAUCUUUACCUGACAGUAUUAGAAUUAUUUAAAUCA-AGAAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUGUCCUUGCAAGCAAAU----- ((...(.(((((((((.....(((((((.(((.(.((((((((((.((..........-.)).)))))))))).).)))))))))).....).)))))))))...))...----- ( -33.60, z-score = -4.57, R) >droMoj3.scaffold_6496 16285329 105 - 26866924 UGAUGGAAAAGGACACGGACGACAUCUUUACCUGACAGUAUUAGAGUUAUUUAAAACA-GGAAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUGUCCUAGCAAAU--------- .........(((((((.....(((((((.(((.(.((((((((((.............-....)))))))))).).)))))))))).....).)))))).......--------- ( -31.43, z-score = -4.02, R) >droVir3.scaffold_12875 9939091 105 + 20611582 UGAUGUAUAAGGACACGGACGACAUCUUUACCUGACAGUAUUAGAAUUAUUUAAAUCA-AGAAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUGUCCUUGCAAAU--------- ...((((..(((((((.....(((((((.(((.(.((((((((((.((..........-.)).)))))))))).).)))))))))).....).))))))))))...--------- ( -32.80, z-score = -4.90, R) >triCas2.ChLG9 7640708 95 + 15222296 UGGUGGAUUGGAGCCCGGAGGACAUCUUUACCUGACAGUAUUAGAAGUAU----AAAAUGUAAUCUAAUGCUGCCCGGUAAGAUGUUAACAGUACUCCU---------------- .........((((..(....((((((((.(((.(.((((((((((..(((----.....))).)))))))))).).)))))))))))....)..)))).---------------- ( -32.50, z-score = -3.68, R) >anoGam1.chr3L 38263618 95 - 41284009 -GAUGAGCAUGGGCUCGGACGACAUCUUUACCUGACAGUAUUAGAAAUAUC--CGAUAACAUAUCUAAUGCUGCCCGGUAAGAUGUGAACAGACACCC----------------- -..(((((....)))))....(((((((.(((.(.((((((((((......--..........)))))))))).).))))))))))............----------------- ( -28.09, z-score = -3.32, R) >consensus UGAUGGAUAAGGACACGGACGACAUCUUUACCUGACAGUAUUAGAGUUAU____AAAA_AGGAUCUAAUGCUGCCCGGUAAGAUGUGAACAGAUGUCCUUGCAAAU_________ ........((((((.......(((((((.(((.(.((((((((((..................)))))))))).).))))))))))........))))))............... (-28.03 = -27.92 + -0.11)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:28:12 2011