
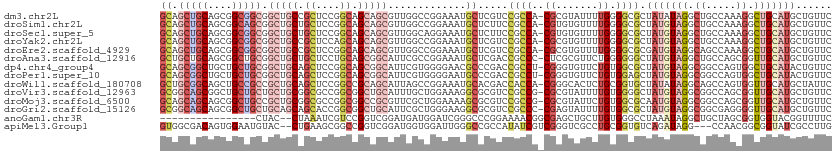
| Sequence ID | dm3.chr2L |
|---|---|
| Location | 2,962,727 – 2,962,838 |
| Length | 111 |
| Max. P | 0.669140 |

| Location | 2,962,727 – 2,962,838 |
|---|---|
| Length | 111 |
| Sequences | 14 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 71.79 |
| Shannon entropy | 0.66981 |
| G+C content | 0.65979 |
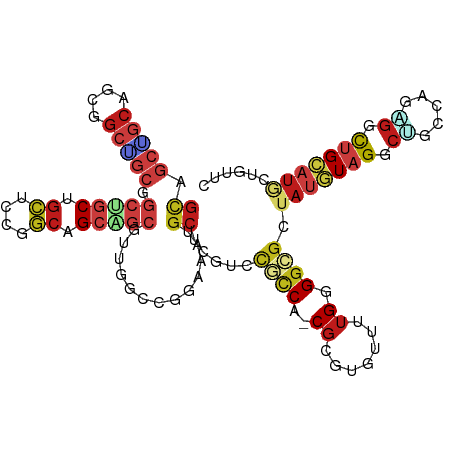
| Mean single sequence MFE | -54.99 |
| Consensus MFE | -21.55 |
| Energy contribution | -22.78 |
| Covariance contribution | 1.23 |
| Combinations/Pair | 1.63 |
| Mean z-score | -1.22 |
| Structure conservation index | 0.39 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.38 |
| SVM RNA-class probability | 0.669140 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2L 2962727 111 - 23011544 GCAGCUGCAGCGGCGGCGGCUGCCGCUCCGGCAGCAGCGUUGGCCGGAAAUGCUCGUCCGCCA-CGCGUAUUUUGGGGCGCUAUAUAGGCUGCCAAAGGCUGCAUGCUGUUC (((((((((((((((((.(((((((...)))))))(((((...((.(((((((.(((.....)-)).))))))).)))))))......))))))....)))))).))))).. ( -59.20, z-score = -2.02, R) >droSim1.chr2L 2921400 111 - 22036055 GCAGCUGCAGCGGCAGCGGCUGCUGCUCCGGCAGCAGCGUUGGCCGGAAAUGCUCUUCCGCCA-CGUGUGUUUUGGGGCGCUAUGUAGGCUGCCAAAGGCUGCAUGCUGUUC (((((((((((((((((.((((((((....))))))))...(((.((((......)))))))(-((((((((....))))).))))..))))))....)))))).))))).. ( -59.40, z-score = -2.28, R) >droSec1.super_5 1118625 111 - 5866729 GCAGCUGCAGCGGCGGCGGCUGCUGCUCCGGCAGCAGCGUUGGCAGGAAAUGCUCUUCCGCCA-CGUGUGUUUUGGGGCGCUAUGUAGGCUGCCAAAGGCUGCAUGCUGUUC (((((((((((((((((.((((((((....))))))))...(((.((((......)))))))(-((((((((....))))).))))..))))))....)))))).))))).. ( -58.70, z-score = -2.07, R) >droYak2.chr2L 2956108 111 - 22324452 GCAGCUGCAGCGGCGGCGGCUGCCGCUCCAGCAGCAGCGUUGGCCGGAAAUGCUCGUCCGCCA-CGCGUGUUUUGGGGCGCUAUGUAGGCUGCCAAAGGCUGCAUGCUGUUC (((((((((((((((((.((.((.(((((((.((((((((.(((.(((........)))))))-))).)))))))))))))...))..))))))....)))))).))))).. ( -58.70, z-score = -1.44, R) >droEre2.scaffold_4929 3008451 111 - 26641161 GCAGCUGCAGCGGCGGCGGCUGCCGCUCCGGCAGCAGCGUUGGCCGGAAAUGCUCGUCCGCCA-CGCGUGUUUUGGGGCGCGAUGUAGGCAGCCAAAGGCUGCAUGCUGUUC ((((((((((((....)((((((((((((((.((((((((.(((.(((........)))))))-))).)))))))))))((...)).)))))))....)))))).))))).. ( -57.40, z-score = -0.75, R) >droAna3.scaffold_12916 789597 111 - 16180835 GCUGCUGCAGCGGCUGCGGCUGCUGCUCCUGCAGCGGCAUUCGCCGGAAAUGCUCGACCGCCC-CUCGCGUUCUGGGGGGCUAUGUAGGCUGCCAGCGGUUGCAUGCUGUUC ((....))((((((((((((((((((.((((((.((((....)))).............((((-((((.....))))))))..))))))..).))))))))))).)))))). ( -56.70, z-score = -1.40, R) >dp4.chr4_group4 2397844 111 + 6586962 GCAGCGGCUGCUGCUGCGGCUGCAGCUCCGGCAGCGGCAUUCGUGGGGAACGCCCGACCGCCU-CGGGUGUUCUGUGGCGCUAUGUAGGCGGCCAGUGGCUGCAUACUGUUC (((((.((((((((((.(((....))).))))))))))........(((((((((((.....)-)))))))))).(((((((.....))).))))...)))))......... ( -59.40, z-score = -1.67, R) >droPer1.super_10 205402 111 + 3432795 GCAGCGGCUGCUGCUGCGGCUGCAGCUCCGGCAGCGGCAUUCGUGGGGAAUGCCCGACCGCCU-CGGGUGUUCUGUGGAGCUAUGUAGGCGGCCAGUGGCUGCAUACUGUUC (((((.((((((((((.(((....))).)))))))(((((((.....))))))).(.((((((-((...((((....))))..)).)))))).)))).)))))......... ( -57.40, z-score = -1.58, R) >droWil1.scaffold_180708 7614009 111 + 12563649 GCUGCGGCAGCUGCCGCCGCUGCAGCUCCGGCCGCAGCAUUAGCCGGAAAUGCACGACCACCA-CGGGCACUCUGCGGUGCUAUAUAGGCAGCCAGUGGUUGCAUGCUAUUC ((((((((.((....)).))))))))((((((..........))))))...((((((((((..-...((.....))((((((.....)))).)).)))))))..)))..... ( -47.40, z-score = -0.18, R) >droVir3.scaffold_12963 19430485 111 + 20206255 GCGGCAGCGGCUGCUGCUGCUGCGGCGCCGGCGGCUGCAUUUGCUGGAAAGGCGCGUCCGCCG-CGCGUAUUUUGUGGGGCUAUGUAGGCGGCCAGCGGUUGCAUGCUGUUC (((((((((((((((((((((((((((((..((((.......))))....)))))....(((.-((((.....)))).)))..)))).)))).)))))))))).)))))).. ( -60.00, z-score = -0.90, R) >droMoj3.scaffold_6500 19997926 111 + 32352404 GCAGCAGCAGCGGCUGCCGCUGCGGCGCCGGCGGCCGCGUUCGCUGGAAAAGCGCGUCCGCCG-CGCGUAUUCUGUGGCGCAAUGUAGGCGGCCAGCGGUUGCAUGCUGUUC (((((((((((.(((((((((((.((((((((((.((((((.........)))))).))))).-.(((.....))))))))...))).)))).)))).))))).)))))).. ( -64.40, z-score = -1.44, R) >droGri2.scaffold_15126 2809494 111 - 8399593 GCGGCAGCAGCGGCUGCUGCAGCAGCACCGGCGGCUGCAUUCGCUGGGAAGGCGCGUCCGCCC-CGAGUAUUUUGUGGCGCUAUGUAGGCGGCGAGGGGUUGCAUGCUGUUC ((((((((....))))))))((((((((((((((......)))))))......(((...((((-(..........((.((((.....)))).)).)))))))).))))))). ( -51.70, z-score = 0.36, R) >anoGam1.chr3R 46854678 94 - 53272125 ----------------CUAC--CUAAAUCGUCCGGUCGGAUGAUGGAUCGGGCCCGGAAAACGGCGAGCUGCUUGUGGGCCUAAAUAGGCUGCUAGCGGUGGUACGGUUUUC ----------------.(((--(....((((((((((........))))))))(((.....))).))((((((.(..(.(((....))))..).))))))))))........ ( -34.40, z-score = -1.08, R) >apiMel3.Group1 8377382 107 + 25854376 GUGGCGACAGUGGAAUGUAC--CUGAAGCGGCCGGUCGGAUGGUGGAUUGGGCCGCCAUAUCGUCGGGUCGCCUGCGGUGUCAGAUAGG---CCAACGGCGGUAUCGCCUUG ..((((((((.(.......)--)))..((((((.(.((((((((((......))))))).))).).))))))((((.(((((.....))---)..)).))))..)))))... ( -45.00, z-score = -0.67, R) >consensus GCAGCUGCAGCGGCUGCGGCUGCUGCUCCGGCAGCAGCGUUGGCCGGAAAUGCUCGUCCGCCA_CGCGUGUUUUGGGGCGCUAUGUAGGCUGCCAGAGGCUGCAUGCUGUUC ((.(((((....))))).(((((.((....)).))))).............)).....((((..............)))).(((((((.((.....)).)))))))...... (-21.55 = -22.78 + 1.23)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 21:12:40 2011