| Sequence ID | dm3.chr2R |
|---|---|
| Location | 11,488,680 – 11,488,781 |
| Length | 101 |
| Max. P | 0.950382 |

| Location | 11,488,680 – 11,488,781 |
|---|---|
| Length | 101 |
| Sequences | 9 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 80.11 |
| Shannon entropy | 0.43352 |
| G+C content | 0.51064 |
| Mean single sequence MFE | -34.97 |
| Consensus MFE | -15.83 |
| Energy contribution | -16.19 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.61 |
| Structure conservation index | 0.45 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.56 |
| SVM RNA-class probability | 0.950382 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
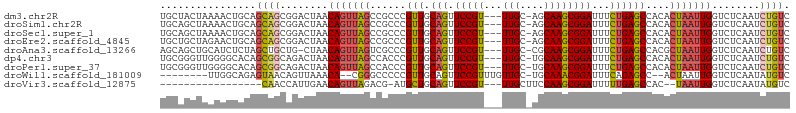
>dm3.chr2R 11488680 101 - 21146708 UGCUACUAAAACUGCAGCAGCGGACUAACAGUUAGCCGCCCGUUGCAGUUCCGU---UUGC-AGCAAGCGGAUUUCUGAGCCACACUAAUUGGUCUCAAUCUGUC (((......(((((((((.((((.(((.....)))))))..)))))))))....---..))-)....((((((...(((((((.......))).)))))))))). ( -35.20, z-score = -2.74, R) >droSim1.chr2R 10217311 101 - 19596830 UGCAGCUAAAACUGCAGCAGCGGACUAACAGUUAGCCGCCCGUUGCAGUUCCGU---UUGC-AGCAAGCGGAUUUCUGAGCCACACUAAUUGGUCUCAAUCUGUC .((((.....((((((((.((((.(((.....)))))))..))))))))(((((---(((.-..))))))))....(((((((.......))).))))..)))). ( -37.10, z-score = -2.91, R) >droSec1.super_1 8979137 101 - 14215200 UGCAGCUAAAACUGCAGCAGCGGACUAACAGUUAGCCGCCCGUUGCAGUUCCGU---UUGC-AGCAAGCGGAUUUCUGAGCCACACUAAUUGGUCUCAAUCUGUC .((((.....((((((((.((((.(((.....)))))))..))))))))(((((---(((.-..))))))))....(((((((.......))).))))..)))). ( -37.10, z-score = -2.91, R) >droEre2.scaffold_4845 8211052 101 + 22589142 UGCUGCUAGAACUGCAGCAGCGGACUAACAGUUAGCCGCCCGUUGCAGUUCCGU---UUGC-AGCAAGCGGAUUUCUGAGCCACACUAAUUGGUCUCAAUCUGUC ((((((..((((((((((.((((.(((.....)))))))..))))))))))...---..))-)))).((((((...(((((((.......))).)))))))))). ( -44.90, z-score = -4.79, R) >droAna3.scaffold_13266 7178017 100 + 19884421 AGCAGCUGCAUCUCUAGCUGCUG-CUAACAGUUAGUCGCCCGUUGCAGUUCCGU---UUGC-CGCAAGCGGAUUUCUGAGCCACGCUAAUUGGUCUCAAUCUGUC ((((((.((.......)).))))-))..(((((((.((...(((.(((.(((((---(((.-..))))))))...))))))..)))))))))............. ( -34.20, z-score = -2.10, R) >dp4.chr3 80184 101 + 19779522 UGCGGGUUGGGGCACAGCGGCAGACUAACAGUUAGCCACCCGUUGCAGUUCCGU---UUGC-UGCAAGCGGAUUUCUGAGCCACACUAAUUGGUCUCAAUCUGUC .((((((((((((.(((.(((.(((.....))).)))..((((((((((.....---..))-))).)))))..................))))))))))))))). ( -37.90, z-score = -1.99, R) >droPer1.super_37 59976 101 + 760358 UGCGGGUUGGGGCACAGCGGCAGACUAACAGUUAGCCACCCGUUGCAGUUCCGU---UUGC-UGCAAGCGGAUUUCUGAGCCACACUAAUUGGUCUCAAUCUGUC .((((((((((((.(((.(((.(((.....))).)))..((((((((((.....---..))-))).)))))..................))))))))))))))). ( -37.90, z-score = -1.99, R) >droWil1.scaffold_181009 1956400 92 + 3585778 --------UUGGCAGAGUAACAGUUAAACA--CGGGCCCCCGUUGCAGUUCCGUUUGUUGC-UGCAAACGGAUUUCAGAGCC--ACUAAUUGGUCUCAAUAUGUC --------..(((((((((((.((.....)--)((....)))))))...(((((((((...-.)))))))))..)).(((((--(.....))).)))....)))) ( -27.60, z-score = -1.81, R) >droVir3.scaffold_12875 14092298 82 + 20611582 -----------------CAACCAUUGAACAGUUAGACG-AUGCUGCAGUUCCGU---UUGCUUCCAAGCGGAUUUUUGAGCCAC--UAAUUGGUCUCAAUAUGUC -----------------.....(((((.(((((((...-..(((.(((.(((((---(((....))))))))...))))))..)--))))))...)))))..... ( -22.80, z-score = -2.25, R) >consensus UGCAGCUAAAACUGCAGCAGCGGACUAACAGUUAGCCGCCCGUUGCAGUUCCGU___UUGC_AGCAAGCGGAUUUCUGAGCCACACUAAUUGGUCUCAAUCUGUC ................((((........(((((((......(((.(((.(((((...(((....))))))))...))))))....)))))))........)))). (-15.83 = -16.19 + 0.36)

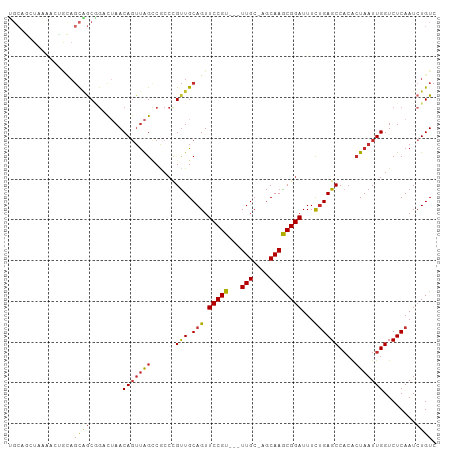
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:25:04 2011