| Sequence ID | dm3.chr2R |
|---|---|
| Location | 10,327,658 – 10,327,801 |
| Length | 143 |
| Max. P | 0.989206 |

| Location | 10,327,658 – 10,327,801 |
|---|---|
| Length | 143 |
| Sequences | 7 |
| Columns | 145 |
| Reading direction | reverse |
| Mean pairwise identity | 88.72 |
| Shannon entropy | 0.23708 |
| G+C content | 0.48393 |
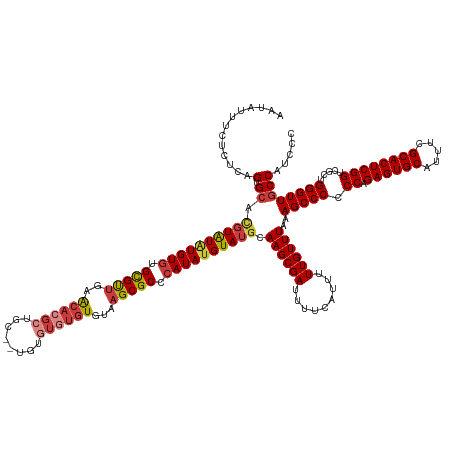
| Mean single sequence MFE | -44.99 |
| Consensus MFE | -38.48 |
| Energy contribution | -39.57 |
| Covariance contribution | 1.09 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.57 |
| Structure conservation index | 0.86 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.36 |
| SVM RNA-class probability | 0.989206 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
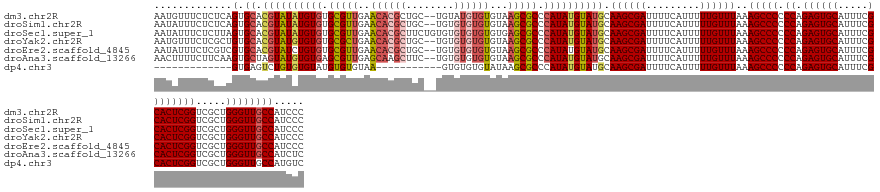
>dm3.chr2R 10327658 143 - 21146708 AAUGUUUCUCUCAGUGCACGUAUAUGUGUGCGUUGAACACGCUGC--UGUAUGUGUGUAAGCGCCCAUAUGUAUGCAAGCGAUUUUCAUUUUUGUUUAAAGCCCCCCAGAGUGCAUUUCGCACUCGGUCGCUGGGUUGCCAUCCC ...((....((((((((.((((((((((.(((((..((((((...--.....)))))).))))).)))))))))).((((((.........))))))...))...((.((((((.....))))))))..))))))..))...... ( -47.20, z-score = -2.98, R) >droSim1.chr2R 9092306 143 - 19596830 AAUAUUUCUCUCAGUGCACGUAUAUGUGUGCGUUGAACACGCUGC--UGUGUGUGUGUAAGCGCCCAUAUGUAUGCAAGCGAUUUUCAUUUUUGUUUAAAGCCCCCCAGAGUGCAUUUCGCACUCGGUCGCUGGGUUGCCAUCCC .........((((((((.((((((((((.(((((..((((((...--...))))))...))))).)))))))))).((((((.........))))))...))...((.((((((.....))))))))..)))))).......... ( -48.10, z-score = -3.26, R) >droSec1.super_1 7849298 145 - 14215200 AAUAUUUCUCUUAGUGCACGUAUAUGUGUGCGUUGAACACGCUUCUGUGUGUGUGUGUGAGCGCCCAUAUGUAUGCAAGCGAUUUUCAUUUUUGUUUAAAGCCCCCCAGAGUGCAUUUCGCACUCGGUCGCUGGGUUGCCAUCCC .............(.((.((((((((((.(((((.(((((((........)))))).).))))).)))))))))).((((((.........))))))..(((((.((.((((((.....)))))))).....))))))))..... ( -45.30, z-score = -2.58, R) >droYak2.chr2R 10261939 143 - 21139217 AAUGUUUCUCGCUGUGCACGUAUGUGUGUGCGCUGAACACGCUGC--UGUGUGUGUGUAAGCGCCCAUAUGUAUGCAAGCGAUUUUCAUUUUUGUUUAAAGCCCCCCAGAGUGCAUUUCGCACUCGGUCGCUGGGUUGCCAUCCC ((((....(((((.((((..((((((.(.(((((..((((((...--...))))))...))))))))))))..)))))))))....)))).........(((..((..((((((.....))))))))..)))(((.......))) ( -55.20, z-score = -4.15, R) >droEre2.scaffold_4845 7071655 143 + 22589142 AAUAUUUCUCGUCGUGCACGUAUCUGUGUGCGUUGAACACGCUGC--UGUGUGUGUGUAAGCGCCCAUAUGUAUGCAAGCGAUUUUCAUUUUUGUUUAAAGCCCCCCAGAGUGCAUUUCGCACUCGGUCGCUGGGUUGCCAUCCC ..........(((((...(((((.((((.(((((..((((((...--...))))))...))))).)))).)))))...)))))................(((..((..((((((.....))))))))..)))(((.......))) ( -45.70, z-score = -2.39, R) >droAna3.scaffold_13266 14667157 143 - 19884421 AACUUUUCUUCAAGUGCUAGUAUGUGUGAGCGUUGAGCAAGCUUC--UGUGUGUGUGUAAGCGCCCAUAUGUAUGCAAGCGAUUUUCAUUUUUGUUUAAAGCCCCCCAGAGUGCAUUUCGCACUCGGUCGCUGGGUUGCCAUCUC .((((......))))(((.(((((((((.(((((..(((.((...--...)).)))...))))).)))))))))...)))...................(((((.((.((((((.....)))))))).....)))))........ ( -39.60, z-score = -0.97, R) >dp4.chr3 15424245 121 + 19779522 -------------GUGAGUCUGUGUGUAUGUGUGUAA-----------GUGUGUGUAUAAGCGCCCAUAUGUAUGCAAGCGAUUUUCAUUUUUGUUUAAAGCCCCCCAGAGUGCAUUUCGCACUCGGUCGCUGGGUUGCCAUGUC -------------(.(((((.((.((((((..(((..-----------((((........))))..)))..)))))).))))))).)............(((((.((.((((((.....)))))))).....)))))........ ( -33.80, z-score = -1.66, R) >consensus AAUAUUUCUCUCAGUGCACGUAUAUGUGUGCGUUGAACACGCUGC__UGUGUGUGUGUAAGCGCCCAUAUGUAUGCAAGCGAUUUUCAUUUUUGUUUAAAGCCCCCCAGAGUGCAUUUCGCACUCGGUCGCUGGGUUGCCAUCCC .............(.((.((((((((((.(((((..((((((........))))))...))))).)))))))))).((((((.........))))))..(((((.((.((((((.....)))))))).....))))))))..... (-38.48 = -39.57 + 1.09)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:22:33 2011