
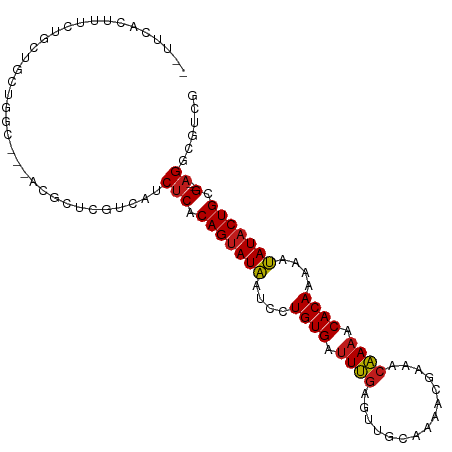
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 10,103,276 – 10,103,375 |
| Length | 99 |
| Max. P | 0.999945 |

| Location | 10,103,276 – 10,103,375 |
|---|---|
| Length | 99 |
| Sequences | 13 |
| Columns | 106 |
| Reading direction | forward |
| Mean pairwise identity | 72.37 |
| Shannon entropy | 0.64092 |
| G+C content | 0.41251 |
| Mean single sequence MFE | -24.27 |
| Consensus MFE | -10.37 |
| Energy contribution | -10.32 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.43 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.28 |
| SVM RNA-class probability | 0.987597 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 10103276 99 + 21146708 --GUCGCUUUUCGCUGCUGGC---ACGCUGGUCAUCUCACAGUAUAAUCCUGUGAUUUGGAUUGCAAAACGAAACAAAACACAAAAAUAUACUGCG--AGGCGUCG --(.(((........((..(.---...)..))...(((.(((((((....((((.((((..(((.....)))..)))).))))....))))))).)--))))).). ( -28.10, z-score = -2.28, R) >droSec1.super_1 7611672 99 + 14215200 --UUCGCUUUUUGCUGCUGGC---ACGCUGGUCAUCUCACAGUAUAAUCCUGUGAUUUGAGUUGCAAAACGAAACAAAACACAAAAAUAUACUGCG--AGGCGUCG --..(((........((..(.---...)..))...(((.(((((((....((((.((((..(((.....)))..)))).))))....))))))).)--)))))... ( -25.60, z-score = -1.43, R) >droSim1.chr2R 8866640 99 + 19596830 --UUCGCUUUCUGCUGCUGGC---ACGCUGGUCAUCUCACAGUAUAAUCCUGUGAUUUGAGUUGCAAAACGAAACAAAACACAAAAAUAUACUGCG--AGGCGUCG --..(((........((..(.---...)..))...(((.(((((((....((((.((((..(((.....)))..)))).))))....))))))).)--)))))... ( -25.60, z-score = -1.52, R) >droYak2.chr2R 10041116 99 + 21139217 --GUCACUUUCUGCUGCUGGC---ACGCUGGUCAUCUCACAGUAUAAUCCUGUGAUUUGAGUUGCAAAAGGAAACAAAACACAAAAAUAUACUGCG--AGGCGUCG --..........((.((..(.---...)..))...(((.(((((((....((((.((((.....)))).(....)....))))....))))))).)--)))).... ( -23.00, z-score = -0.57, R) >droEre2.scaffold_4845 6849797 102 - 22589142 --GUCACUUUCUGCUGCUGGCGGCACGCUGGUCAUCUCACAGUAUAAUCCUGUGAUUUGAUUUGCAAAACGAAACAAAACACAAAAAUAUACUGCG--AGGCGUCG --..........((.((..(((...)))..))...(((.(((((((....((((.((((.((((.....)))).)))).))))....))))))).)--)))).... ( -28.20, z-score = -2.26, R) >droAna3.scaffold_13266 14064359 100 - 19884421 UGCACUGUUUCAGCUGCUGAC---ACGCUUGGCAUCUCACAGUAUAAUCCUGUGAUUUGAGUUUCAAA-CCAAACAAAACACAAAAAUAUACUGCG--AGGCGACG .((((((...))).)))....---.(((((.(((..((((((.......))))))((((.((((....-.......)))).)))).......))).--)))))... ( -23.70, z-score = -1.48, R) >dp4.chr3 16336980 94 + 19779522 ------CACUCUCUUGAUAGC---ACGCCCAGCAUCUCACAGUAUAAUCCUGUGAUUUGCGUUUCAGA-CACAACAAAACACAAAAAUAUACUGCG--AGGCGUCA ------.............(.---(((((..(((..((((((.......))))))((((.((((....-.......)))).)))).......))).--.)))))). ( -21.40, z-score = -2.17, R) >droPer1.super_4 6736411 93 - 7162766 ------CACUCUCU-GAUAGC---ACGCCCAGCAUCUCACAGUAUAAUCCUGUGAUUUGCGUUUCAGA-CACAACAAAACACAAAAAUAUACUGCG--AGGCGUCA ------........-....(.---(((((..(((..((((((.......))))))((((.((((....-.......)))).)))).......))).--.)))))). ( -21.40, z-score = -2.24, R) >droWil1.scaffold_180700 4769494 103 - 6630534 -UUUUAAAAUGGUUGCGUGGAUUUCCAUUUCGCAGCUCACAGUAUAAUCCUGUGAUUUGAGUUGACAAAUUGAACAAAACACAAAAAUAUACUGCG--AGACGCAA -...........((((((((....)))((((((((.((((((.......))))))((((.(((..............))).))))......)))))--)))))))) ( -26.04, z-score = -2.41, R) >droVir3.scaffold_12875 3006184 98 - 20611582 --AUGGAUU---UUUGUUGUUGCUACAGCCGAUAACUCACAGUAUAAUCCUGUGAUUUGAUUUUCAAAA-UAAACAAAACACAAAAGUAUACUGCG--AGACAUCG --.......---...(((((....)))))((((..(((.(((((((....((((.((((.(((......-.))))))).))))....))))))).)--))..)))) ( -24.20, z-score = -3.33, R) >droMoj3.scaffold_6496 15637758 101 - 26866924 --UUUAAUUCAAUUUGUUGUUGUUUCAGCCGAUAACUCACAGUAUAAUCCUGUGAUUUGAUUUUUAUAA-UAAACAAAACACAAAAAUAUACUGCG--AGACAUCG --.............((((......))))((((..(((.(((((((....((((.((((.(((......-.))))))).))))....))))))).)--))..)))) ( -21.80, z-score = -3.39, R) >droGri2.scaffold_15245 14076217 102 + 18325388 --UUAAAAUUUGGUUGUCCUUGCUGCUGGCGAUAACUCACAGUAUAAUCCUGUGAUUUGAUUUUCAAAAAUAAACAAAACACAAAAAUAUACUGCG--AGACAUCG --.......(..((.((....)).))..)((((..(((.(((((((....((((.((((.(((........))))))).))))....))))))).)--))..)))) ( -21.90, z-score = -2.79, R) >triCas2.ChLG1X 7039779 96 + 8109244 -----ACUUGCCUGUGGUGACAAAACCAACUCC-CCGCACAGAAUUUUCCUGUGAUUGAAUC----AAGCACACCCAUUCACAAGCAAAUCCUGCGCAGGGCGCCA -----....((((.((((......)))).....-..((.(((.((((.(.(((((.((....----.........)).))))).).)))).))).)).)))).... ( -24.52, z-score = -1.26, R) >consensus __UUCACUUUCUGCUGCUGGC___ACGCUCGUCAUCUCACAGUAUAAUCCUGUGAUUUGAGUUGCAAAACGAAACAAAACACAAAAAUAUACUGCG__AGGCGUCG .......................................(((((((....((((.((((...............)))).))))....)))))))............ (-10.37 = -10.32 + -0.05)

| Location | 10,103,276 – 10,103,375 |
|---|---|
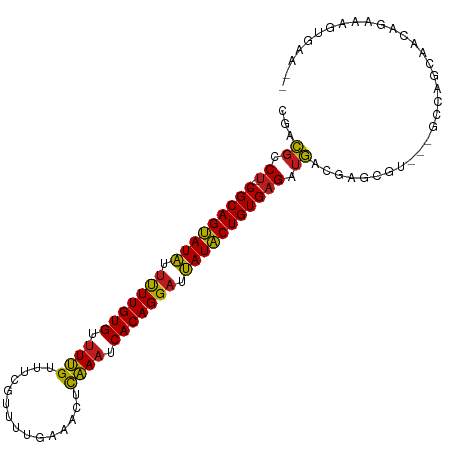
| Length | 99 |
| Sequences | 13 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 72.37 |
| Shannon entropy | 0.64092 |
| G+C content | 0.41251 |
| Mean single sequence MFE | -32.66 |
| Consensus MFE | -20.32 |
| Energy contribution | -19.91 |
| Covariance contribution | -0.41 |
| Combinations/Pair | 1.25 |
| Mean z-score | -3.64 |
| Structure conservation index | 0.62 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 5.10 |
| SVM RNA-class probability | 0.999945 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 10103276 99 - 21146708 CGACGCCU--CGCAGUAUAUUUUUGUGUUUUGUUUCGUUUUGCAAUCCAAAUCACAGGAUUAUACUGUGAGAUGACCAGCGU---GCCAGCAGCGAAAAGCGAC-- ...(((((--(((((((((.(((((((.((((..(.(.....).)..)))).))))))).))))))))))).......((.(---(....)))).....)))..-- ( -32.40, z-score = -3.28, R) >droSec1.super_1 7611672 99 - 14215200 CGACGCCU--CGCAGUAUAUUUUUGUGUUUUGUUUCGUUUUGCAACUCAAAUCACAGGAUUAUACUGUGAGAUGACCAGCGU---GCCAGCAGCAAAAAGCGAA-- ...(((((--(((((((((.(((((((.((((....(((....))).)))).))))))).))))))))))).......((.(---(....)))).....)))..-- ( -31.70, z-score = -2.94, R) >droSim1.chr2R 8866640 99 - 19596830 CGACGCCU--CGCAGUAUAUUUUUGUGUUUUGUUUCGUUUUGCAACUCAAAUCACAGGAUUAUACUGUGAGAUGACCAGCGU---GCCAGCAGCAGAAAGCGAA-- ...(((((--(((((((((.(((((((.((((....(((....))).)))).))))))).))))))))))).......((.(---(....)))).....)))..-- ( -31.70, z-score = -2.77, R) >droYak2.chr2R 10041116 99 - 21139217 CGACGCCU--CGCAGUAUAUUUUUGUGUUUUGUUUCCUUUUGCAACUCAAAUCACAGGAUUAUACUGUGAGAUGACCAGCGU---GCCAGCAGCAGAAAGUGAC-- ...(((((--(((((((((.(((((((.((((...............)))).))))))).))))))))))).......((.(---(....)))).....)))..-- ( -29.36, z-score = -2.48, R) >droEre2.scaffold_4845 6849797 102 + 22589142 CGACGCCU--CGCAGUAUAUUUUUGUGUUUUGUUUCGUUUUGCAAAUCAAAUCACAGGAUUAUACUGUGAGAUGACCAGCGUGCCGCCAGCAGCAGAAAGUGAC-- ...(((((--(((((((((.(((((((.((((.((.(.....).)).)))).))))))).))))))))))).......((.(((.....))))).....)))..-- ( -32.20, z-score = -2.89, R) >droAna3.scaffold_13266 14064359 100 + 19884421 CGUCGCCU--CGCAGUAUAUUUUUGUGUUUUGUUUGG-UUUGAAACUCAAAUCACAGGAUUAUACUGUGAGAUGCCAAGCGU---GUCAGCAGCUGAAACAGUGCA .(.(((((--(((((((((.(((((((.((((...(.-.......).)))).))))))).)))))))))))..((...))))---).).(((.(((...)))))). ( -32.70, z-score = -2.59, R) >dp4.chr3 16336980 94 - 19779522 UGACGCCU--CGCAGUAUAUUUUUGUGUUUUGUUGUG-UCUGAAACGCAAAUCACAGGAUUAUACUGUGAGAUGCUGGGCGU---GCUAUCAAGAGAGUG------ ..((((((--(((((((((.(((((((.....(((((-(.....))))))..))))))).)))))))))).......)))))---...............------ ( -31.91, z-score = -2.89, R) >droPer1.super_4 6736411 93 + 7162766 UGACGCCU--CGCAGUAUAUUUUUGUGUUUUGUUGUG-UCUGAAACGCAAAUCACAGGAUUAUACUGUGAGAUGCUGGGCGU---GCUAUC-AGAGAGUG------ ..((((((--(((((((((.(((((((.....(((((-(.....))))))..))))))).)))))))))).......)))))---......-........------ ( -31.91, z-score = -2.85, R) >droWil1.scaffold_180700 4769494 103 + 6630534 UUGCGUCU--CGCAGUAUAUUUUUGUGUUUUGUUCAAUUUGUCAACUCAAAUCACAGGAUUAUACUGUGAGCUGCGAAAUGGAAAUCCACGCAACCAUUUUAAAA- ..(((.((--(((((((((.(((((((.((((...............)))).))))))).))))))))))).)))(((((((............)))))))....- ( -33.46, z-score = -4.94, R) >droVir3.scaffold_12875 3006184 98 + 20611582 CGAUGUCU--CGCAGUAUACUUUUGUGUUUUGUUUA-UUUUGAAAAUCAAAUCACAGGAUUAUACUGUGAGUUAUCGGCUGUAGCAACAACAAA---AAUCCAU-- (((((.((--(((((((((.(((((((.(((((((.-......))).)))).))))))).))))))))))).)))))((....)).........---.......-- ( -31.60, z-score = -5.16, R) >droMoj3.scaffold_6496 15637758 101 + 26866924 CGAUGUCU--CGCAGUAUAUUUUUGUGUUUUGUUUA-UUAUAAAAAUCAAAUCACAGGAUUAUACUGUGAGUUAUCGGCUGAAACAACAACAAAUUGAAUUAAA-- (((((.((--(((((((((.(((((((.(((((((.-......))).)))).))))))).))))))))))).)))))...........................-- ( -30.90, z-score = -5.41, R) >droGri2.scaffold_15245 14076217 102 - 18325388 CGAUGUCU--CGCAGUAUAUUUUUGUGUUUUGUUUAUUUUUGAAAAUCAAAUCACAGGAUUAUACUGUGAGUUAUCGCCAGCAGCAAGGACAACCAAAUUUUAA-- (((((.((--(((((((((.(((((((.(((((((........))).)))).))))))).))))))))))).)))))..........((....)).........-- ( -31.00, z-score = -4.75, R) >triCas2.ChLG1X 7039779 96 - 8109244 UGGCGCCCUGCGCAGGAUUUGCUUGUGAAUGGGUGUGCUU----GAUUCAAUCACAGGAAAAUUCUGUGCGG-GGAGUUGGUUUUGUCACCACAGGCAAGU----- .(((.(((((((((((((((.(((((((.(((((......----.))))).))))))).)))))))))))))-)).)))...((((((......)))))).----- ( -43.80, z-score = -4.41, R) >consensus CGACGCCU__CGCAGUAUAUUUUUGUGUUUUGUUUCGUUUUGAAACUCAAAUCACAGGAUUAUACUGUGAGAUGACGAGCGU___GCCAGCAACAGAAAGUGAA__ ..........(((((((((.(((((((.((((...............)))).))))))).)))))))))..................................... (-20.32 = -19.91 + -0.41)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:22:06 2011