
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 9,842,173 – 9,842,313 |
| Length | 140 |
| Max. P | 0.999138 |

| Location | 9,842,173 – 9,842,313 |
|---|---|
| Length | 140 |
| Sequences | 12 |
| Columns | 150 |
| Reading direction | forward |
| Mean pairwise identity | 76.32 |
| Shannon entropy | 0.49083 |
| G+C content | 0.58364 |
| Mean single sequence MFE | -61.65 |
| Consensus MFE | -23.94 |
| Energy contribution | -23.12 |
| Covariance contribution | -0.81 |
| Combinations/Pair | 1.50 |
| Mean z-score | -3.38 |
| Structure conservation index | 0.39 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.83 |
| SVM RNA-class probability | 0.995648 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 9842173 140 + 21146708 -AAUGCGUGCCACACUCACCGUAGACUUUCCCAAUGAUGCCCACGGUGGAGGUCAUCUGGAGAUCCGGA---AAAGUCAGCGGCAG------CGUGUGCGGAUGGAGCUGAGCUGGCCCACCGGGAGCCAGCUCACGCUCCGUGGACAUU -.....(((((((.....(((((((((((.((...((((.((........)).)))).((....)))).---)))))).((....)------)...)))))..((((((((((((((((....)).))))))))).))))))))).))). ( -64.40, z-score = -2.97, R) >droSim1.chr2R 8276791 140 + 19596830 -AAUGCGUGCCACACUCACCGUAGACUUUCCCAAUGAUGCCCACGGUGGAGGUCAUCUGGAGAUCCGGA---AAAGUCAGCGGCAG------CGUGUGCGGAUGGAGCUGAGCUGGCCCACCGGGAGCCAGCUCACGCUCCGUGGACAUU -.....(((((((.....(((((((((((.((...((((.((........)).)))).((....)))).---)))))).((....)------)...)))))..((((((((((((((((....)).))))))))).))))))))).))). ( -64.40, z-score = -2.97, R) >droSec1.super_239 12514 140 + 19797 -AAUGCGUGCCACACUCACCGUAGACUUUCCCAAUGAUGCCCACGGUGGAGGUCAUCUGGAGAUCCGGA---AAAGUCAGCGGCAG------CGUGUGCGGAUGGAGCUGAGCUGGCCCACCGGGAGCCAGCUCACGCUCCGUGGACAUU -.....(((((((.....(((((((((((.((...((((.((........)).)))).((....)))).---)))))).((....)------)...)))))..((((((((((((((((....)).))))))))).))))))))).))). ( -64.40, z-score = -2.97, R) >droYak2.chr2R 14027328 140 - 21139217 -AUUGUGUGCCACACUCACCGUAGACUUUCCCAAUGAUGCCCACGGCGGAGGUCAUCUGGAGAUCCGGA---AAAGUCAGCGGCAG------CGUGUGCGGAUGGAGCUGAGCUGGCCCACCAGGAGCCAGCUCACGCUCCGUGGACAUU -...((((.((((.....(((((((((((.((......(((...)))(((..((.....))..))))).---)))))).((....)------)...)))))..((((((((((((((((....)).))))))))).))))))))))))). ( -66.30, z-score = -3.73, R) >droEre2.scaffold_4845 6609172 140 - 22589142 -AUUGGGUGCCACACUCACCGUAGACUUUCCCAAUGAUGCCCACGGCGGAGGUCAUCUGGAGAUCCGGA---AAAGUCAGCGGCAA------CGUGUGCGGAUGGAGCUGAGCUGGCCCACCAGGAGCCAGCUCACGCUCCGUGGACAUU -....((((((((.....(((((((((((.((......(((...)))(((..((.....))..))))).---)))))).(((....------))).)))))..((((((((((((((((....)).))))))))).))))))))).)))) ( -68.50, z-score = -4.33, R) >droAna3.scaffold_13266 17152529 139 + 19884421 -GAUGUGUGUGA-ACUCACCGUAGACCUUUCCAAUGAUGCCCACCGCGGAGGUGAUCUGCAGGUCCGGG---AACGUCAAUGGCAA------CGUGUGCGGGUGGAGCUGAGCUGGCCCCCCGGGAGCCAGCUCGCGCUCCGUAGACAUC -(((((.((...-.....(((((.((.((.(((.((((((((((((((((.....))))).)))..)))---..))))).))).))------.)).)))))..((((((((((((((((....)).))))))))).))))).)).))))) ( -69.10, z-score = -3.99, R) >dp4.chr3 8155542 135 + 19779522 -----AAUGAAA-ACUUACCGUAGACCUUUCCAAUGAUGCCCACGGUGGUGUUGAUCUGGAGAUCGGGA---AAUGUCAACGGCAG------CGUGUGCGUCAGGAGCUGAGCUGGCCCACCAGAAGCCAGCUCGCGCUCCGUUGACAUU -----.......-.......((.(((........((((((.((((.((.(((((((((.(....).)))---....)))))).)).------)))).))))))((((((((((((((.........))))))))).)))))))).))... ( -56.70, z-score = -3.68, R) >droPer1.super_4 2033042 135 + 7162766 -----AAUGAAA-ACUUACCGUAGACCUUUCCAAUGAUGCCCACGGUGGUGUUGAUCUGGAGAUCGGGA---AAUGUCAACGGCAG------CGUGUGCGUCAGGAGCUGAGCUGGCCCGCCAGGAGCCAGCUCGCGCUCCGUUGACAUU -----.......-.......((.(((........((((((.((((.((.(((((((((.(....).)))---....)))))).)).------)))).))))))((((((((((((((((....)).))))))))).)))))))).))... ( -61.50, z-score = -4.35, R) >droWil1.scaffold_181009 1336300 140 + 3585778 -UACACUCAUUUUACUUACCAUACACUUUGCCAAUUAUGCCCACGGUAGAGCUCAGCUGGAGAUCGGGA---AAUGUCAACGGCAG------CGUAUGCGUAUGGAGCUGAGCUGGCCCACCAGGUGCCAGCUCCCGCUCCGUUGACAUU -....(((.................(((((((............)))))))(((.....)))...))).---(((((((((.(((.------....)))....(((((.((((((((((....)).))))))))..)))))))))))))) ( -52.70, z-score = -3.19, R) >droVir3.scaffold_12875 14392570 146 - 20611582 AGUUCGGCUUGC-ACUUACCAUAAACCUUGCCCAUAAUGCCCACCGCAGAGUUCAGCUGCAGCGCAGUGCCCAAAUCGGGUGAAAAUGUCAACGGCAGCGUAUGGAGCUGAGCUGGCCCGCCGG---CCAGCUCCCGCUCCGUUGACAUU .((((((((.(.-((((..((.......)).......(((.....))))))).)))))).))).....((((.....))))...(((((((((((.((((...(((((((.(((((....))))---))))))))))))))))))))))) ( -60.70, z-score = -3.36, R) >droMoj3.scaffold_6496 23813169 137 + 26866924 ----CGUUCUGCCACUUACCAUAAACCUUGCCAAUAAUGCCCACCGCUGUGUUCAGUUGCAAUGAGGUGCCCAGAUCUGGGGGAAA------CGGUAGCGUAUGGAGCUGGGCUGGUCCGCCAG---CCAGCUCCCGCUCCGUUGACAUC ----.(((..........((....((((((.((((..((..(((....)))..))))))...)))))).((((....)))))).((------(((.((((...((((((((.((((....))))---))))))))))))))))))))... ( -51.40, z-score = -1.44, R) >droGri2.scaffold_15112 4064466 147 - 5172618 AGUAAGAGCUGAAACUUACCAUAAACCUUGCCAAUAAUGCCCACGGCUGUGUUCAGUUGCAGUGCUGUGCCCAAAUCGGGUGGAAAUGUCAACGGCAGCGUAUGGAGCUGAGCUGGGCCCCCCG---CCAGCUCCCGCUCCGUUGACAUC .(((((........)))))...........(((........((((((..(((......)))..))))))(((.....))))))..((((((((((.((((...(((((((.((.((....)).)---)))))))))))))))))))))). ( -59.70, z-score = -3.60, R) >consensus _AAUGCGUGCCACACUCACCGUAGACCUUCCCAAUGAUGCCCACGGCGGAGGUCAUCUGGAGAUCCGGA___AAAGUCAGCGGCAG______CGUGUGCGGAUGGAGCUGAGCUGGCCCACCAGGAGCCAGCUCACGCUCCGUUGACAUU ..................((((................((.....))........((....))................))))..................(((((((.(((((((...........)))))))..)))))))....... (-23.94 = -23.12 + -0.81)

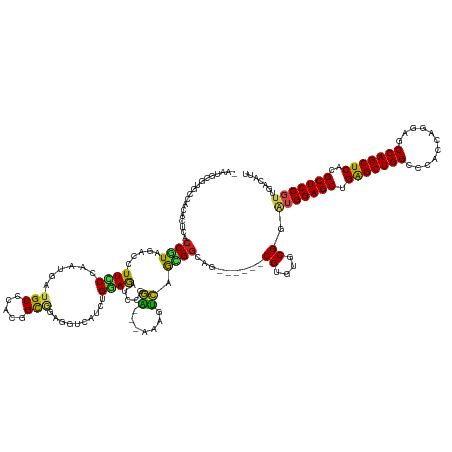
| Location | 9,842,173 – 9,842,313 |
|---|---|
| Length | 140 |
| Sequences | 12 |
| Columns | 150 |
| Reading direction | reverse |
| Mean pairwise identity | 76.32 |
| Shannon entropy | 0.49083 |
| G+C content | 0.58364 |
| Mean single sequence MFE | -64.16 |
| Consensus MFE | -28.41 |
| Energy contribution | -28.11 |
| Covariance contribution | -0.31 |
| Combinations/Pair | 1.19 |
| Mean z-score | -3.72 |
| Structure conservation index | 0.44 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.67 |
| SVM RNA-class probability | 0.999138 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 9842173 140 - 21146708 AAUGUCCACGGAGCGUGAGCUGGCUCCCGGUGGGCCAGCUCAGCUCCAUCCGCACACG------CUGCCGCUGACUUU---UCCGGAUCUCCAGAUGACCUCCACCGUGGGCAUCAUUGGGAAAGUCUACGGUGAGUGUGGCACGCAUU- (((((....(((((.(((((((((((.....))))))))))))))))....((.((((------((((((..((((((---(((((....)).((((.((........)).))))...)))))))))..)))).))))))))..)))))- ( -73.20, z-score = -5.26, R) >droSim1.chr2R 8276791 140 - 19596830 AAUGUCCACGGAGCGUGAGCUGGCUCCCGGUGGGCCAGCUCAGCUCCAUCCGCACACG------CUGCCGCUGACUUU---UCCGGAUCUCCAGAUGACCUCCACCGUGGGCAUCAUUGGGAAAGUCUACGGUGAGUGUGGCACGCAUU- (((((....(((((.(((((((((((.....))))))))))))))))....((.((((------((((((..((((((---(((((....)).((((.((........)).))))...)))))))))..)))).))))))))..)))))- ( -73.20, z-score = -5.26, R) >droSec1.super_239 12514 140 - 19797 AAUGUCCACGGAGCGUGAGCUGGCUCCCGGUGGGCCAGCUCAGCUCCAUCCGCACACG------CUGCCGCUGACUUU---UCCGGAUCUCCAGAUGACCUCCACCGUGGGCAUCAUUGGGAAAGUCUACGGUGAGUGUGGCACGCAUU- (((((....(((((.(((((((((((.....))))))))))))))))....((.((((------((((((..((((((---(((((....)).((((.((........)).))))...)))))))))..)))).))))))))..)))))- ( -73.20, z-score = -5.26, R) >droYak2.chr2R 14027328 140 + 21139217 AAUGUCCACGGAGCGUGAGCUGGCUCCUGGUGGGCCAGCUCAGCUCCAUCCGCACACG------CUGCCGCUGACUUU---UCCGGAUCUCCAGAUGACCUCCGCCGUGGGCAUCAUUGGGAAAGUCUACGGUGAGUGUGGCACACAAU- ..(((....(((((.(((((((((.((....))))))))))))))))....((.((((------((((((..((((((---(((((....)).((((.((........)).))))...)))))))))..)))).))))))))..)))..- ( -72.40, z-score = -5.14, R) >droEre2.scaffold_4845 6609172 140 + 22589142 AAUGUCCACGGAGCGUGAGCUGGCUCCUGGUGGGCCAGCUCAGCUCCAUCCGCACACG------UUGCCGCUGACUUU---UCCGGAUCUCCAGAUGACCUCCGCCGUGGGCAUCAUUGGGAAAGUCUACGGUGAGUGUGGCACCCAAU- ..(((....(((((.(((((((((.((....)))))))))))))))).....(((((.------(..(((..((((((---(((((....)).((((.((........)).))))...)))))))))..)))..)))))))))......- ( -69.90, z-score = -4.59, R) >droAna3.scaffold_13266 17152529 139 - 19884421 GAUGUCUACGGAGCGCGAGCUGGCUCCCGGGGGGCCAGCUCAGCUCCACCCGCACACG------UUGCCAUUGACGUU---CCCGGACCUGCAGAUCACCUCCGCGGUGGGCAUCAUUGGAAAGGUCUACGGUGAGU-UCACACACAUC- ((((((((((((((..(((((((((((...))))))))))).)))))...(((..(((------((......))))).---...(((..((.....))..)))))))))))))))..((((....))))..(((...-.))).......- ( -59.80, z-score = -2.15, R) >dp4.chr3 8155542 135 - 19779522 AAUGUCAACGGAGCGCGAGCUGGCUUCUGGUGGGCCAGCUCAGCUCCUGACGCACACG------CUGCCGUUGACAUU---UCCCGAUCUCCAGAUCAACACCACCGUGGGCAUCAUUGGAAAGGUCUACGGUAAGU-UUUCAUU----- (((((((((((((((.((((((((((.....)))))))))).((.......))...))------)).)))))))))))---....((((....))))......(((((((((.((....))...)))))))))....-.......----- ( -58.20, z-score = -4.38, R) >droPer1.super_4 2033042 135 - 7162766 AAUGUCAACGGAGCGCGAGCUGGCUCCUGGCGGGCCAGCUCAGCUCCUGACGCACACG------CUGCCGUUGACAUU---UCCCGAUCUCCAGAUCAACACCACCGUGGGCAUCAUUGGAAAGGUCUACGGUAAGU-UUUCAUU----- (((((((((((((((.((((((((((.....)))))))))).((.......))...))------)).)))))))))))---....((((....))))......(((((((((.((....))...)))))))))....-.......----- ( -60.10, z-score = -4.74, R) >droWil1.scaffold_181009 1336300 140 - 3585778 AAUGUCAACGGAGCGGGAGCUGGCACCUGGUGGGCCAGCUCAGCUCCAUACGCAUACG------CUGCCGUUGACAUU---UCCCGAUCUCCAGCUGAGCUCUACCGUGGGCAUAAUUGGCAAAGUGUAUGGUAAGUAAAAUGAGUGUA- ....(((..(((((..((((((((.((....)))))))))).))))).(((.((((((------(((((((((.....---..........))))...((((......))))......)))..))))))))))).......))).....- ( -55.36, z-score = -2.39, R) >droVir3.scaffold_12875 14392570 146 + 20611582 AAUGUCAACGGAGCGGGAGCUGG---CCGGCGGGCCAGCUCAGCUCCAUACGCUGCCGUUGACAUUUUCACCCGAUUUGGGCACUGCGCUGCAGCUGAACUCUGCGGUGGGCAUUAUGGGCAAGGUUUAUGGUAAGU-GCAAGCCGAACU (((((((((((((((((((((((---(.((....)).)).)))))))...)))).))))))))))).........((((((((((.((((((((.......))))))))...(((((((((...))))))))).)))-))...))))).. ( -64.60, z-score = -2.26, R) >droMoj3.scaffold_6496 23813169 137 - 26866924 GAUGUCAACGGAGCGGGAGCUGG---CUGGCGGACCAGCCCAGCUCCAUACGCUACCG------UUUCCCCCAGAUCUGGGCACCUCAUUGCAACUGAACACAGCGGUGGGCAUUAUUGGCAAGGUUUAUGGUAAGUGGCAGAACG---- ..(((((((((((((((((((((---((((....)))).))))))))...)))).)))------)...((..((((((..((..((((((((...........))))))))((....)))).))))))..))....))))).....---- ( -52.50, z-score = -1.27, R) >droGri2.scaffold_15112 4064466 147 + 5172618 GAUGUCAACGGAGCGGGAGCUGG---CGGGGGGCCCAGCUCAGCUCCAUACGCUGCCGUUGACAUUUCCACCCGAUUUGGGCACAGCACUGCAACUGAACACAGCCGUGGGCAUUAUUGGCAAGGUUUAUGGUAAGUUUCAGCUCUUACU (((((((((((((((((((((((---(.((....)).)).)))))))...)))).))))))))))).((((((.....)))....((.(..(..(((....)))..)..))).....)))..........((((((........)))))) ( -57.50, z-score = -1.97, R) >consensus AAUGUCAACGGAGCGUGAGCUGGCUCCUGGUGGGCCAGCUCAGCUCCAUACGCACACG______CUGCCGCUGACUUU___UCCGGAUCUCCAGAUGACCUCCACCGUGGGCAUCAUUGGGAAAGUCUACGGUAAGUGUGACACCCAUU_ .........(((((..(((((((...........))))))).))))).......................................................(((.((((((............)))))).)))................ (-28.41 = -28.11 + -0.31)

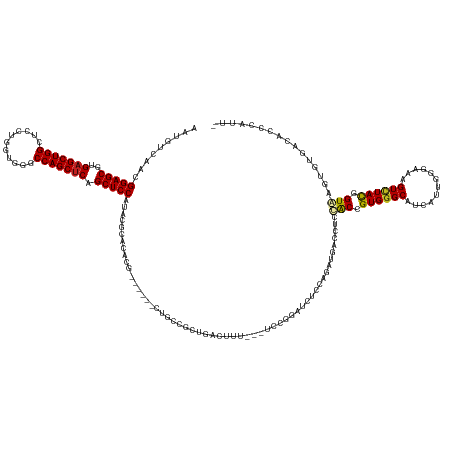
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:21:21 2011