| Sequence ID | dm3.chr2R |
|---|---|
| Location | 9,767,846 – 9,767,950 |
| Length | 104 |
| Max. P | 0.912230 |

| Location | 9,767,846 – 9,767,950 |
|---|---|
| Length | 104 |
| Sequences | 14 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 83.68 |
| Shannon entropy | 0.36304 |
| G+C content | 0.60874 |
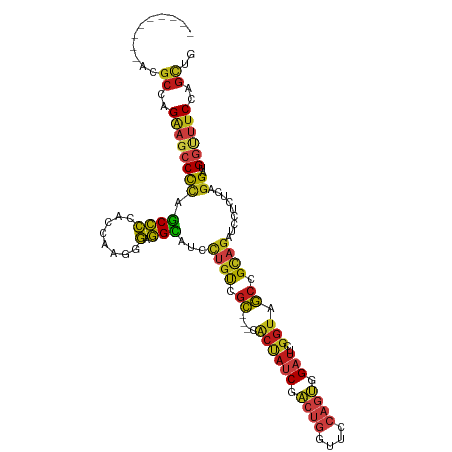
| Mean single sequence MFE | -41.73 |
| Consensus MFE | -25.08 |
| Energy contribution | -25.43 |
| Covariance contribution | 0.35 |
| Combinations/Pair | 1.52 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.60 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.22 |
| SVM RNA-class probability | 0.912230 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 9767846 104 - 21146708 ---------ACGCUAGAAGCCCCAGCCCCACCAAGGGAGGCAUCCUGUCGC---CACUAUCGACUGGUUCCAGUGGAUUCGGUAGCCGCAGAUCCUCCCAGGAUGGUUUCCAGCUG ---------..(((.((((((((.(......)..((((((.(((.(((.((---.((((((.((((....)))).)))..))).)).)))))))))))).))..)))))).))).. ( -41.80, z-score = -2.71, R) >droSim1.chr2R_random 2320132 104 - 2996586 ---------ACGCUAGAAGCCCCAGCCCCACCAAGGGAGGCAUCCUGUCGC---CACUAUCGACUGGUUCCAGUGGAUUCGGUAGCCGCAGAUCCUCCCAGGAUGGUUUCCAGCUG ---------..(((.((((((((.(......)..((((((.(((.(((.((---.((((((.((((....)))).)))..))).)).)))))))))))).))..)))))).))).. ( -41.80, z-score = -2.71, R) >droSec1.super_1 7294728 104 - 14215200 ---------ACGCUAGAAGCCCCAGCCCCACCAAGGGAGGCAUCCUGUCGC---CACUAUCGACUGGUUCCAGUGGAUUCGGUAGCCGCAGAUCCUCCCAGGAUGGUUUCCAGCUG ---------..(((.((((((((.(......)..((((((.(((.(((.((---.((((((.((((....)))).)))..))).)).)))))))))))).))..)))))).))).. ( -41.80, z-score = -2.71, R) >droYak2.chr2R 13953329 104 + 21139217 ---------ACGCUAGAAGCCCCAGCCCCACCAAGGGAGGCAUUCUGUCGC---CACUAUCGACUGGUUCCAGUGGAUUCGGUAGCCGCAGAUCCUCCCAGGAUGGUUUCCAGCUG ---------..(((.((((((((.(......)..((((((...(((((.((---.((((((.((((....)))).)))..))).)).))))).)))))).))..)))))).))).. ( -43.20, z-score = -3.24, R) >droEre2.scaffold_4845 6537245 104 + 22589142 ---------ACGCUAGAAGCCCCAGCCCCACCAAGGGAGGCAUCCUGUCGC---CACUAUCGACUGGUUCCAGUGGAUUCGGUAGCCGCAGAUCCUCCCAGGAUGGUUUCCAGCUG ---------..(((.((((((((.(......)..((((((.(((.(((.((---.((((((.((((....)))).)))..))).)).)))))))))))).))..)))))).))).. ( -41.80, z-score = -2.71, R) >droAna3.scaffold_13266 17070706 104 - 19884421 ---------ACGCCAGAAGCCCCAGCCCCACCAAGGGAGGCAUCCUGUCGC---CACUAUCGACUGGUUCCAGUGGAUUCGGUAGCCGCAGGUCCUCCCAGGACGGUUUCCAGCUG ---------..((..((((((((.(......)..((((((...(((((.((---.((((((.((((....)))).)))..))).)).))))).)))))).))..))))))..)).. ( -42.70, z-score = -2.55, R) >dp4.chr3 8043412 113 - 19779522 CGGAGCAGAACGCUAGGAGCCCCAGCCCCACCAAGGGAGGCAUUCUGUCGC---CACUAUCGACUGGUUCCAGUGGAUUCGGUAGCCGCAGAUCCUCUCAGGACGGCUUCCAGCUG ...........(((.((((((...(((((......)).)))..(((((.((---.((((((.((((....)))).)))..))).)).)))))(((.....))).)))))).))).. ( -42.40, z-score = -0.90, R) >droPer1.super_4 1956908 113 - 7162766 CGGAGCAGAACGCCAGGAGCCCCAGCCCCACCAAGGGAGGCAUUCUGUCGC---CACUAUCGACUGGUUCCAGUGGAUUCGGUAGCCGCAGAUCCUCUCAGGACGGCUUCCAGCUG ...(((.(((.(((.((.((....)).)).((..((((((...(((((.((---.((((((.((((....)))).)))..))).)).))))).)))))).))..))))))..))). ( -43.60, z-score = -1.23, R) >droWil1.scaffold_181009 2143712 104 + 3585778 ---------ACGCCAGAAGCCCUAGCCCCACCAAGGGAGGCAUUCUGUCGC---CACUAUCGACUGGUUCCAGUGGAUUUGGUAGCCGCAGAUCGUCUCAGGAUGGCUUCCAGUUG ---------..((..((((((....((((.....))(((((..(((((.((---.((((((.((((....)))).)))..))).)).)))))..))))).))..))))))..)).. ( -39.90, z-score = -2.18, R) >droVir3.scaffold_12875 14304442 104 + 20611582 ---------ACGCCAGAAGCCCCAGCCCGACCAAGGGAGGUAUACUGUCGC---CACUAUCGACUGGUUCCAGUGGAUUCGGUAGCCGCAGAUCCUCUCAGGAUGGCUUCCAGCUC ---------..((..((((((((...((......))((((.((.((((.((---.((((((.((((....)))).)))..))).)).))))))))))...))..))))))..)).. ( -39.30, z-score = -1.75, R) >droMoj3.scaffold_6496 23722571 104 - 26866924 ---------ACGCCAGAAGCCCCAGCCCGACCAAGGGAGGCAUACUGUCGC---CACUAUCGACUGGUUCCAGUGGAUUCGGUAGCCGCAGAUCCUCUCAGGAUGGCUUCCAGCUC ---------..((..((((((((...((......))((((.((.((((.((---.((((((.((((....)))).)))..))).)).))))))))))...))..))))))..)).. ( -39.30, z-score = -1.61, R) >droGri2.scaffold_15112 3977931 104 + 5172618 ---------ACGCCAGAAGCCCCAGCCCGACCAAGGGAGGCAUACUGUCGC---CACUAUCGACUGGUUCCAGUGGAUUCGGUAGCCGCAGAUCCUCUCAGGAUGGCUUCCAGCUC ---------..((..((((((((...((......))((((.((.((((.((---.((((((.((((....)))).)))..))).)).))))))))))...))..))))))..)).. ( -39.30, z-score = -1.61, R) >anoGam1.chr3L 22763456 107 - 41284009 ---------AUGCACGAUCGCCCAGCCCGACCAAGGGUGGCCCGUCGCCGCUGUCGCCCGCGAGCGGAUCGAGCGGAUUCGGUAGCCGGCGAUCGUCGCAGGACGGUUUCCAGCUA ---------.((((((((((((..((((......))))((((((...(((((....(((....).))....)))))...)))..)))))))))))).)))(((.....)))..... ( -51.40, z-score = -1.45, R) >triCas2.ChLG7 7140271 101 - 17478683 ---------ACACCCG----CUCACCUUCACCGAGCAAAGGCUUGAGCCCG--UUAUCACCAGGAGGCUCAAGCGGUUUCUCGUCUCGUAGGUCGAGUCAAGAUGGUUUCCAUCUU ---------......(----(((((((....((((.((..((((((((((.--(.......).).)))))))))..)).))))......)))).)))).(((((((...))))))) ( -35.90, z-score = -3.21, R) >consensus _________ACGCCAGAAGCCCCAGCCCCACCAAGGGAGGCAUCCUGUCGC___CACUAUCGACUGGUUCCAGUGGAUUCGGUAGCCGCAGAUCCUCUCAGGAUGGUUUCCAGCUG ...........((..((((((((.((((........).)))...((((.((.......(((.((((....)))).)))......)).)))).........))..))))))..)).. (-25.08 = -25.43 + 0.35)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:21:06 2011