
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 9,666,118 – 9,666,238 |
| Length | 120 |
| Max. P | 0.890947 |

| Location | 9,666,118 – 9,666,238 |
|---|---|
| Length | 120 |
| Sequences | 14 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.43 |
| Shannon entropy | 0.51762 |
| G+C content | 0.60476 |
| Mean single sequence MFE | -47.49 |
| Consensus MFE | -24.28 |
| Energy contribution | -24.13 |
| Covariance contribution | -0.15 |
| Combinations/Pair | 1.50 |
| Mean z-score | -1.67 |
| Structure conservation index | 0.51 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.10 |
| SVM RNA-class probability | 0.890947 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 9666118 120 - 21146708 GCCUUCGGUGGUGGCGCCACUGGCAUCUGGUCCAACUCACACUACAUCUGGAAGCAGUUCCAGCGAGCUGGCUCCAUUUCCGGUGACCGUGUGUGGCGUCUGCCACUCUGGCAAUACUAC (((...((((((((((((((((((.....).)))....((((..((.(((((((..(..((((....))))..)..)))))))))...)))))))))))).))))))..)))........ ( -49.30, z-score = -2.40, R) >anoGam1.chr3R 2605590 120 - 53272125 GCGCUCGGUGGUGCUGCUAGCGGUGUGUUCUCCAACUCGGACUUUUUGUGGUCCCAGCUGCAGAAAGCCGGUGCGAUUAGUGGUGAUCGCUGCUGGCGUAUGCCGCUCUGGCAGUACUAC .......((((((((((((((((((((((((.((.((.(((((......))))).)).)).)))).(((((((((((((....))))))).)))))).)))))))))..))))))))))) ( -59.00, z-score = -4.05, R) >droSim1.chr2R 8106441 120 - 19596830 GCCUUCGGUGGUGGCGCCACUGGCAUCUGGUCCAACUCUCACUACAUCUGGAAGCAGUUCCAGCGAGCUGGCUCCAUUUCCGGUGACCGUGUGUGGCGUCUGCCACUCUGGCAAUACUAC (((...((((((((((((((..((((..((((...............(((((((..(..((((....))))..)..))))))).)))))))))))))))).))))))..)))........ ( -48.29, z-score = -2.26, R) >droSec1.super_1 7193332 120 - 14215200 GCCUUCGGUGGUGGCGCCACUGGCAUCUGGUCCAACUCUCACUACAUCUGGAAGCAGUUCCAGCGAGCUGGCUCCAUUUCCGGUGACCGUGUGUGGCGUCUGCCACUCUGGCAAUACUAC (((...((((((((((((((..((((..((((...............(((((((..(..((((....))))..)..))))))).)))))))))))))))).))))))..)))........ ( -48.29, z-score = -2.26, R) >droYak2.chr2R 13851020 120 + 21139217 GCCUUCGGUGGCGGCGCCACUGGCAUCUGGUCCAACUCCCACUACAUUUGGAAGCAGUUCCAGCGGGCCGGCUCCAUUUCCGGUGACCGUGUGUGGCGUCUGCCCCUCUGGCAGUACUAC (((...((.((((((((((((((...(((.(((((............)))))..)))..)))(((((((((........)))))..))))..)))))).)))))))...)))........ ( -50.50, z-score = -1.60, R) >droEre2.scaffold_4845 6435993 120 + 22589142 GCCUUUGGCGGUGGCGCCACUGGCAUCUGGUCCAACUCUCACUACAUUUGGAAGCAGUUCCAGCGGGCCGGCUCCAUUUCCGGUGACCGUGUGUGGCGUCUGCCACUCUGGCAGUACUAC (((..((((((..((((((((((...(((.(((((............)))))..)))..)))(((((((((........)))))..))))..)))))))))))))....)))........ ( -47.70, z-score = -1.23, R) >droAna3.scaffold_13266 16969638 120 - 19884421 GCCUUCGGUGGCGGUGCUACUGGCAUCUUCUCCAACUCGCACUACAUCUGGAAGCAGUUCCAGCGCGCCGGUGCCAUCUCCGGCGAUCGGUUGUGGCGUCUUCCCCUGUGGCACUACUAC (((......)))(((((((((((........)))...............(((((..((..((((.((((((........))))))....))))..))..)))))...))))))))..... ( -43.30, z-score = -0.95, R) >dp4.chr3 5340762 120 + 19779522 GCCUUCGGCGGCGGAGCCACCGGUGUCUUCUCCAACUCCCACUACAUCUGGAAGCAGUUCCAGCGCGCUGGAUCCCUCACUGGCGACCGCGUGUGGCGCCUGCCGCUCUGGCAGUACUAC (((...((((((((.(((((.((.(....).))..............((((((....)))))).((((.((.((((.....)).)))))))))))))..))))))))..)))........ ( -48.50, z-score = -1.19, R) >droPer1.super_4 3167727 120 - 7162766 GCCUUCGGCGGCGGGGCCACCGGUGUCUUCUCCAACUCCCACUACAUCUGGAAGCAGUUCCAGCGCGCUGGAUCCCUCACUGGCGACCGUGUGUGGCGCCUGCCGCUCUGGCAGUACUAC (((...((((((((((((((((((.......................((((((....))))))..(((..(........)..)))))))...))))).)))))))))..)))........ ( -51.40, z-score = -1.67, R) >droWil1.scaffold_181009 2508736 120 + 3585778 GCUUUCGGAGGCGGUGCCUCUGGCAUUUUCUCUAACUCACACUACAUCUGGAAGCAAUUCCAACGGGCUGGCUCCCUAACCGGUGACCGUUUAUGGCGUAUGCCUCUAUGGCAAUACUAC (((...(((((..(((((...)))))..)))))................((((....))))((((((((((........)))))..)))))...)))(((((((.....))).))))... ( -37.40, z-score = -1.91, R) >droVir3.scaffold_12875 8321557 120 - 20611582 GCGUACGGCGGCGGUGCUUCUGGCAUCUUCUCCAACUCGCAUUACGUCUGGAAACAGUUCCAGCGUGCUGGCUCCCUGACUGGUGAUCGCCUUUGGCGCAUGCCCCUCUGGCAAUACUAC ((((..(((((((((((.....)))))............((.((((.((((((....)))))))))).))))((((.....)).)).))))....)))).((((.....))))....... ( -40.10, z-score = -0.62, R) >droMoj3.scaffold_6496 7956589 120 + 26866924 GCCUACGGCGGCGGUGCGUCCGGCAUCUUCUCCAACUCGCACUACGUGUGGAAGCAGUUCCAGCGCGCCGGAUCCCUGACGGGCGAUCGCCUGUGGCGCAUGCCCCUCUGGCAAUACUAC (((...((.(((((((((((((((......((((...((.....))..)))).((.((....))))))))))).....((((((....)))))).)))).))))))...)))........ ( -49.90, z-score = -1.03, R) >droGri2.scaffold_15245 1770561 120 - 18325388 GCCUAUGGCGGCGGUGCCUCCGGCAUCUUCUCCAAUUCGCACUAUGUGUGGAAACAAUUCCAGAAGGCUGGUUCCUUGACCGGUGAUCGCAUGUGGCGCAUGCCCCUGUGGAAGUACUAU ..((((((.(((((((((...))))))...........((.((((((((((((....)))).((..(((((((....)))))))..)))))))))).))..))).))))))......... ( -42.80, z-score = -0.97, R) >triCas2.ChLG2 5349463 120 + 12900155 GCCUUGGGGUCCUCCGCGUCUGGAGUUUUCAGCAACAGCCAGACGAUGUGGUCGCAAGUGCGCAAAGCCGGGGUUAUAACCGGGGACCGCGUCUGGAGACUCCCUCUGUGGAAGUUCUUU .((..((((((.(((..((((((.(((......)))..))))))(((((((((((....))......((((........)))).))))))))).)))))).))).....))......... ( -48.40, z-score = -1.23, R) >consensus GCCUUCGGCGGCGGCGCCACUGGCAUCUUCUCCAACUCGCACUACAUCUGGAAGCAGUUCCAGCGAGCUGGCUCCAUUACCGGUGACCGUGUGUGGCGUCUGCCCCUCUGGCAGUACUAC (((...((.(((((((((..(((........................((((((....))))))...(((((........)))))..))).....))))).)))).))..)))........ (-24.28 = -24.13 + -0.15)

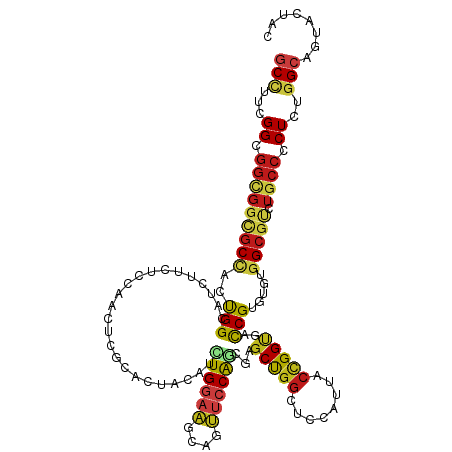
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:20:56 2011