
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 9,267,657 – 9,267,783 |
| Length | 126 |
| Max. P | 0.782886 |

| Location | 9,267,657 – 9,267,769 |
|---|---|
| Length | 112 |
| Sequences | 12 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 82.05 |
| Shannon entropy | 0.39840 |
| G+C content | 0.42635 |
| Mean single sequence MFE | -32.33 |
| Consensus MFE | -14.72 |
| Energy contribution | -14.84 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.28 |
| Structure conservation index | 0.46 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.67 |
| SVM RNA-class probability | 0.782886 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
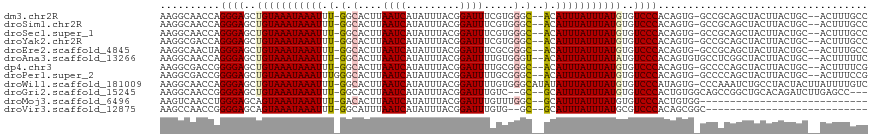
>dm3.chr2R 9267657 112 - 21146708 AAGGCAACCAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUCGUGGGC--ACAUUUAUUUAUGUGUCCCACAGUG-GCCGCAGCUACUUACUGC--ACUUUGCC ..(((((.((((((((((((((((.(((.-.......)))..))))))))).))))((((((--((((......))))).)))))((((-((....))))))..))).--...))))) ( -37.00, z-score = -3.11, R) >droSim1.chr2R 7725099 112 - 19596830 AAGGCAACCAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUCGUGGGC--ACAUUUAUUUAUGUGUCCCACAGUG-GCCGCAGCUACUUACUGC--ACUUUGCC ..(((((.((((((((((((((((.(((.-.......)))..))))))))).))))((((((--((((......))))).)))))((((-((....))))))..))).--...))))) ( -37.00, z-score = -3.11, R) >droSec1.super_1 6790724 112 - 14215200 AAGGCAACCAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUCGUGGGC--ACAUUUAUUUAUGUGUCCCACAGUG-GCCGCAGCUACUUACUGC--ACUUUGCC ..(((((.((((((((((((((((.(((.-.......)))..))))))))).))))((((((--((((......))))).)))))((((-((....))))))..))).--...))))) ( -37.00, z-score = -3.11, R) >droYak2.chr2R 13450626 112 + 21139217 AAGGCGACCAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUCGUGGGC--ACAUUUAUUUAUGUGUCCCACAGUG-GCCGCAGCUACUUACUGC--ACUUUGCC ..(((.((..(..(.(((((((((.(((.-.......)))..))))))))).)..)((((((--((((......))))).))))).)).-)))((((.......))))--........ ( -37.50, z-score = -3.18, R) >droEre2.scaffold_4845 6043217 112 + 22589142 AAGGCAACUAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUCGCGGGC--ACAUUUAUUUAUGUGUCCCACAGUG-GCCGCAGCUACUUACUGC--ACUUUGCC ..(((((...(..(.(((((((((.(((.-.......)))..))))))))).)..)((((((--((((......))))))))...((((-((....))))))....))--...))))) ( -34.50, z-score = -2.59, R) >droAna3.scaffold_13266 16575820 113 - 19884421 AAGGCAACCAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUUGUGGGU--ACAUUUAUUUAUAUGUCCCACAGUGUGCCUCGGCUACUUACUGC--ACUUUUUC ((((((...(((.(((((...........-(((((..((((.........))))(((((((.--((((........))))))))))).))))).))))).)))..)))--.))).... ( -30.10, z-score = -2.10, R) >dp4.chr3 19138178 112 + 19779522 AAGGCGACCGGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUUGCGGGC--ACAUUUAUUUAUGUGUCCCACAGUG-GCCCCAGCUACUUACUGC--ACUUUUCG ....(((..((((.(((((..........-.(((...((((.........)))).)))((((--((((......)))))))).))))).-.)))).((........))--.....))) ( -32.30, z-score = -1.90, R) >droPer1.super_2 8229179 113 + 9036312 AAGGCGACCGGGGAGCUGUAAAUAAAUUUGGGCACUUAAUCAUAUUUACGGAUUUUGCGGGC--ACAUUUAUUUAUGUGUCCCACAGUG-GCCCCAGCUACUUACUGC--ACUUUCCG ........(((..(((((((((((...((((....))))...)))))))))....(((((((--((((......))))))))...((((-((....))))))....))--)))..))) ( -35.80, z-score = -2.49, R) >droWil1.scaffold_181009 1454101 116 + 3585778 AAGGCAACCAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUUGUGGGCAUAUAUUUAUUUAUGUGUCCCAUAGUG-CCCAAAUCUGCCUACUACUUAUUUUGUC .(((((..(((....))).......((((-(((((..((((.........)))).((((((((((((......)))))).)))))))))-.)))))).)))))............... ( -31.50, z-score = -2.24, R) >droGri2.scaffold_15245 10788031 110 - 18325388 AAGGCAACCGGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUGUC--GC--GCAUUUAUUUAUGUGUCCCACUGUGGCAGCCGGCUGCACAGAUCUUGAGCC--- ..(((....(((..((.((((((((((.(-((((...((((.........))))))))--).--..))))))))))))..))).(((((.(((....)))))))).......)))--- ( -31.90, z-score = -0.88, R) >droMoj3.scaffold_6496 5332103 87 - 26866924 AAGUCAACCUGGGAGCAGUAAAUAAAUUU-GACACUUAAUCAUAUUUACGGAUUUGUUUGGC--GCAUUUAUUUAUGUGUCCCACUGUGG---------------------------- ..............(((((((((((((((-(.................)))))))))))(((--((((......)))))))..)))))..---------------------------- ( -17.33, z-score = -0.29, R) >droVir3.scaffold_12875 20440375 86 + 20611582 AAGCCAACCGGGGAGCAGUAAAUAAAUUU-GGCAUUUAAUCAUAUUUACGGAUUUGUG--GC--GCAUUUAUUUAUGCGUCCCACAGCGGC--------------------------- ..(((.....(((((((.(((((((((.(-(.(((..((((.........)))).)))--.)--).)))))))))))).)))).....)))--------------------------- ( -26.00, z-score = -2.35, R) >consensus AAGGCAACCAGGGAGCUGUAAAUAAAUUU_GGCACUUAAUCAUAUUUACGGAUUUCGUGGGC__ACAUUUAUUUAUGUGUCCCACAGUG_GCCGCAGCUACUUACUGC__ACUUUGCC ..........((((..(((((((((((....((....((((.........))))......))....)))))))))))..))))................................... (-14.72 = -14.84 + 0.12)


| Location | 9,267,692 – 9,267,783 |
|---|---|
| Length | 91 |
| Sequences | 11 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 82.89 |
| Shannon entropy | 0.35583 |
| G+C content | 0.35664 |
| Mean single sequence MFE | -22.24 |
| Consensus MFE | -11.93 |
| Energy contribution | -12.11 |
| Covariance contribution | 0.18 |
| Combinations/Pair | 1.30 |
| Mean z-score | -1.90 |
| Structure conservation index | 0.54 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.566140 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 9267692 91 - 21146708 ------------CUGCCUUUUUUUUAAAGGCAACCAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUCGUGGGC--ACAUUUAUUUAUGUGU ------------.(((((((.....))))))).(((.(((((((((((((.(((.-.......)))..))))))))).)))).)))((--((((......)))))) ( -28.70, z-score = -4.44, R) >droSim1.chr2R 7725134 89 - 19596830 ------------CUGCCU--UUUUUAAAGGCAACCAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUCGUGGGC--ACAUUUAUUUAUGUGU ------------.(((((--(.....)))))).(((.(((((((((((((.(((.-.......)))..))))))))).)))).)))((--((((......)))))) ( -27.30, z-score = -3.93, R) >droSec1.super_1 6790759 89 - 14215200 --------------CUUCCUUUUUUAAAGGCAACCAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUCGUGGGC--ACAUUUAUUUAUGUGU --------------...((((.....))))...(((.(((((((((((((.(((.-.......)))..))))))))).)))).)))((--((((......)))))) ( -21.90, z-score = -2.49, R) >droYak2.chr2R 13450661 89 + 21139217 ------------CUGCCU--UUUUUAAAGGCGACCAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUCGUGGGC--ACAUUUAUUUAUGUGU ------------.(((((--(.....)))))).(((.(((((((((((((.(((.-.......)))..))))))))).)))).)))((--((((......)))))) ( -26.60, z-score = -3.52, R) >droEre2.scaffold_4845 6043252 89 + 22589142 --------------UUGCCUUUUUUAAAGGCAACUAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUCGCGGGC--ACAUUUAUUUAUGUGU --------------(((((((.....)))))))((.(..(.(((((((((.(((.-.......)))..))))))))).)..)..))((--((((......)))))) ( -22.40, z-score = -2.67, R) >droAna3.scaffold_13266 16575856 101 - 19884421 --UUCCAUCCCUCCCCUCCGUUUUUAAAGGCAACCAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUUGUGGGU--ACAUUUAUUUAUAUGU --.....(((((.....((.........)).....)))))(((((((((((((.(-(.(((..((((.........))))..)).).)--).))))))))))).)) ( -18.90, z-score = 0.30, R) >dp4.chr3 19138213 97 + 19779522 ------GUACGCAGGCUGCUUUUUUAAAGGCGACCGGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUUGCGGGC--ACAUUUAUUUAUGUGU ------...((((((.((((((....)))))).))......(((((((((.(((.-.......)))..)))))))))....)))).((--((((......)))))) ( -24.20, z-score = -1.50, R) >droPer1.super_2 8229214 98 + 9036312 ------GUACGCACGCUGCUUUUUUAAAGGCGACCGGGGAGCUGUAAAUAAAUUUGGGCACUUAAUCAUAUUUACGGAUUUUGCGGGC--ACAUUUAUUUAUGUGU ------.......((((...........)))).(((.(((((((((((((...((((....))))...))))))))).)))).)))((--((((......)))))) ( -25.00, z-score = -1.74, R) >droWil1.scaffold_181009 1454138 104 + 3585778 CUGCGAAUACGAAUGCGAC-UUUUUAAAGGCAACCAGGGAGCUGUAAAUAAAUUU-GGCACUUAAUCAUAUUUACGGAUUUUGUGGGCAUAUAUUUAUUUAUGUGU .(((.....((....)).(-((....)))))).(((.(((((((((((((.(((.-.......)))..))))))))).)))).)))(((((((......))))))) ( -20.70, z-score = -0.70, R) >droMoj3.scaffold_6496 5332113 89 - 26866924 -------------AUUUCUU-UUUUAAAGUCAACCUGGGAGCAGUAAAUAAAUUU-GACACUUAAUCAUAUUUACGGAUUUGUUUGGC--GCAUUUAUUUAUGUGU -------------.......-.............(((....)))(((((((((((-(.................))))))))))))((--((((......)))))) ( -13.13, z-score = -0.04, R) >droVir3.scaffold_12875 20440386 88 + 20611582 -------------AUAUCUUAUUUUAAAGCCAACCGGGGAGCAGUAAAUAAAUUU-GGCAUUUAAUCAUAUUUACGGAUUUG--UGGC--GCAUUUAUUUAUGCGU -------------................((....))...(((.(((((((((.(-(.(((..((((.........)))).)--)).)--).)))))))))))).. ( -15.80, z-score = -0.14, R) >consensus _____________UGCUCCUUUUUUAAAGGCAACCAGGGAGCUGUAAAUAAAUUU_GGCACUUAAUCAUAUUUACGGAUUUCGUGGGC__ACAUUUAUUUAUGUGU .................................(((.(((((((((((((..................))))))))).)))).)))....((((......)))).. (-11.93 = -12.11 + 0.18)


Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:19:55 2011