
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 8,221,417 – 8,221,525 |
| Length | 108 |
| Max. P | 0.888489 |

| Location | 8,221,417 – 8,221,525 |
|---|---|
| Length | 108 |
| Sequences | 13 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 84.69 |
| Shannon entropy | 0.35358 |
| G+C content | 0.56731 |
| Mean single sequence MFE | -37.86 |
| Consensus MFE | -32.65 |
| Energy contribution | -32.80 |
| Covariance contribution | 0.16 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.31 |
| Structure conservation index | 0.86 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.09 |
| SVM RNA-class probability | 0.888489 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 8221417 108 + 21146708 ----CAUAUUGCAGCA---UUGCCGUGACCAGGAUUCGAACCUGGGUUACCACGGCCACAACGUGGGGUCCUAACCACUAGACGAUCACGGCUACGUCGGGUUGUUGGGUGGAGC ----.........((.---..(((....(((((.......)))))....(((((.......)))))))).....(((((.(((((((.((((...))))))))))).))))).)) ( -38.60, z-score = -0.86, R) >droSim1.chr2R 12903775 108 - 19596830 ----UUUAAUUCAACA---UUGCCGUGACCAGGAUUCGAACCUGGGUUACCACGGCCACAACGUGGGGUCCUAACCACUAGACGAUCACGGCUGCAUUGGGUUGCUGGGCGCGGC ----...(((((((..---..((((((((((((.......)))))(((.(((((.......)))))(((....)))....)))..)))))))....)))))))((((....)))) ( -38.60, z-score = -1.44, R) >droSec1.super_1 5767205 108 + 14215200 ----CAUAUCGCAGCA---UUGCCGUGACCAGGAUUCGAACCUGGGUUACCACGGCCACAACGUGGGGUCCUAACCACUAGACGAUCACGGCUACGUCGGGUUGUUGGAUGGAGG ----..((((.(((((---..((((((((((((.......)))))(((.(((((.......)))))(((....)))....)))..)))))))..........))))))))).... ( -37.40, z-score = -0.79, R) >droYak2.chr2R 12410095 108 - 21139217 ----CAUAUCGCAGCA---UUGCCGUGACCAGGAUUCGAACCUGGGUUACCACGGCCACAACGUGGGGUCCUAACCACUAGACGAUCACGGCUACGUCGGGUUGACGGACGAAGC ----......((....---..((((((((((((.......)))))(((.(((((.......)))))(((....)))....)))..)))))))..((((.(.....).))))..)) ( -39.50, z-score = -1.79, R) >droEre2.scaffold_4845 5018250 108 - 22589142 ----CAUAUCGCAGCA---UUGCCGUGACCAGGAUUCGAACCUGGGUUACCACGGCCACAACGUGGGGUCCUAACCACUAGACGAUCACGGCUACGUCGGGUUGUAGGAUGAGGC ----......((..((---((((((((((((((.......)))))(((.(((((.......)))))(((....)))....)))..)))))))((((......)))).))))..)) ( -37.60, z-score = -0.98, R) >droAna3.scaffold_13266 5513148 108 + 19884421 ----CAGGUUUCAACA---UUGCCGUGACCAGGAUUCGAACCUGGGUUACCACGGCCACAACGUGGGGUCCUAACCACUAGACGAUCACGGCUACGCCCGGCUGUUGAAAGAUGC ----((..((((((((---..((((((((((((.......)))))(((.(((((.......)))))(((....)))....)))..)))))))...((...))))))))))..)). ( -39.70, z-score = -2.14, R) >dp4.chr3 16978841 108 - 19779522 ----CGUAAUGCAGCA---UUGCCGUGACCAGGAUUCGAACCUGGGUUACCACGGCCACAACGUGGGGUCCUAACCACUAGACGAUCACGGCUACCUCGGGCUGUUGGAACUCGG ----.((...(((((.---..((((((((((((.......)))))(((.(((((.......)))))(((....)))....)))..)))))))..(....))))))....)).... ( -38.30, z-score = -1.16, R) >droPer1.super_2 6045618 108 - 9036312 ----CGUAAUGCAGCA---UUGCCGUGACCAGGAUUCGAACCUGGGUUACCACGGCCACAACGUGGGGUCCUAACCACUAGACGAUCACGGCUACCUCGGGCUGUUGGAACUCGG ----.((...(((((.---..((((((((((((.......)))))(((.(((((.......)))))(((....)))....)))..)))))))..(....))))))....)).... ( -38.30, z-score = -1.16, R) >droWil1.scaffold_180745 459631 108 + 2843958 ----CGUAUUGCAGCA---UUGCCGUGACCAGGAUUCGAACCUGGGUUACCACGGCCACAACGUGGGGUCCUAACCACUAGACGAUCACGGCUACGUCGGGUGCUUAAUAAACAG ----..(((((.((((---((((((((((((((.......)))))(((.(((((.......)))))(((....)))....)))..))))))).......)))))))))))..... ( -37.21, z-score = -1.84, R) >droVir3.scaffold_13324 2649084 108 + 2960039 ----UGAGUUACAUCA---CUGCCGUGACCAGGAUUCGAACCUGGGUUACCACGGCCACAACGUGGGGUCCUAACCACUAGACGAUCACGGCUACGUCUGGCUGUUGAAACGCAU ----..(((((.((..---..((((((((((((.......)))))(((.(((((.......)))))(((....)))....)))..)))))))...)).)))))............ ( -34.20, z-score = -0.82, R) >droMoj3.scaffold_6496 641981 104 - 26866924 UGCCUUCCUUUGGGCA---UUGUCGUGACCAGGAUUCGAACCUGGGUUACCACGGCCACAACGUGGGGUCCUAACCACUAGACGAUCACGGCUACGCCGGGUUAUUA-------- .((((.......(((.---..((((((((((((.......)))))(((.(((((.......)))))(((....)))....)))..)))))))...))))))).....-------- ( -35.81, z-score = -0.91, R) >droGri2.scaffold_15112 4900648 111 + 5172618 ----UAGGCUCCAGCAAUAUUGCCGUGACCAGGAUUCGAACCUGGGUUACCACGGCCACAACGUGGGGUCCUAACCACUAGACGAUCACGGCUAUGCCUGGGCGAUGCAAGCCGA ----..((((...(((.....((((((((((((.......)))))(((.(((((.......)))))(((....)))....)))..)))))))..(((....))).))).)))).. ( -43.20, z-score = -2.26, R) >anoGam1.chr2L 19552905 108 - 48795086 ----CUACUGGCAACA---CUGCCGUGACCAGGAUUCGAACCUGGGUUACUACGGCCACAACGUAGGGUCCUAACCACUAGACGAUCACGGCUUCAGAUGCUUGGUUGGAAAGGG ----...(.((((.(.---..((((((((((((.......)))))(((.(((((.......)))))(((....)))....)))..)))))))....).)))).)........... ( -33.70, z-score = -0.92, R) >consensus ____CAUAUUGCAGCA___UUGCCGUGACCAGGAUUCGAACCUGGGUUACCACGGCCACAACGUGGGGUCCUAACCACUAGACGAUCACGGCUACGUCGGGUUGUUGGAAGACGC ...........((((......((((((((((((.......)))))(((.(((((.......)))))(((....)))....)))..)))))))........))))........... (-32.65 = -32.80 + 0.16)

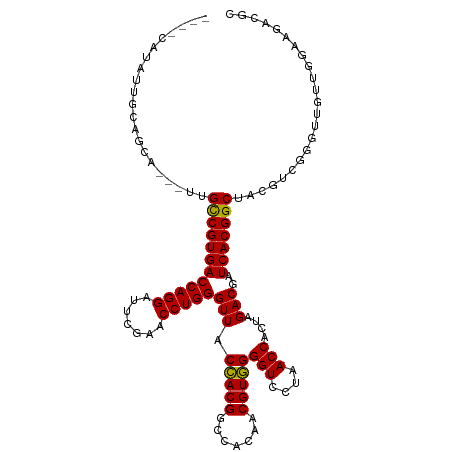
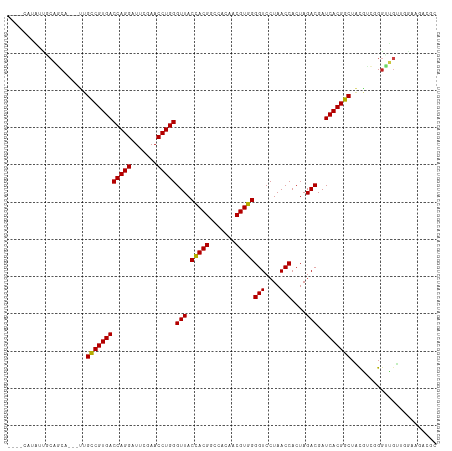
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:17:36 2011