
| Sequence ID | dm3.chr2R |
|---|---|
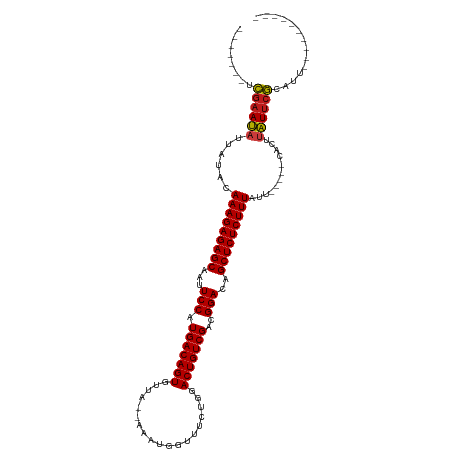
| Location | 8,057,876 – 8,057,971 |
| Length | 95 |
| Max. P | 0.999646 |

| Location | 8,057,876 – 8,057,971 |
|---|---|
| Length | 95 |
| Sequences | 14 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 81.59 |
| Shannon entropy | 0.36706 |
| G+C content | 0.37263 |
| Mean single sequence MFE | -28.72 |
| Consensus MFE | -22.62 |
| Energy contribution | -22.64 |
| Covariance contribution | 0.02 |
| Combinations/Pair | 1.08 |
| Mean z-score | -4.61 |
| Structure conservation index | 0.79 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 4.13 |
| SVM RNA-class probability | 0.999646 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
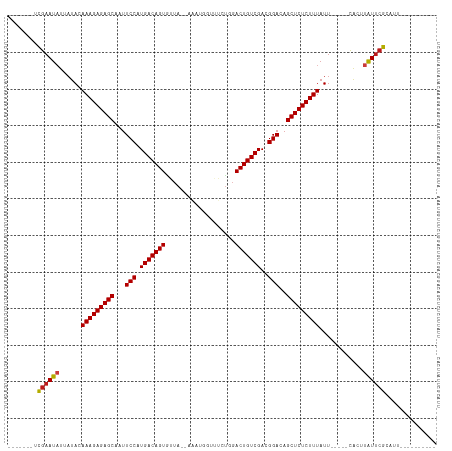
>dm3.chr2R 8057876 95 + 21146708 -------UCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUGAUAUUAAAUAGUUUCUGGACUGUCGACGGACAGCUCUCUUUAUU-----CACUUAUUCGCAUU---------- -------.((((((......(((((((((...(((.(((((((((.(((....))).))...)))))))..)))..)))))))))...-----....))))))....---------- ( -27.12, z-score = -4.04, R) >droSim1.chr2R_random 2148105 95 + 2996586 -------UCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUGAUAUUAAAUGCUUUCUGGACUGUCGACGGACAGCUCUCUUUAUU-----CACUUAUUCGCAUU---------- -------.((((((......(((((((((...((((.((.(((..........))).)))))).((((....)))))))))))))...-----....))))))....---------- ( -27.12, z-score = -3.97, R) >droSec1.super_1 5602978 95 + 14215200 -------UCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUGAUAUUAAAUGGUUUCUGGACUGUCGACGGACAGCUCUCUUUAUU-----CACUUAUUCGCAUU---------- -------.((((((......(((((((((...(((.(((((((((.(((....))).))...)))))))..)))..)))))))))...-----....))))))....---------- ( -27.02, z-score = -3.63, R) >droYak2.chr2R 12245779 95 - 21139217 -------UCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUGAUAUUAAAUGAAUUCUGGACUGUCGACGGACAGCUCUCUUUAUU-----CACUUAUUCGCAUA---------- -------.((((((......(((((((((...(((.(((((((...................)))))))..)))..)))))))))...-----....))))))....---------- ( -26.53, z-score = -3.78, R) >droEre2.scaffold_4845 4852855 95 - 22589142 -------UCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUGAUAUUAAAUGGUUUCUGGACUGUCGACGGACAGCUCUCUUUAUU-----CACUUAUUCGCAUA---------- -------.((((((......(((((((((...(((.(((((((((.(((....))).))...)))))))..)))..)))))))))...-----....))))))....---------- ( -27.02, z-score = -3.60, R) >droAna3.scaffold_13266 17315582 91 - 19884421 -------UCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUGUUA----UAGGUAUCUGGACUGUCGACGGACAGCUCUCUUUAUU-----CACGUAUUCGCAUU---------- -------.(((((((.....(((((((((...(((.(((((((....----(((....))).)))))))..)))..)))))))))...-----...)))))))....---------- ( -29.00, z-score = -3.99, R) >dp4.chr3 418422 93 + 19779522 -------UCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUA--AAUUGGUACGUUGACUGUCGACGGACAGCUCUCUUUAUU-----CACCUAUUCGCAUU---------- -------.((((((......(((((((((...(((.(((((((....--(((......))).)))))))..)))..)))))))))...-----....))))))....---------- ( -26.82, z-score = -4.12, R) >droPer1.super_2 605319 93 + 9036312 -------UCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUA--AAUUGGUACGUUGACUGUCGACGGACAGCUCUCUUUAUU-----CACCUAUUCGCAUU---------- -------.((((((......(((((((((...(((.(((((((....--(((......))).)))))))..)))..)))))))))...-----....))))))....---------- ( -26.82, z-score = -4.12, R) >droWil1.scaffold_181141 2061257 95 - 5303230 -------UCGAAUAUCAUACAAAGAGAGCAAUUCCAUGACAGUUUUAAAAUCGUUUAACUGGACUGUCGACGGACAGCUCUCUUUAUU-----CACUUAUUCGCUUU---------- -------.((((((......(((((((((...(((.(((((((((((((....)))))...))))))))..)))..)))))))))...-----....))))))....---------- ( -28.82, z-score = -5.09, R) >droVir3.scaffold_12875 781566 93 - 20611582 -------UCGAAUAUCAUACAAAGAGAGCAAUUCCAUGACAGUAUUACAUUU--GUUACUGGACUGUCGACGGACAGCUCUCUUUAUU-----CACUUAUUCGCUUU---------- -------.((((((......(((((((((...((((((((((........))--)))).)))).((((....)))))))))))))...-----....))))))....---------- ( -29.32, z-score = -5.43, R) >droMoj3.scaffold_6496 6376232 94 + 26866924 -------UCGAAUAUCAUACAAAGAGAGCAAUUCCAUGACAGUUUUACAAUC--UUUACUGGACUGUCGACGGACAGCUCUCUUUAUU-----CACUUAUUCGCAUUU--------- -------.((((((......(((((((((...(((.(((((((((.......--......)))))))))..)))..)))))))))...-----....)))))).....--------- ( -28.84, z-score = -5.95, R) >droGri2.scaffold_15245 147100 93 - 18325388 -------UCGAAUAUCAUACAAAGAGAGCAAUUCCAUGACAGUAUUAAAAUU--UAUACUGGACUGUCGACGGACAGCUCUCUUUAUU-----CACUUAUUCGCUUU---------- -------.((((((......(((((((((..........((((((.......--.))))))...((((....)))))))))))))...-----....))))))....---------- ( -27.12, z-score = -5.23, R) >anoGam1.chr2L 16793194 89 + 48795086 -------UCGAAUUGUACAUAAAGAGAGCAAUUCCAUGACAGUGA------AUUAUAUCCCUACUGUCGACGGAUAGCUCUCUUUAUU-----UUCAUAUUCGAUUG---------- -------((((((((...(((((((((((...(((.((((((((.------..........))))))))..)))..))))))))))).-----..)).))))))...---------- ( -32.90, z-score = -6.81, R) >apiMel3.Group1 18691515 113 + 25854376 UUGAACAUAGCAGUGUCUACAAAGAGAGCAACUCCAUGACAGUGUC----UAUUAUAACCAUACUGUCGAUGGAUAGCUCUCUUUAUAGCGCGCGCCUUUCGUCGUUUUUCUUCAUU ..(((....((.(((.(((.(((((((((...(((((((((((((.----..........)))))))).)))))..))))))))).))))))))....)))................ ( -37.70, z-score = -4.81, R) >consensus _______UCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUGUUA__AAAUGGUUUCUGGACUGUCGACGGACAGCUCUCUUUAUU_____CACUUAUUCGCAUU__________ ........((((((......(((((((((...(((.(((((((...................)))))))..)))..)))))))))............)))))).............. (-22.62 = -22.64 + 0.02)

| Location | 8,057,876 – 8,057,971 |
|---|---|
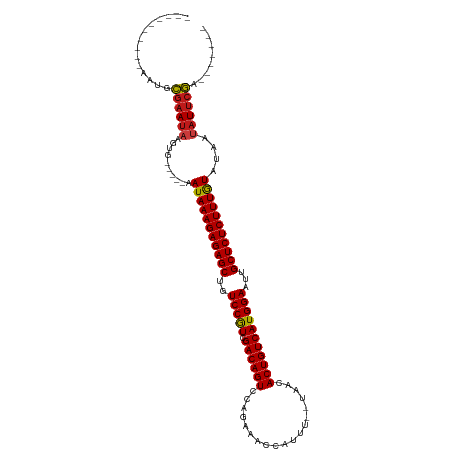
| Length | 95 |
| Sequences | 14 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 81.59 |
| Shannon entropy | 0.36706 |
| G+C content | 0.37263 |
| Mean single sequence MFE | -34.99 |
| Consensus MFE | -29.70 |
| Energy contribution | -29.79 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.11 |
| Mean z-score | -6.02 |
| Structure conservation index | 0.85 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.48 |
| SVM RNA-class probability | 0.998771 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
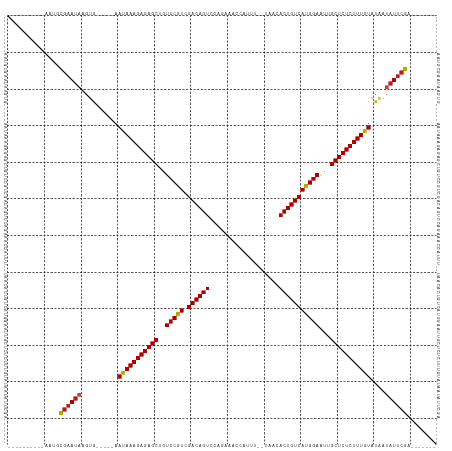
>dm3.chr2R 8057876 95 - 21146708 ----------AAUGCGAAUAAGUG-----AAUAAAGAGAGCUGUCCGUCGACAGUCCAGAAACUAUUUAAUAUCACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGA------- ----------....((((((..((-----.(((((((((((..(((((.((((((...................)))))))))))...))))))))))).)).)))))).------- ( -33.81, z-score = -5.89, R) >droSim1.chr2R_random 2148105 95 - 2996586 ----------AAUGCGAAUAAGUG-----AAUAAAGAGAGCUGUCCGUCGACAGUCCAGAAAGCAUUUAAUAUCACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGA------- ----------....((((((..((-----.(((((((((((..(((((.((((((...................)))))))))))...))))))))))).)).)))))).------- ( -33.81, z-score = -5.52, R) >droSec1.super_1 5602978 95 - 14215200 ----------AAUGCGAAUAAGUG-----AAUAAAGAGAGCUGUCCGUCGACAGUCCAGAAACCAUUUAAUAUCACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGA------- ----------....((((((..((-----.(((((((((((..(((((.((((((...................)))))))))))...))))))))))).)).)))))).------- ( -33.81, z-score = -5.97, R) >droYak2.chr2R 12245779 95 + 21139217 ----------UAUGCGAAUAAGUG-----AAUAAAGAGAGCUGUCCGUCGACAGUCCAGAAUUCAUUUAAUAUCACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGA------- ----------....((((((..((-----.(((((((((((..(((((.((((((...................)))))))))))...))))))))))).)).)))))).------- ( -33.81, z-score = -5.66, R) >droEre2.scaffold_4845 4852855 95 + 22589142 ----------UAUGCGAAUAAGUG-----AAUAAAGAGAGCUGUCCGUCGACAGUCCAGAAACCAUUUAAUAUCACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGA------- ----------....((((((..((-----.(((((((((((..(((((.((((((...................)))))))))))...))))))))))).)).)))))).------- ( -33.81, z-score = -5.93, R) >droAna3.scaffold_13266 17315582 91 + 19884421 ----------AAUGCGAAUACGUG-----AAUAAAGAGAGCUGUCCGUCGACAGUCCAGAUACCUA----UAACACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGA------- ----------....((((((..((-----.(((((((((((..(((((.((((((...........----....)))))))))))...))))))))))).)).)))))).------- ( -34.06, z-score = -5.95, R) >dp4.chr3 418422 93 - 19779522 ----------AAUGCGAAUAGGUG-----AAUAAAGAGAGCUGUCCGUCGACAGUCAACGUACCAAUU--UAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGA------- ----------....((((((..((-----.(((((((((((..(((((.((((((.............--....)))))))))))...))))))))))).)).)))))).------- ( -35.03, z-score = -6.02, R) >droPer1.super_2 605319 93 - 9036312 ----------AAUGCGAAUAGGUG-----AAUAAAGAGAGCUGUCCGUCGACAGUCAACGUACCAAUU--UAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGA------- ----------....((((((..((-----.(((((((((((..(((((.((((((.............--....)))))))))))...))))))))))).)).)))))).------- ( -35.03, z-score = -6.02, R) >droWil1.scaffold_181141 2061257 95 + 5303230 ----------AAAGCGAAUAAGUG-----AAUAAAGAGAGCUGUCCGUCGACAGUCCAGUUAAACGAUUUUAAAACUGUCAUGGAAUUGCUCUCUUUGUAUGAUAUUCGA------- ----------....((((((....-----.(((((((((((..(((((.((((((....(((((....))))).)))))))))))...)))))))))))....)))))).------- ( -35.40, z-score = -6.20, R) >droVir3.scaffold_12875 781566 93 + 20611582 ----------AAAGCGAAUAAGUG-----AAUAAAGAGAGCUGUCCGUCGACAGUCCAGUAAC--AAAUGUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUGAUAUUCGA------- ----------....((((((....-----.(((((((((((..(((((.((((((...((.((--....)).)))))))))))))...)))))))))))....)))))).------- ( -34.30, z-score = -6.12, R) >droMoj3.scaffold_6496 6376232 94 - 26866924 ---------AAAUGCGAAUAAGUG-----AAUAAAGAGAGCUGUCCGUCGACAGUCCAGUAAA--GAUUGUAAAACUGUCAUGGAAUUGCUCUCUUUGUAUGAUAUUCGA------- ---------.....((((((....-----.(((((((((((..(((((.((((((.((((...--.))))....)))))))))))...)))))))))))....)))))).------- ( -35.50, z-score = -6.69, R) >droGri2.scaffold_15245 147100 93 + 18325388 ----------AAAGCGAAUAAGUG-----AAUAAAGAGAGCUGUCCGUCGACAGUCCAGUAUA--AAUUUUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUGAUAUUCGA------- ----------....((((((....-----.(((((((((((..(((((.((((((....((..--.....))..)))))))))))...)))))))))))....)))))).------- ( -33.60, z-score = -6.15, R) >anoGam1.chr2L 16793194 89 - 48795086 ----------CAAUCGAAUAUGAA-----AAUAAAGAGAGCUAUCCGUCGACAGUAGGGAUAUAAU------UCACUGUCAUGGAAUUGCUCUCUUUAUGUACAAUUCGA------- ----------...((((((.((..-----.(((((((((((..(((((.((((((..(.......)------..)))))))))))...)))))))))))...))))))))------- ( -35.80, z-score = -6.97, R) >apiMel3.Group1 18691515 113 - 25854376 AAUGAAGAAAAACGACGAAAGGCGCGCGCUAUAAAGAGAGCUAUCCAUCGACAGUAUGGUUAUAAUA----GACACUGUCAUGGAGUUGCUCUCUUUGUAGACACUGCUAUGUUCAA ..((((.(.......(....)(((.(.((((((((((((((..(((((.((((((...(((......----))))))))))))))...))))))))))))).).))))..).)))). ( -42.10, z-score = -5.23, R) >consensus __________AAUGCGAAUAAGUG_____AAUAAAGAGAGCUGUCCGUCGACAGUCCAGAAACCAUUU__UAACACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGA_______ ..............((((((..........(((((((((((..(((((.((((((...................)))))))))))...)))))))))))....))))))........ (-29.70 = -29.79 + 0.09)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:17:06 2011