
| Sequence ID | dm3.chr2R |
|---|---|
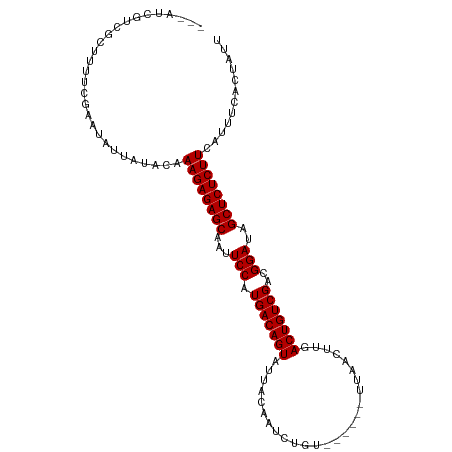
| Location | 8,057,642 – 8,057,747 |
| Length | 105 |
| Max. P | 0.999489 |

| Location | 8,057,642 – 8,057,747 |
|---|---|
| Length | 105 |
| Sequences | 15 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 78.20 |
| Shannon entropy | 0.50757 |
| G+C content | 0.36773 |
| Mean single sequence MFE | -25.03 |
| Consensus MFE | -20.82 |
| Energy contribution | -20.82 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.81 |
| Structure conservation index | 0.83 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.61 |
| SVM RNA-class probability | 0.999031 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 8057642 105 + 21146708 AUAAUCGUCGCCUUUCGAAUGUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUACAAUCGGU-----CUUAACUUGACUGUCGACGGAUAGCUCUCUUCAUUUCACAAUU (((((..(((.....)))..)))))...((((((((...(((.(((((((............-----.........)))))))..)))..))))))))............ ( -23.80, z-score = -2.09, R) >droAna3.scaffold_13266 17315378 104 - 19884421 ---AUCGUCGCCUUUCGAAUGUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUACAAUCCGUGU---UUUAACUUGACUGUCGACGGAUAGCUCUCUUCAUUUCACUAUU ---.(((........)))..........((((((((...(((.(((((((..(((.....))).---.........)))))))..)))..))))))))............ ( -23.20, z-score = -1.88, R) >droVir3.scaffold_12875 781368 101 - 20611582 ---GUCGUCGCUUUUCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUACAAUC--UG----UAUAGCUUGACUGUCGACGGAUAGCUCUCUUCAUUUCGCUAUU ---.(((........)))..........((((((((...(((.(((((((..(((....--.)----)).......)))))))..)))..))))))))............ ( -24.50, z-score = -1.87, R) >droMoj3.scaffold_6496 6376056 103 + 26866924 ---AUCGUCGAUUUUCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUAAAAUAUGUA----UAUAACUUGACUGUCGACGGAUAGCUCUCUUCAUUUCGCUAUU ---....(((.....)))..........((((((((...(((.(((((((......(((....----)))......)))))))..)))..))))))))............ ( -24.00, z-score = -2.38, R) >droGri2.scaffold_15245 146894 103 - 18325388 ---AUCGUCGCUUUUCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUACAAUCGGUA----UAUAACUUGACUGUCGACGGAUAGCUCUCUUCAUUUCGCUAUU ---.(((........)))..........((((((((...(((.(((((((..(((.....)))----.........)))))))..)))..))))))))............ ( -23.50, z-score = -1.94, R) >apiMel3.Group1 18691508 90 + 25854376 AUAAUCGUUGAACAUAGCAGUGUCUACAAAGAGAGCAACUCCAUGACAGUGUC-UAUUAUAA----------CCAUACUGUCGAUGGAUAGCUCUCUUUAU--------- ......((.((.(((....))))).))(((((((((...(((((((((((((.-........----------..)))))))).)))))..)))))))))..--------- ( -30.20, z-score = -4.62, R) >anoGam1.chr2L 16793180 97 + 48795086 UUGCACGUUGAAUAUCGAAUUGUACAUAAAGAGAGCAAUUCCAUGACAGUG---AAUUAUAU----------CCCUACUGUCGACGGAUAGCUCUCUUUAUUUUCAUAUU .((((..(((.....)))..)))).(((((((((((...(((.((((((((---........----------...))))))))..)))..)))))))))))......... ( -29.10, z-score = -4.73, R) >droWil1.scaffold_181141 2061049 110 - 5303230 AAAACCGUCGAUUUUCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUACAAUUCGGAUGUAUUAAACUGGACUGUCGACGGAUAGCUCUCUUCAUUUCGCUAUU .......(((.....)))..........((((((((...(((.(((((((..((((.......)))).........)))))))..)))..))))))))............ ( -24.70, z-score = -1.68, R) >droEre2.scaffold_4845 4852621 105 - 22589142 CUAAUCGUCGCCUUUCGAAUGUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUACAAUCGGU-----AUUAACUUGACUGUCGACGGAUAGCUCUCUUCAUUUCACAAUU ....(((........))).(((......((((((((...(((.(((((((..(((.....))-----)........)))))))..)))..))))))))......)))... ( -24.50, z-score = -2.40, R) >droYak2.chr2R 12245557 105 - 21139217 CUAAUCGUCGCCUUUCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUACAAUUGGU-----AUUAACUUGACUGUCGACGGAUAGCUCUCUUCAUUUCACUAUU ....(((........)))..........((((((((...(((.(((((((..(((.....))-----)........)))))))..)))..))))))))............ ( -23.60, z-score = -2.51, R) >droSec1.super_1 5602745 105 + 14215200 AUAAUCGUCGCCUUUCGAAUGUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUACAAUCGGU-----CUUAACUUGACUGUCGACGGAUAGCUCUCUUCAUUUCACAAUU (((((..(((.....)))..)))))...((((((((...(((.(((((((............-----.........)))))))..)))..))))))))............ ( -23.80, z-score = -2.09, R) >triCas2.ChLG7 1799805 91 + 17478683 ----UCGCUGUUUUUGAAUUCGUUCAUAAAGAGAGCAACUCCAUGACAGUG-UAAAUUAUAU--------------ACUGUCGACGGAUAGCUCUCUUCAUUUUCGAAUU ----............((((((...((.((((((((...(((.((((((((-((.....)))--------------)))))))..)))..)))))))).))...)))))) ( -30.90, z-score = -5.11, R) >dp4.chr3 418209 99 + 19779522 -----CGGCGCUUUUCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUACAAUCUGA------UUAACUUGACUGUCGACGGAUAGCUCUCUUUAUUUCACUAUU -----......................(((((((((...(((.(((((((............------........)))))))..)))..)))))))))........... ( -22.95, z-score = -2.72, R) >droPer1.super_2 605106 99 + 9036312 -----CGGCGCUUUUCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUACAAUCUGA------UUAACUUGACUGUCGACGGAUAGCUCUCUUUAUUUCACUAUU -----......................(((((((((...(((.(((((((............------........)))))))..)))..)))))))))........... ( -22.95, z-score = -2.72, R) >droSim1.chr2R_random 2148105 89 + 2996586 --------------UCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUGAUAUUAAAUGC-------UUUCUGGACUGUCGACGGACAGCUCUCUUUAUUCACUUAUU --------------..(((((.......((((((((...((((.((.(((..........))-------).)))))).((((....)))))))))))))))))....... ( -23.71, z-score = -3.35, R) >consensus ___AUCGUCGCUUUUCGAAUAUUAUACAAAGAGAGCAAUUCCAUGACAGUAUUACAAUCUGU______UUAACUUGACUGUCGACGGAUAGCUCUCUUCAUUUCACUAUU ............................((((((((...(((.(((((((..........................)))))))..)))..))))))))............ (-20.82 = -20.82 + -0.00)

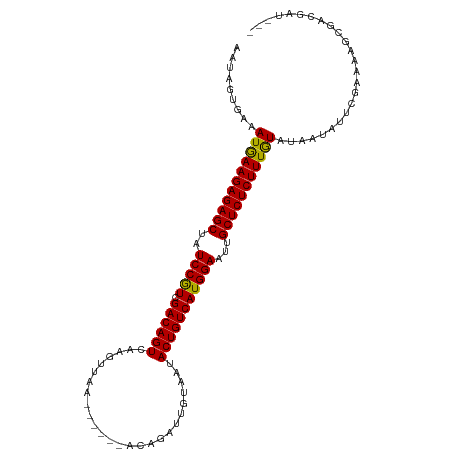
| Location | 8,057,642 – 8,057,747 |
|---|---|
| Length | 105 |
| Sequences | 15 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 78.20 |
| Shannon entropy | 0.50757 |
| G+C content | 0.36773 |
| Mean single sequence MFE | -31.25 |
| Consensus MFE | -27.31 |
| Energy contribution | -26.91 |
| Covariance contribution | -0.40 |
| Combinations/Pair | 1.14 |
| Mean z-score | -4.18 |
| Structure conservation index | 0.87 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.94 |
| SVM RNA-class probability | 0.999489 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 8057642 105 - 21146708 AAUUGUGAAAUGAAGAGAGCUAUCCGUCGACAGUCAAGUUAAG-----ACCGAUUGUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAACAUUCGAAAGGCGACGAUUAU ((((((...(..((((((((..(((((.((((((..((((...-----...)))).....)))))))))))...))))))))..).......((....))..)))))).. ( -32.50, z-score = -3.70, R) >droAna3.scaffold_13266 17315378 104 + 19884421 AAUAGUGAAAUGAAGAGAGCUAUCCGUCGACAGUCAAGUUAAA---ACACGGAUUGUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAACAUUCGAAAGGCGACGAU--- ....((...(..((((((((..(((((.((((((..((((...---.....)))).....)))))))))))...))))))))..)...))..((....)).......--- ( -30.80, z-score = -3.24, R) >droVir3.scaffold_12875 781368 101 + 20611582 AAUAGCGAAAUGAAGAGAGCUAUCCGUCGACAGUCAAGCUAUA----CA--GAUUGUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGAAAAGCGACGAC--- .....((..(..((((((((..(((((.(((((((((.((...----.)--).)))....)))))))))))...))))))))..).......(((.....)))))..--- ( -29.90, z-score = -3.26, R) >droMoj3.scaffold_6496 6376056 103 - 26866924 AAUAGCGAAAUGAAGAGAGCUAUCCGUCGACAGUCAAGUUAUA----UACAUAUUUUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGAAAAUCGACGAU--- .....((..(..((((((((..(((((.((((((...((((..----.........)))))))))))))))...))))))))..).......(((.....)))))..--- ( -30.00, z-score = -4.00, R) >droGri2.scaffold_15245 146894 103 + 18325388 AAUAGCGAAAUGAAGAGAGCUAUCCGUCGACAGUCAAGUUAUA----UACCGAUUGUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGAAAAGCGACGAU--- .....((..(..((((((((..(((((.((((((...((((((----.......)))))))))))))))))...))))))))..).......(((.....)))))..--- ( -32.30, z-score = -4.18, R) >apiMel3.Group1 18691508 90 - 25854376 ---------AUAAAGAGAGCUAUCCAUCGACAGUAUGG----------UUAUAAUA-GACACUGUCAUGGAGUUGCUCUCUUUGUAGACACUGCUAUGUUCAACGAUUAU ---------(((((((((((..(((((.((((((...(----------((......-))))))))))))))...))))))))))).((((......)))).......... ( -31.00, z-score = -4.15, R) >anoGam1.chr2L 16793180 97 - 48795086 AAUAUGAAAAUAAAGAGAGCUAUCCGUCGACAGUAGGG----------AUAUAAUU---CACUGUCAUGGAAUUGCUCUCUUUAUGUACAAUUCGAUAUUCAACGUGCAA .........(((((((((((..(((((.((((((..(.----------......).---.)))))))))))...)))))))))))((((...............)))).. ( -31.66, z-score = -4.97, R) >droWil1.scaffold_181141 2061049 110 + 5303230 AAUAGCGAAAUGAAGAGAGCUAUCCGUCGACAGUCCAGUUUAAUACAUCCGAAUUGUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGAAAAUCGACGGUUUU .....((..(..((((((((..(((((.((((((.((((((.........))))))....)))))))))))...))))))))..).......(((.....)))))..... ( -32.50, z-score = -3.86, R) >droEre2.scaffold_4845 4852621 105 + 22589142 AAUUGUGAAAUGAAGAGAGCUAUCCGUCGACAGUCAAGUUAAU-----ACCGAUUGUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAACAUUCGAAAGGCGACGAUUAG ((((((...(..((((((((..(((((.((((((.....((((-----....))))....)))))))))))...))))))))..).......((....))..)))))).. ( -32.70, z-score = -3.94, R) >droYak2.chr2R 12245557 105 + 21139217 AAUAGUGAAAUGAAGAGAGCUAUCCGUCGACAGUCAAGUUAAU-----ACCAAUUGUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGAAAGGCGACGAUUAG .........(..((((((((..(((((.((((((.....((((-----....))))....)))))))))))...))))))))..).((((..(((.....)))..)))). ( -30.20, z-score = -3.61, R) >droSec1.super_1 5602745 105 - 14215200 AAUUGUGAAAUGAAGAGAGCUAUCCGUCGACAGUCAAGUUAAG-----ACCGAUUGUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAACAUUCGAAAGGCGACGAUUAU ((((((...(..((((((((..(((((.((((((..((((...-----...)))).....)))))))))))...))))))))..).......((....))..)))))).. ( -32.50, z-score = -3.70, R) >triCas2.ChLG7 1799805 91 - 17478683 AAUUCGAAAAUGAAGAGAGCUAUCCGUCGACAGU--------------AUAUAAUUUA-CACUGUCAUGGAGUUGCUCUCUUUAUGAACGAAUUCAAAAACAGCGA---- ((((((...(((((((((((..(((((.((((((--------------.((.....))-.)))))))))))...)))))))))))...))))))............---- ( -36.20, z-score = -6.99, R) >dp4.chr3 418209 99 - 19779522 AAUAGUGAAAUAAAGAGAGCUAUCCGUCGACAGUCAAGUUAA------UCAGAUUGUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGAAAAGCGCCG----- .........(((((((((((..(((((.((((((..((((..------...)))).....)))))))))))...)))))))))))....................----- ( -28.90, z-score = -4.16, R) >droPer1.super_2 605106 99 - 9036312 AAUAGUGAAAUAAAGAGAGCUAUCCGUCGACAGUCAAGUUAA------UCAGAUUGUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGAAAAGCGCCG----- .........(((((((((((..(((((.((((((..((((..------...)))).....)))))))))))...)))))))))))....................----- ( -28.90, z-score = -4.16, R) >droSim1.chr2R_random 2148105 89 - 2996586 AAUAAGUGAAUAAAGAGAGCUGUCCGUCGACAGUCCAGAAA-------GCAUUUAAUAUCACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGA-------------- .........(((((((((((..(((((.((((((.......-------............)))))))))))...)))))))))))...........-------------- ( -28.71, z-score = -4.71, R) >consensus AAUAGUGAAAUGAAGAGAGCUAUCCGUCGACAGUCAAGUUAA______ACAGAUUGUAAUACUGUCAUGGAAUUGCUCUCUUUGUAUAAUAUUCGAAAAGCGACGAU___ .........(((((((((((..(((((.((((((..........................)))))))))))...)))))))))))......................... (-27.31 = -26.91 + -0.40)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:17:05 2011