
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 8,055,252 – 8,055,365 |
| Length | 113 |
| Max. P | 0.775146 |

| Location | 8,055,252 – 8,055,365 |
|---|---|
| Length | 113 |
| Sequences | 11 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 78.38 |
| Shannon entropy | 0.45831 |
| G+C content | 0.56333 |
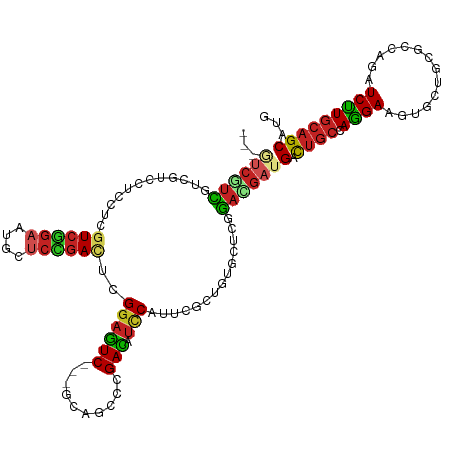
| Mean single sequence MFE | -43.23 |
| Consensus MFE | -16.40 |
| Energy contribution | -16.26 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.48 |
| Mean z-score | -2.36 |
| Structure conservation index | 0.38 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.65 |
| SVM RNA-class probability | 0.775146 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
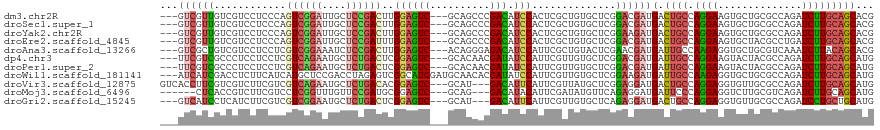
>dm3.chr2R 8055252 113 - 21146708 ---GUCGUUGUCGUCCUCCCAGUCGGAUUGCUCCGACUUGGAGUC---GCAGCCCGACAUCCACUCGCUGUGCUCGGACGAUGACUGCCAGGAAGUGCUGCGCCAGAUCUUGCAGCACG ---((.((..((((((..(.(((((((....))))))).)(((.(---(((((.............)))))))))))))))..)).))......((((((((........)))))))). ( -51.12, z-score = -3.33, R) >droSec1.super_1 5600360 113 - 14215200 ---GUCGUUGUCGUCCUCCCAGUCGGAUUGCUCCGACUUGGAGUC---GCAGCCCGACAUCCACUCGCUGUGCUCGGACGAUGACUGCCAGGAAGUGCUGCGCCAGAUCUUGCAGCACG ---((.((..((((((..(.(((((((....))))))).)(((.(---(((((.............)))))))))))))))..)).))......((((((((........)))))))). ( -51.12, z-score = -3.33, R) >droYak2.chr2R 12243185 113 + 21139217 ---GUCGUUGUCGUCCUCCCAGUCGGAUUGCUCCGACUUGGAGUC---GCAGCCCGACAUCCACUCGCUGUGCUCGGAAGAUGACUGCCAGGAAGUGCUGCGCCAGAUCUUGCAGCACG ---...((.((((((.(((.(((((((....)))))))..(((.(---(((((.............)))))))))))).)))))).))......((((((((........)))))))). ( -47.42, z-score = -2.49, R) >droEre2.scaffold_4845 4850247 113 + 22589142 ---GUCGUUGUCGUCCUCCCAGUCGGAUUGCUCCGAUUUGGAGUC---GCAGCCCGACAUCCACUCGCUGUGCUCGGACGAUGACUGCCAGGAAGUGCUACGCCUGAUCUUGCAGCACG ---((.((..((((((..(.(((((((....))))))).)(((.(---(((((.............)))))))))))))))..)).))......(((((..((........))))))). ( -42.62, z-score = -1.66, R) >droAna3.scaffold_13266 17313397 113 + 19884421 ---GUCGCUGUCGUCCUCCUCGUCGGAAAUCUCCGACUUGGAGUC---ACAGGGAGACAUCCAUUCGCUGUACUCGAACGAUGAUUGCCAAGAGGUGCUGCGUCAAAUCUUACAGCACG ---...((.((((((((((..((((((....))))))..))))..---((((.(((.......))).))))........)))))).))......((((((............)))))). ( -38.20, z-score = -1.67, R) >dp4.chr3 416393 113 - 19779522 ---UUCGUCGCCCUCCUCCUCGUCAGAAUGCUCUGACUCGGAGUC---GCACAACGAUAUCCAUUCGUUGUGCUCGGACGAUGAUUGCCAGGAAGUACUACGCCAGAUCUUGCAGCAUG ---.((((((.((..((((..((((((....))))))..))))..---(((((((((.......)))))))))..)).)))))).((((((((..............)))))..))).. ( -41.54, z-score = -3.20, R) >droPer1.super_2 603284 113 - 9036312 ---UUCGUCGCCCUCCUCCUCGUCAGAAUGCUCUGACUCGGAGUC---GCACAACGAUAUCCAUUCGUUGUGCUCGGACGAUGAUUGCCAGGAAGUACUACGCCAGAUCUUGCAGCAUG ---.((((((.((..((((..((((((....))))))..))))..---(((((((((.......)))))))))..)).)))))).((((((((..............)))))..))).. ( -41.54, z-score = -3.20, R) >droWil1.scaffold_181141 2059275 116 + 5303230 ---AUCAUCGACCUCUUCAUCAUGCUCCGACCUAGAGUCGGCAUCGAUGCAACACGAUAUCCAUUCGUUGUGCUCGGAAGAUGAUUGCCAAGAGGUGCUGCGCCAGAUCUUGCAGCAUG ---.......(((((((((((.....(((((.....)))))..((((.(((..((((.......))))..)))))))..)))))......))))))((((((........))))))... ( -37.90, z-score = -1.26, R) >droVir3.scaffold_12875 779545 113 + 20611582 GUCACCUUCGUCGUCUUCGUCGUCAGAAUGCUCUGACACGGAGUC---GCAU---GACAUUCAUUCGUUAUGCUCGGAGGAUGACUGCCAGGAGGUGUUGCGCCAGAUCUUGCAGCAUG ((((((((((..(.((((((.((((((....)))))))))))).)---((((---(((........))))))).)))))).)))).........((((((((........)))))))). ( -46.80, z-score = -2.97, R) >droMoj3.scaffold_6496 6374221 107 - 26866924 ------CUCACCGUCUUCGUCCUCGGUUUGUUCCGAUGCGGAGUC---GCAG---GACAUACAUUCGAUAUGUUCAGAGGAUGAUUCCCAGGAGGUCUUGCGUCAGAUCUUGCAGCAUG ------(((.......((((((((((......))..((((....)---)))(---(((((((....).))))))).)))))))).......)))((.(((((........))))).)). ( -30.84, z-score = 0.13, R) >droGri2.scaffold_15245 145099 110 + 18325388 ---GUCAUCCUCAUCUUCGUCGUCGGAAUGCUCUGACUCGGAGUC---GCAU---GACAUUCAUUCGUUGUGCUCAGAGGAUGACUGCCAGGAGGUGUUGCGCCAGAUCCUGCUGCAUG ---(((((((((..(((((..((((((....)))))).)))))..---(((.---.((........))..)))...)))))))))((((((((((((...))))...)))))..))).. ( -46.40, z-score = -2.93, R) >consensus ___GUCGUCGUCGUCCUCCUCGUCGGAAUGCUCCGACUCGGAGUC___GCAGCCCGACAUCCAUUCGCUGUGCUCGGACGAUGACUGCCAGGAAGUGCUGCGCCAGAUCUUGCAGCAUG ....(((((............((((((....))))))..((((((..........))).)))..............)))))((.((((.((((..............)))))))))).. (-16.40 = -16.26 + -0.14)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:17:03 2011