
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 7,486,376 – 7,486,478 |
| Length | 102 |
| Max. P | 0.998545 |

| Location | 7,486,376 – 7,486,478 |
|---|---|
| Length | 102 |
| Sequences | 7 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 71.92 |
| Shannon entropy | 0.50738 |
| G+C content | 0.44811 |
| Mean single sequence MFE | -27.03 |
| Consensus MFE | -15.59 |
| Energy contribution | -19.13 |
| Covariance contribution | 3.54 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.58 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.84 |
| SVM RNA-class probability | 0.995751 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
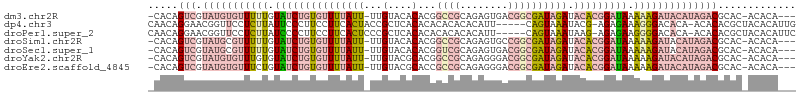
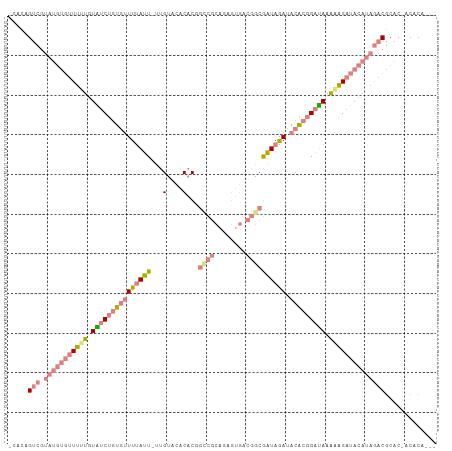
>dm3.chr2R 7486376 102 + 21146708 -CACAGUCGUAUGUGUUUUUGUAUCUGUGUUUUAUU-UUGUACACACGGCCGCAGAGUGACGGCGAUAGAUACACGGAUAAAAAGAUACAUAGACGCAC-ACACA--- -....(((.(((((((((((.(((((((((((((((-.(((....)))(((((.....).)))))))))).))))))))).))))))))))))))....-.....--- ( -34.10, z-score = -3.94, R) >dp4.chr3 4672954 101 + 19779522 CAACAGGAACGGUUCCUCUUAUUCCCUUCCUUCACUACCGCUCACACACACACACAUU-----CAGUAAAUACG-AGAGAAAGGGACACA-ACACACGCUACACAUUG ....((((.....)))).....((((((.(((.......(((................-----.))).......-.))).))))))....-................. ( -9.69, z-score = 0.44, R) >droPer1.super_2 4861309 101 + 9036312 CAACAGGAACGGUUCCUCUUAUCCCCUUCCUUCACUCCCGCUCACACACACACACAUU-----CAGUAAAUAAG-AGAGAAGGGGACACA-ACACACGCUACACAUUC ....((((.....))))....((((((((.(((......(((................-----.)))......)-)).))))))))....-................. ( -16.53, z-score = -1.55, R) >droSim1.chr2R 6011151 102 + 19596830 -CACAGUCGUAUGCGUUUUUGUAUCUGUGUUUUAUU-UUGUACACACGGCCGCAGAGUGCCGGCGAUAGAUACACGGAUAAAAAGAUACAUAGACGCAC-ACACA--- -....((.((.(((((((.(((((((...(((((((-(((((......(((((.....).))))......)))).))))))))))))))).))))))).-)))).--- ( -31.20, z-score = -2.83, R) >droSec1.super_1 5037112 102 + 14215200 -CACAGUCGUAUGCGUUUUUGUAUCUGUGUUUUAUU-UUGUACACACGGUCGCAGAGUGACGGCGAUAGAUACACGGAUAAAAAGAUACAUAGACGCAC-ACACA--- -....((.((.(((((((.(((((((...(((((((-(((((......(((((.(.....).)))))...)))).))))))))))))))).))))))).-)))).--- ( -30.20, z-score = -2.66, R) >droYak2.chr2R 11670546 102 - 21139217 -CACAGUCGUAUGUGUUUGUGUAUCUGUGUUUUAUU-UUGUACGCACGGCCGCAGAGGGACGGCGAUAGAUACACGGAUAAAAAGAUACAUAGACGCAC-ACACA--- -....(((.(((((((((...(((((((((.....(-((((.(((.((.((......)).)))))))))).)))))))))...))))))))))))....-.....--- ( -35.10, z-score = -3.60, R) >droEre2.scaffold_4845 4279616 102 - 22589142 -CACAGUCGUAUGUGUUUCUGUAUCUGUGUUUUAUU-UUGUACGCACCGCCGCAGAGGGACGGCGAUAGAUACACGGAUAAAAAGAUACAUAGACGCAC-ACACA--- -....(((.(((((((((...((((((((((((((.-.((....)).(((((........)))))))))).)))))))))...))))))))))))....-.....--- ( -32.40, z-score = -2.95, R) >consensus _CACAGUCGUAUGUGUUUUUGUAUCUGUGUUUUAUU_UUGUACACACGGCCGCAGAGUGACGGCGAUAGAUACACGGAUAAAAAGAUACAUAGACGCAC_ACACA___ .....(((.(((((((((((.(((((((((((((((...(....)...((((........)))))))))).))))))))).))))))))))))))............. (-15.59 = -19.13 + 3.54)

| Location | 7,486,376 – 7,486,478 |
|---|---|
| Length | 102 |
| Sequences | 7 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 71.92 |
| Shannon entropy | 0.50738 |
| G+C content | 0.44811 |
| Mean single sequence MFE | -31.13 |
| Consensus MFE | -13.91 |
| Energy contribution | -13.50 |
| Covariance contribution | -0.41 |
| Combinations/Pair | 1.45 |
| Mean z-score | -3.36 |
| Structure conservation index | 0.45 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.40 |
| SVM RNA-class probability | 0.998545 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
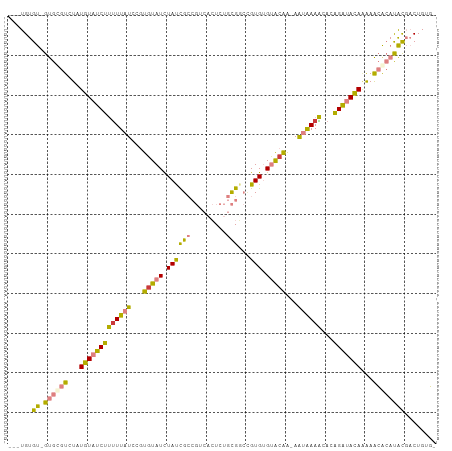
>dm3.chr2R 7486376 102 - 21146708 ---UGUGU-GUGCGUCUAUGUAUCUUUUUAUCCGUGUAUCUAUCGCCGUCACUCUGCGGCCGUGUGUACAA-AAUAAAACACAGAUACAAAAACACAUACGACUGUG- ---.((((-(((......(((((((((((((...(((((.(((.(((((......))))).))).))))).-.))))))...)))))))....))))))).......- ( -30.80, z-score = -3.43, R) >dp4.chr3 4672954 101 - 19779522 CAAUGUGUAGCGUGUGU-UGUGUCCCUUUCUCU-CGUAUUUACUG-----AAUGUGUGUGUGUGUGAGCGGUAGUGAAGGAAGGGAAUAAGAGGAACCGUUCCUGUUG (((((.(.((((..(.(-(.(.(((((((((.(-((...((((((-----.(..(......)..)...))))))))))))))))))...).)).)..))))).))))) ( -23.30, z-score = -1.30, R) >droPer1.super_2 4861309 101 - 9036312 GAAUGUGUAGCGUGUGU-UGUGUCCCCUUCUCU-CUUAUUUACUG-----AAUGUGUGUGUGUGUGAGCGGGAGUGAAGGAAGGGGAUAAGAGGAACCGUUCCUGUUG ......(.((((.((..-..(((((((((((((-((..(((((..-----.((......))..)))))..))))....)))))))))))......)))))).)..... ( -30.20, z-score = -3.06, R) >droSim1.chr2R 6011151 102 - 19596830 ---UGUGU-GUGCGUCUAUGUAUCUUUUUAUCCGUGUAUCUAUCGCCGGCACUCUGCGGCCGUGUGUACAA-AAUAAAACACAGAUACAAAAACGCAUACGACUGUG- ---..(((-((((((.(.(((((((((((((...(((((.(((.((((........)))).))).))))).-.))))))...))))))).).)))))))))......- ( -34.20, z-score = -3.76, R) >droSec1.super_1 5037112 102 - 14215200 ---UGUGU-GUGCGUCUAUGUAUCUUUUUAUCCGUGUAUCUAUCGCCGUCACUCUGCGACCGUGUGUACAA-AAUAAAACACAGAUACAAAAACGCAUACGACUGUG- ---..(((-((((((.(.(((((((((((((...(((((....(((.(((.......))).))).))))).-.))))))...))))))).).)))))))))......- ( -30.10, z-score = -3.32, R) >droYak2.chr2R 11670546 102 + 21139217 ---UGUGU-GUGCGUCUAUGUAUCUUUUUAUCCGUGUAUCUAUCGCCGUCCCUCUGCGGCCGUGCGUACAA-AAUAAAACACAGAUACACAAACACAUACGACUGUG- ---.((((-(((......(((((((((((((...(((((.(((.(((((......))))).))).))))).-.))))))...)))))))....))))))).......- ( -30.80, z-score = -3.41, R) >droEre2.scaffold_4845 4279616 102 + 22589142 ---UGUGU-GUGCGUCUAUGUAUCUUUUUAUCCGUGUAUCUAUCGCCGUCCCUCUGCGGCGGUGCGUACAA-AAUAAAACACAGAUACAGAAACACAUACGACUGUG- ---.((((-(((..(((..((((((((((((...(((((.(((((((((......))))))))).))))).-.))))))...)))))))))..))))))).......- ( -38.50, z-score = -5.23, R) >consensus ___UGUGU_GUGCGUCUAUGUAUCUUUUUAUCCGUGUAUCUAUCGCCGUCACUCUGCGGCCGUGUGUACAA_AAUAAAACACAGAUACAAAAACACAUACGACUGUG_ .........((((((...(((((((((((((...(((((.((((((.(.....).)))...))).)))))...))))))...)))))))...)))))).......... (-13.91 = -13.50 + -0.41)


Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:15:40 2011