
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 5,508,736 – 5,508,848 |
| Length | 112 |
| Max. P | 0.999294 |

| Location | 5,508,736 – 5,508,848 |
|---|---|
| Length | 112 |
| Sequences | 14 |
| Columns | 131 |
| Reading direction | forward |
| Mean pairwise identity | 83.39 |
| Shannon entropy | 0.36392 |
| G+C content | 0.44196 |
| Mean single sequence MFE | -37.25 |
| Consensus MFE | -29.40 |
| Energy contribution | -29.27 |
| Covariance contribution | -0.13 |
| Combinations/Pair | 1.19 |
| Mean z-score | -3.33 |
| Structure conservation index | 0.79 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.51 |
| SVM RNA-class probability | 0.998843 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
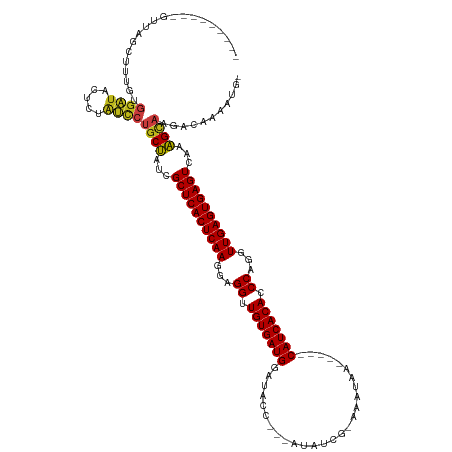
>dm3.chr2R 5508736 112 + 21146708 ---------GUUAGUUUCGUGGAUACUCUGUCCUGCUAUCGCUCACUCAAGGAGGUUGUGAUGGAUACC---AUAUCG-AAAUAA-----CAUCACACCCAGGUUGAGUGAGUCAAAGCAAGACAAAAUG- ---------...................((((.((((.(.((((((((((...((.(((((((......---......-......-----))))))).))...)))))))))).).)))).)))).....- ( -35.77, z-score = -2.97, R) >droGri2.scaffold_15112 722938 111 + 5172618 ---------GUUUUCUU-GUGGAUAUUCUAUCCUGCUAUCGCUCACUCAAGGAGGUUGUGAUGGAUACC---AAAUCG-AAAUAA-----CAUCACACCCAGGUUGAGUGAGUCAAAGCAAGACAAAAUG- ---------......((-(((((((...)))))((((.(.((((((((((...((.(((((((......---......-......-----))))))).))...)))))))))).).))))..))))....- ( -34.87, z-score = -3.34, R) >droMoj3.scaffold_6496 2595139 111 - 26866924 ----------UUAGCUUUGUGGAUAUUCUAUCCUGCUAUCGCUCACUCAAGGAGGUUGUGAUGGAUACC---GAAUCG-AAAUAA-----CAUCACACCCAGGUUGAGUGAGUCAAAGCAAGACAAAAUU- ----------...((((((.(((((...))))).......((((((((((...((.(((((((......---......-......-----))))))).))...))))))))))))))))...........- ( -35.97, z-score = -3.67, R) >droVir3.scaffold_12875 5789436 112 + 20611582 ---------GUUGGCUUAGUGGAUACUCUAUCCUGCUAUCGCUCACUCAAGGAGGUUGUGAUGGAUACC---AAAUCG-AACUAA-----CAUCACACCCAGGUUGAGUGAGUCAAAGCAAGACAAAAUG- ---------(((.((((((((((((...))))).)))...((((((((((...((.(((((((......---......-......-----))))))).))...))))))))))..))))..)))......- ( -34.97, z-score = -2.62, R) >droWil1.scaffold_180701 3433645 113 + 3904529 ---------UUGAACUUUGUGGAUAUUCUAUCCUGCUAUCGCUCACUCAAGGAGGUUGUGAUGGAUACC---AUAUUAUAAAUAA-----CAUCACACCCAGGUUGAGUGAGUCAAAGCAAGGCAAAAUG- ---------......((((((((((...)))))((((.(.((((((((((...((.(((((((......---.............-----))))))).))...)))))))))).).))))..)))))...- ( -36.01, z-score = -3.52, R) >droPer1.super_4 5979582 112 - 7162766 ---------UCCGGCUUUGUGGAUAUUCUAUCCUGCUAUCGCUCACUCAAGGAGGUUGUGAUGGAUACC---AUAUCG-AAAUAA-----CAUCACACCCAGGUUGAGUGAGUCAAAGCAAGACAAAAUG- ---------((..((((((.(((((...))))).......((((((((((...((.(((((((......---......-......-----))))))).))...))))))))))))))))..)).......- ( -36.27, z-score = -3.21, R) >dp4.chr3 14135682 112 + 19779522 ---------UCCGGCUUUGUGGAUAUUCUAUCCUGCUAUCGCUCACUCAAGGAGGUUGUGAUGGAUACC---AUAUCG-AAAUAA-----CAUCACACCCAGGUUGAGUGAGUCAAAGCAAGACAAAAUG- ---------((..((((((.(((((...))))).......((((((((((...((.(((((((......---......-......-----))))))).))...))))))))))))))))..)).......- ( -36.27, z-score = -3.21, R) >droAna3.scaffold_13266 12795462 113 + 19884421 ---------GUUAGUCUUGUGGAUACUCUGUCCUGCUAUCGCUCACUCAAGGAGGUUGUGAUGGAUACC---AUAUCG-AAAUAA-----CAUCACACCCAGGUUGAGUGAGUCAAAGCAAGACAAAAAGG ---------....(((((((((((.....)))).......((((((((((...((.(((((((......---......-......-----))))))).))...))))))))))....)))))))....... ( -38.67, z-score = -3.49, R) >droEre2.scaffold_4929 17792171 112 - 26641161 ---------GUUAGUUUCGUGGAUACUCUGUCCUGCUAUCGCUCACUCAAGGAGGUUGUGAUGGAUACC---AUAUCG-AAAUAA-----CAUCACACCCAGGUUGAGUGAGUCAAAGCAAGACAAAAUG- ---------...................((((.((((.(.((((((((((...((.(((((((......---......-......-----))))))).))...)))))))))).).)))).)))).....- ( -35.77, z-score = -2.97, R) >droYak2.chr2L 18140626 112 + 22324452 ---------GUUAGUUUCGUGGAUACUCUGUCCUGCUAUCGCUCACUCAAGGAGGUUGUGAUGGAUACC---AUAUCG-AAAUAA-----CAUCACACCCAGGUUGAGUGAGUCAAAGCAAGACAAAAUG- ---------...................((((.((((.(.((((((((((...((.(((((((......---......-......-----))))))).))...)))))))))).).)))).)))).....- ( -35.77, z-score = -2.97, R) >droSec1.super_1 3126036 112 + 14215200 ---------GUUAGCUUCGUGGAUACUCUGUCCUGCUAUCGCUCACUCAAGGAGGUUGUGAUGGAUACC---AUAUCG-AAAUAA-----CAUCACACCCAGGUUGAGUGAGUCAAAGCAAGACAAAAUG- ---------...................((((.((((.(.((((((((((...((.(((((((......---......-......-----))))))).))...)))))))))).).)))).)))).....- ( -35.77, z-score = -2.89, R) >droSim1.chr2R 4140604 112 + 19596830 ---------GUUAGCUUUGUGGAUACUCUGUCCUGCUAUCGCUCACUCAAGGAGGUUGUGAUGGAUACC---AUAUCG-AAAUAA-----CAUCACACCCAGGUUGAGUGAGUCAAAGCAAGACAAAAUG- ---------(((.((((((.((((.....)))).......((((((((((...((.(((((((......---......-......-----))))))).))...))))))))))))))))..)))......- ( -36.17, z-score = -2.97, R) >apiMel3.Group1 5097040 120 + 25854376 CUCCUUUUCAUGGAUAUGGUCGAUAAAACAGUC-GCGUUCGCUCACUCAAAAAGGUUGUGAUGACCGCUAGGGCCUGAUACACAGGCAAGCAUCACACCCAGGUUGAGUGAGUCCACGUGA---------- .(((.......))).................((-((((..((((((((((...((.(((((((.((....))(((((.....)))))...))))))).))...))))))))))..))))))---------- ( -47.70, z-score = -4.20, R) >triCas2.ChLG8 1024728 100 - 15773733 -------------AAAUG-UCGGUGAUAUCCAC-CCGGUCGCUCACUCAAGGAGGUUGUGAUG---------GUACAGCAAACA------CAUCACACCCAGGUUGAGUGAGUCACCGAGAUAGAUAGUG- -------------...((-((((((.....)))-)((((.((((((((((...((.(((((((---------((.......)).------))))))).))...)))))))))).)))).)))).......- ( -41.50, z-score = -4.53, R) >consensus _________GUUAGCUUUGUGGAUACUCUAUCCUGCUAUCGCUCACUCAAGGAGGUUGUGAUGGAUACC___AUAUCG_AAAUAA_____CAUCACACCCAGGUUGAGUGAGUCAAAGCAAGACAAAAUG_ ....................((((.....))))((((...((((((((((...((.(((((((...........................))))))).))...))))))))))...))))........... (-29.40 = -29.27 + -0.13)

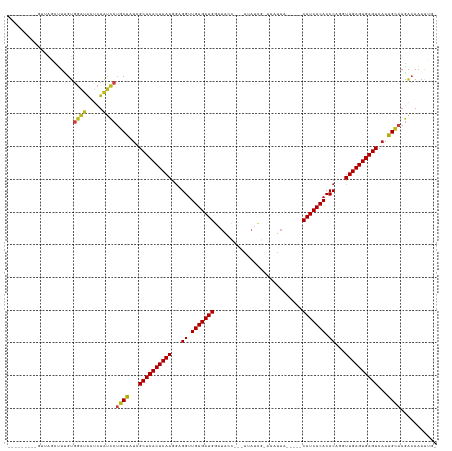
| Location | 5,508,736 – 5,508,848 |
|---|---|
| Length | 112 |
| Sequences | 14 |
| Columns | 131 |
| Reading direction | reverse |
| Mean pairwise identity | 83.39 |
| Shannon entropy | 0.36392 |
| G+C content | 0.44196 |
| Mean single sequence MFE | -43.32 |
| Consensus MFE | -37.37 |
| Energy contribution | -37.22 |
| Covariance contribution | -0.15 |
| Combinations/Pair | 1.24 |
| Mean z-score | -5.30 |
| Structure conservation index | 0.86 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.77 |
| SVM RNA-class probability | 0.999294 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 5508736 112 - 21146708 -CAUUUUGUCUUGCUUUGACUCACUCAACCUGGGUGUGAUG-----UUAUUU-CGAUAU---GGUAUCCAUCACAACCUCCUUGAGUGAGCGAUAGCAGGACAGAGUAUCCACGAAACUAAC--------- -.((((((((((((((((.(((((((((...((.(((((((-----.((((.-......---))))..))))))).))...)))))))))))).)))))))))))))...............--------- ( -44.90, z-score = -5.99, R) >droGri2.scaffold_15112 722938 111 - 5172618 -CAUUUUGUCUUGCUUUGACUCACUCAACCUGGGUGUGAUG-----UUAUUU-CGAUUU---GGUAUCCAUCACAACCUCCUUGAGUGAGCGAUAGCAGGAUAGAAUAUCCAC-AAGAAAAC--------- -.((((((((((((((((.(((((((((...((.(((((((-----.((((.-......---))))..))))))).))...)))))))))))).)))))))))))))......-........--------- ( -43.00, z-score = -5.90, R) >droMoj3.scaffold_6496 2595139 111 + 26866924 -AAUUUUGUCUUGCUUUGACUCACUCAACCUGGGUGUGAUG-----UUAUUU-CGAUUC---GGUAUCCAUCACAACCUCCUUGAGUGAGCGAUAGCAGGAUAGAAUAUCCACAAAGCUAA---------- -.((((((((((((((((.(((((((((...((.(((((((-----.....(-((...)---))....))))))).))...)))))))))))).)))))))))))))..............---------- ( -42.70, z-score = -5.88, R) >droVir3.scaffold_12875 5789436 112 - 20611582 -CAUUUUGUCUUGCUUUGACUCACUCAACCUGGGUGUGAUG-----UUAGUU-CGAUUU---GGUAUCCAUCACAACCUCCUUGAGUGAGCGAUAGCAGGAUAGAGUAUCCACUAAGCCAAC--------- -.((((((((((((((((.(((((((((...((.(((((((-----......-.(((..---...)))))))))).))...)))))))))))).)))))))))))))...............--------- ( -41.51, z-score = -4.55, R) >droWil1.scaffold_180701 3433645 113 - 3904529 -CAUUUUGCCUUGCUUUGACUCACUCAACCUGGGUGUGAUG-----UUAUUUAUAAUAU---GGUAUCCAUCACAACCUCCUUGAGUGAGCGAUAGCAGGAUAGAAUAUCCACAAAGUUCAA--------- -.((((((.(((((((((.(((((((((...((.(((((((-----.((((........---))))..))))))).))...)))))))))))).)))))).))))))...............--------- ( -38.40, z-score = -4.59, R) >droPer1.super_4 5979582 112 + 7162766 -CAUUUUGUCUUGCUUUGACUCACUCAACCUGGGUGUGAUG-----UUAUUU-CGAUAU---GGUAUCCAUCACAACCUCCUUGAGUGAGCGAUAGCAGGAUAGAAUAUCCACAAAGCCGGA--------- -.((((((((((((((((.(((((((((...((.(((((((-----.((((.-......---))))..))))))).))...)))))))))))).))))))))))))).(((........)))--------- ( -44.40, z-score = -5.47, R) >dp4.chr3 14135682 112 - 19779522 -CAUUUUGUCUUGCUUUGACUCACUCAACCUGGGUGUGAUG-----UUAUUU-CGAUAU---GGUAUCCAUCACAACCUCCUUGAGUGAGCGAUAGCAGGAUAGAAUAUCCACAAAGCCGGA--------- -.((((((((((((((((.(((((((((...((.(((((((-----.((((.-......---))))..))))))).))...)))))))))))).))))))))))))).(((........)))--------- ( -44.40, z-score = -5.47, R) >droAna3.scaffold_13266 12795462 113 - 19884421 CCUUUUUGUCUUGCUUUGACUCACUCAACCUGGGUGUGAUG-----UUAUUU-CGAUAU---GGUAUCCAUCACAACCUCCUUGAGUGAGCGAUAGCAGGACAGAGUAUCCACAAGACUAAC--------- ...(((((((((((((((.(((((((((...((.(((((((-----.((((.-......---))))..))))))).))...)))))))))))).))))))))))))................--------- ( -43.30, z-score = -5.29, R) >droEre2.scaffold_4929 17792171 112 + 26641161 -CAUUUUGUCUUGCUUUGACUCACUCAACCUGGGUGUGAUG-----UUAUUU-CGAUAU---GGUAUCCAUCACAACCUCCUUGAGUGAGCGAUAGCAGGACAGAGUAUCCACGAAACUAAC--------- -.((((((((((((((((.(((((((((...((.(((((((-----.((((.-......---))))..))))))).))...)))))))))))).)))))))))))))...............--------- ( -44.90, z-score = -5.99, R) >droYak2.chr2L 18140626 112 - 22324452 -CAUUUUGUCUUGCUUUGACUCACUCAACCUGGGUGUGAUG-----UUAUUU-CGAUAU---GGUAUCCAUCACAACCUCCUUGAGUGAGCGAUAGCAGGACAGAGUAUCCACGAAACUAAC--------- -.((((((((((((((((.(((((((((...((.(((((((-----.((((.-......---))))..))))))).))...)))))))))))).)))))))))))))...............--------- ( -44.90, z-score = -5.99, R) >droSec1.super_1 3126036 112 - 14215200 -CAUUUUGUCUUGCUUUGACUCACUCAACCUGGGUGUGAUG-----UUAUUU-CGAUAU---GGUAUCCAUCACAACCUCCUUGAGUGAGCGAUAGCAGGACAGAGUAUCCACGAAGCUAAC--------- -.((((((((((((((((.(((((((((...((.(((((((-----.((((.-......---))))..))))))).))...)))))))))))).)))))))))))))...............--------- ( -44.90, z-score = -5.65, R) >droSim1.chr2R 4140604 112 - 19596830 -CAUUUUGUCUUGCUUUGACUCACUCAACCUGGGUGUGAUG-----UUAUUU-CGAUAU---GGUAUCCAUCACAACCUCCUUGAGUGAGCGAUAGCAGGACAGAGUAUCCACAAAGCUAAC--------- -.((((((((((((((((.(((((((((...((.(((((((-----.((((.-......---))))..))))))).))...)))))))))))).)))))))))))))...............--------- ( -44.90, z-score = -5.83, R) >apiMel3.Group1 5097040 120 - 25854376 ----------UCACGUGGACUCACUCAACCUGGGUGUGAUGCUUGCCUGUGUAUCAGGCCCUAGCGGUCAUCACAACCUUUUUGAGUGAGCGAACGC-GACUGUUUUAUCGACCAUAUCCAUGAAAAGGAG ----------...(((((((((((((((...((((((((((...(((((.....)))))((....)).)))))).))))..)))))))))......(-((........)))......))))))........ ( -42.00, z-score = -2.84, R) >triCas2.ChLG8 1024728 100 + 15773733 -CACUAUCUAUCUCGGUGACUCACUCAACCUGGGUGUGAUG------UGUUUGCUGUAC---------CAUCACAACCUCCUUGAGUGAGCGACCGG-GUGGAUAUCACCGA-CAUUU------------- -...((((((((.((((..(((((((((...((.(((((((------.((.......))---------))))))).))...)))))))))..)))))-))))))).......-.....------------- ( -42.30, z-score = -4.71, R) >consensus _CAUUUUGUCUUGCUUUGACUCACUCAACCUGGGUGUGAUG_____UUAUUU_CGAUAU___GGUAUCCAUCACAACCUCCUUGAGUGAGCGAUAGCAGGAUAGAGUAUCCACAAAGCUAAC_________ ...(((((((((((((((.(((((((((...((.(((((((...........................))))))).))...)))))))))))).))))))))))))......................... (-37.37 = -37.22 + -0.15)


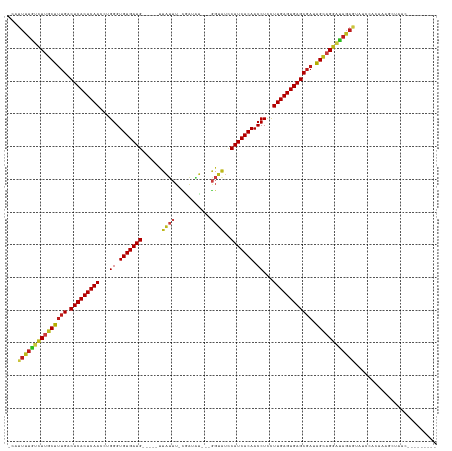
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:10:36 2011