
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 5,183,074 – 5,183,200 |
| Length | 126 |
| Max. P | 0.874771 |

| Location | 5,183,074 – 5,183,200 |
|---|---|
| Length | 126 |
| Sequences | 11 |
| Columns | 132 |
| Reading direction | forward |
| Mean pairwise identity | 79.54 |
| Shannon entropy | 0.44290 |
| G+C content | 0.65387 |
| Mean single sequence MFE | -60.30 |
| Consensus MFE | -33.34 |
| Energy contribution | -32.79 |
| Covariance contribution | -0.55 |
| Combinations/Pair | 1.45 |
| Mean z-score | -1.39 |
| Structure conservation index | 0.55 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.540865 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
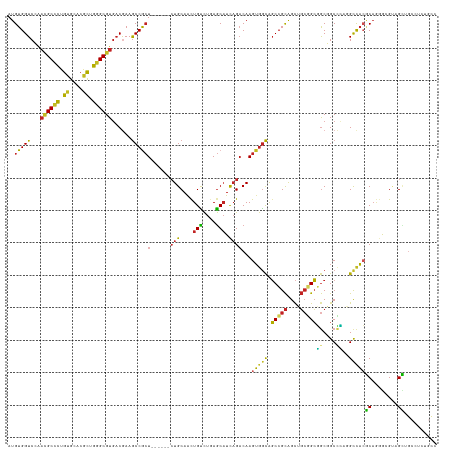
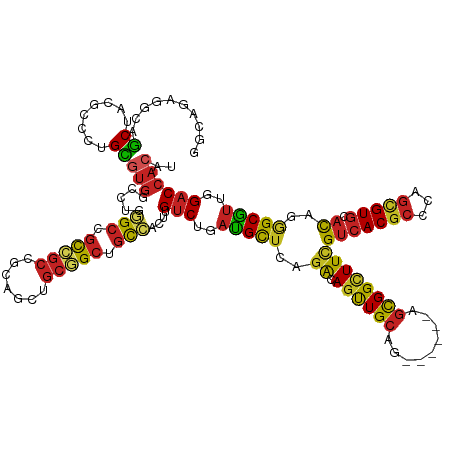
>dm3.chr2R 5183074 126 + 21146708 AUGUGGUCCAACGCCCUGUGCACGCUGGGCGUGACGAAGCCGCU------CUGCAACUGUCUGAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCGGCCCAGGCCACGCAGGGCGUAGCUGCCUCUGCC ..((((((..((((((.((....)).))))))......((((((------.(((.((((((((.....))))).))).)))(((((....)))))))))))...))))))((((((((....)))).)))). ( -67.70, z-score = -2.12, R) >droSim1.chr2R 3840732 126 + 19596830 AUGUGGUCCAACGCCCUGUGCACGCUGGGCGUGACGAAGCCGCU------CUGCAACUGUCUGAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCGGCCCAGGCCACGCAGGGCGUAGCUGCCUCUGCC ..((((((..((((((.((....)).))))))......((((((------.(((.((((((((.....))))).))).)))(((((....)))))))))))...))))))((((((((....)))).)))). ( -67.70, z-score = -2.12, R) >droSec1.super_1 2815320 126 + 14215200 AUGUGGUCCAACGCCCUGUGCACGCUGGGCGUGACGAAGCCGCU------CUGCAACUGUCUGAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCGGCCCAGGCCACGCAGGGCGUAGCUGCCUCUGCC ..((((((..((((((.((....)).))))))......((((((------.(((.((((((((.....))))).))).)))(((((....)))))))))))...))))))((((((((....)))).)))). ( -67.70, z-score = -2.12, R) >droYak2.chr2L 17838322 126 + 22324452 AUGUGGUCCAACGCCCUGUGCACGCUGGGCGUGACGAAGCCGCU------CUGCAACUGUCUGAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCGGCCCAGGCUACGCAGGGCGUAGCUGCCUCUGCC ..((((((..((((((.((....)).))))))......((((((------.(((.((((((((.....))))).))).)))(((((....)))))))))))...))))))((((((((....)))).)))). ( -65.40, z-score = -1.69, R) >droEre2.scaffold_4929 9166685 126 - 26641161 AUGUGGUCCAACGCCCUGUGCACGCUGGGCGUGACGAAGCCGCU------CUGCAACUGUCUGAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCGGCCCAGGCCACGCAGGGCGUAGCUGCCUCUGCC ..((((((..((((((.((....)).))))))......((((((------.(((.((((((((.....))))).))).)))(((((....)))))))))))...))))))((((((((....)))).)))). ( -67.70, z-score = -2.12, R) >droAna3.scaffold_13266 11494946 126 - 19884421 AUGUGGUCCAACGCCCUGUGCACCCUGGGCGUGACGAAGCCGCU------CUGCAACUGUCUGAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCGGCCCAGGCCACGCAGGGCGUUGCUGCCUCUGCU ..(.(((.((((((((((((...(((((((((......))((((------.(((.((((((((.....))))).))).)))(((((....))))))))))))))))...))))))))))))..))).).... ( -69.70, z-score = -2.90, R) >dp4.chr3 9251803 117 + 19779522 AUGUGGUCGAACGCCCUGUGCACGCUGGGCGUGACGAAGCCGCU------CUGCAACUGCCUCAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCUGCCCAGGUCACACAGGGGGGCGUG--------- ......(((.((((((.((....)).))))))..))).(((.((------(((....(((....)))...(((..(.(((((((((.......))))))))))..)))...))))).)))...--------- ( -55.90, z-score = -0.56, R) >droPer1.super_4 4574400 117 + 7162766 AUGUGGUCGAACGCCCUGUGCACGCUGGGCGUGACGAAGCCGCU------CUGCAACUGCCUCAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCUGCCCAGGUCACACAGGGGGGCGUG--------- ......(((.((((((.((....)).))))))..))).(((.((------(((....(((....)))...(((..(.(((((((((.......))))))))))..)))...))))).)))...--------- ( -55.90, z-score = -0.56, R) >droGri2.scaffold_15245 8564812 126 - 18325388 AUGUGGUCCAAUGCUCUGUGCACACUGGGCGUGACAAAGCCACU------CUGCAGUUGCCUAAGCAUCAGACAAGUGGCGGCCGCAGCAGCCGCCGCUGCGGCAGCCAACCAAAACGCUGGCGCAGCCAUC .....(((..(((((..(.(((.((((((.(((.......))).------)).))))))))..)))))..)))....((((((.......))))))((((((.((((..........)))).)))))).... ( -52.20, z-score = -0.55, R) >droVir3.scaffold_12875 1225370 118 + 20611582 AUGUGGUCCAAUGCUCUGUGCACACUGGGCGUGACAAAGCCACU------GUGCAACUGCCUGAGCAUCAGACAAGUGGCGGCAGCAGCCGCUGCUGCUGCGGCAGCCAACCAAAAUGCUGGCG-------- .....(((..((((((..((((((...(((........)))..)------))))).......))))))..)))..((.((((((((((...)))))))))).)).((((.(......).)))).-------- ( -52.20, z-score = -1.06, R) >anoGam1.chr2L 36132224 131 - 48795086 AUGUGGUCUGCAGCGUUAUGUACAUUGGGCGUUUCGAAACCGAUUUGGGGCUGCAACUGUUUGAACUUUCGCCAAGGAAUGUCCGUGGUACGUGGUACUGCUACUGCCCCACCCAA-GCGGUUGCCUCAGCC .((.(((..((((..((((((((..((((((((((.....(((...((..(.((....))..)..)).)))....))))))))))..))))))))..)))).(((((.........-))))).))).))... ( -41.20, z-score = 0.49, R) >consensus AUGUGGUCCAACGCCCUGUGCACGCUGGGCGUGACGAAGCCGCU______CUGCAACUGUCUGAGCAUCAGACAAGUGGCAGCCGCAGCUGCGGCGGCGGCCCAGGCCACGCAGGGCGUAGCUGCCUCUGCC ..(((((...((((((.((....)).))))))......((((((.......(((..........))).......)))))).(((((....)))))..........)))))((........)).......... (-33.34 = -32.79 + -0.55)


| Location | 5,183,074 – 5,183,200 |
|---|---|
| Length | 126 |
| Sequences | 11 |
| Columns | 132 |
| Reading direction | reverse |
| Mean pairwise identity | 79.54 |
| Shannon entropy | 0.44290 |
| G+C content | 0.65387 |
| Mean single sequence MFE | -60.80 |
| Consensus MFE | -33.87 |
| Energy contribution | -33.42 |
| Covariance contribution | -0.45 |
| Combinations/Pair | 1.47 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.56 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.02 |
| SVM RNA-class probability | 0.874771 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
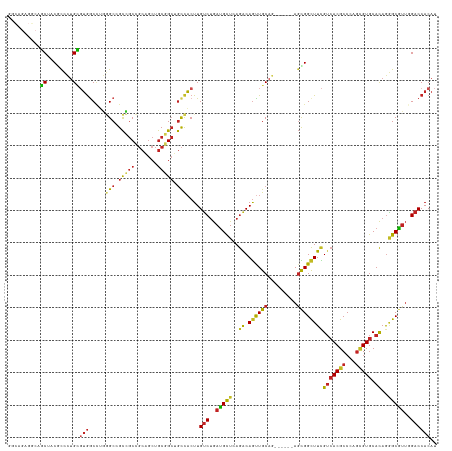
>dm3.chr2R 5183074 126 - 21146708 GGCAGAGGCAGCUACGCCCUGCGUGGCCUGGGCCGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUCAGACAGUUGCAG------AGCGGCUUCGUCACGCCCAGCGUGCACAGGGCGUUGGACCACAU .((((.(((......)))))))((((((.(((((((((((((....)))))(((.(((.(((((.....)))))))).))))------.)))))))....((((((..........)))))).)).)))).. ( -64.80, z-score = -1.55, R) >droSim1.chr2R 3840732 126 - 19596830 GGCAGAGGCAGCUACGCCCUGCGUGGCCUGGGCCGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUCAGACAGUUGCAG------AGCGGCUUCGUCACGCCCAGCGUGCACAGGGCGUUGGACCACAU .((((.(((......)))))))((((((.(((((((((((((....)))))(((.(((.(((((.....)))))))).))))------.)))))))....((((((..........)))))).)).)))).. ( -64.80, z-score = -1.55, R) >droSec1.super_1 2815320 126 - 14215200 GGCAGAGGCAGCUACGCCCUGCGUGGCCUGGGCCGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUCAGACAGUUGCAG------AGCGGCUUCGUCACGCCCAGCGUGCACAGGGCGUUGGACCACAU .((((.(((......)))))))((((((.(((((((((((((....)))))(((.(((.(((((.....)))))))).))))------.)))))))....((((((..........)))))).)).)))).. ( -64.80, z-score = -1.55, R) >droYak2.chr2L 17838322 126 - 22324452 GGCAGAGGCAGCUACGCCCUGCGUAGCCUGGGCCGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUCAGACAGUUGCAG------AGCGGCUUCGUCACGCCCAGCGUGCACAGGGCGUUGGACCACAU ......(((....((((((((.(((..((((((....(.((.((((((..((((.(((.(((((.....)))))))).))))------.)))))).)).)..))))))..))).))))))))..).)).... ( -62.30, z-score = -1.36, R) >droEre2.scaffold_4929 9166685 126 + 26641161 GGCAGAGGCAGCUACGCCCUGCGUGGCCUGGGCCGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUCAGACAGUUGCAG------AGCGGCUUCGUCACGCCCAGCGUGCACAGGGCGUUGGACCACAU .((((.(((......)))))))((((((.(((((((((((((....)))))(((.(((.(((((.....)))))))).))))------.)))))))....((((((..........)))))).)).)))).. ( -64.80, z-score = -1.55, R) >droAna3.scaffold_13266 11494946 126 + 19884421 AGCAGAGGCAGCAACGCCCUGCGUGGCCUGGGCCGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUCAGACAGUUGCAG------AGCGGCUUCGUCACGCCCAGGGUGCACAGGGCGUUGGACCACAU ......((...((((((((((.(((.(((((((....(.((.((((((..((((.(((.(((((.....)))))))).))))------.)))))).)).)..))))))).))).))))))))))..)).... ( -70.10, z-score = -3.07, R) >dp4.chr3 9251803 117 - 19779522 ---------CACGCCCCCCUGUGUGACCUGGGCAGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUGAGGCAGUUGCAG------AGCGGCUUCGUCACGCCCAGCGUGCACAGGGCGUUCGACCACAU ---------..((..(((((((((.((((((((.((.(((((..(((((((((((.(..((.....)).).)))))))))))------.)))))...))...)))))).)))))))))).)..))....... ( -59.20, z-score = -2.38, R) >droPer1.super_4 4574400 117 - 7162766 ---------CACGCCCCCCUGUGUGACCUGGGCAGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUGAGGCAGUUGCAG------AGCGGCUUCGUCACGCCCAGCGUGCACAGGGCGUUCGACCACAU ---------..((..(((((((((.((((((((.((.(((((..(((((((((((.(..((.....)).).)))))))))))------.)))))...))...)))))).)))))))))).)..))....... ( -59.20, z-score = -2.38, R) >droGri2.scaffold_15245 8564812 126 + 18325388 GAUGGCUGCGCCAGCGUUUUGGUUGGCUGCCGCAGCGGCGGCUGCUGCGGCCGCCACUUGUCUGAUGCUUAGGCAACUGCAG------AGUGGCUUUGUCACGCCCAGUGUGCACAGAGCAUUGGACCACAU ..((((...((((((......)))))).((((((((((...)))))))))).))))...(((..((((((.((((((((...------.(((((...)))))...)))).))).).))))))..)))..... ( -61.60, z-score = -1.87, R) >droVir3.scaffold_12875 1225370 118 - 20611582 --------CGCCAGCAUUUUGGUUGGCUGCCGCAGCAGCAGCGGCUGCUGCCGCCACUUGUCUGAUGCUCAGGCAGUUGCAC------AGUGGCUUUGUCACGCCCAGUGUGCACAGAGCAUUGGACCACAU --------.((((((......)))))).((.((((((((....)))))))).)).....(((..((((((.(.....(((((------(.((((........).))).))))))).))))))..)))..... ( -55.10, z-score = -1.79, R) >anoGam1.chr2L 36132224 131 + 48795086 GGCUGAGGCAACCGC-UUGGGUGGGGCAGUAGCAGUACCACGUACCACGGACAUUCCUUGGCGAAAGUUCAAACAGUUGCAGCCCCAAAUCGGUUUCGAAACGCCCAAUGUACAUAACGCUGCAGACCACAU (((((.(((......-(((((((((((.(((((.((((...))))...((((.(((......))).)))).....))))).)))))...(((....)))...)))))).((.....))))).))).)).... ( -42.10, z-score = -0.83, R) >consensus GGCAGAGGCAGCUACGCCCUGCGUGGCCUGGGCCGCCGCCGCAGCUGCGGCUGCCACUUGUCUGAUGCUCAGACAGUUGCAG______AGCGGCUUCGUCACGCCCAGCGUGCACAGGGCGUUGGACCACAU ..........((........))(((.....(((.(((((.......))))).)))....(((..(((((..((.((((((.........))))))))(((((((...))))).))..)))))..)))))).. (-33.87 = -33.42 + -0.45)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:09:56 2011