| Sequence ID | dm3.chr2R |
|---|---|
| Location | 5,072,978 – 5,073,079 |
| Length | 101 |
| Max. P | 0.920320 |

| Location | 5,072,978 – 5,073,079 |
|---|---|
| Length | 101 |
| Sequences | 9 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 64.79 |
| Shannon entropy | 0.67829 |
| G+C content | 0.59977 |
| Mean single sequence MFE | -43.47 |
| Consensus MFE | -14.68 |
| Energy contribution | -14.07 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.65 |
| Mean z-score | -1.92 |
| Structure conservation index | 0.34 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.28 |
| SVM RNA-class probability | 0.920320 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 5072978 101 + 21146708 GUGAACCGGAGCCUGAGCUCACGGGUCGGUGUCUGUGCUCCACGGGCAG---GUGACCGGACUGGGUGUUCGGAUGCUG---------AAGAUCCGUGGCCAGGUACUGGCAG .....((((.(((((.((.((((((((..((((((((...))))))))(---(((.((((((.....)))))).)))).---------..))))))))))))))).))))... ( -51.10, z-score = -3.01, R) >droAna3.scaffold_13266 6119629 86 + 19884421 ------GGGAGCCGGCGCUCAAGGCUUCCGGCCUGUAUUCCACAGG------------GGAUUGGGUGGCAGGAUCCUC---------GAGAUCCUUGGCCAGGUAUUGGCAG ------(((((((.........))))))).(((.(((((((.(((.------------...)))))((((((((((...---------..))))))..))))))))).))).. ( -33.80, z-score = -0.11, R) >droEre2.scaffold_4929 9058093 101 - 26641161 GUGAGCCGGAGCCUGAGCUCACGGGUCUGUGCCGGUGCUCCACGGGCAG---GUGACCGACCUGGGUGUGCGACUGCUG---------AAUAGCCGUGGACAGGUACUGGCAG ..((.(((((((....)))).))).))..((((((((((((((((.(((---((.....)))))(((........))).---------.....)))))))...))))))))). ( -47.10, z-score = -1.41, R) >droYak2.chr2L 17728304 101 + 22324452 GUGAGCCGGAGCCUGAGCUCACGGGUCGGUGCUUGUGCUCCACGGGCAG---GCGACCGGACUGGGUGUUCGAAUGCUG---------AAUAUCUGUGGCCAGGUACUGGCAG ....(((((.(((((.((.(((((..((((((((((.(.....).))))---)).))))..))(((((((((.....))---------))))))))))))))))).))))).. ( -50.00, z-score = -3.01, R) >droSec1.super_1 2705292 101 + 14215200 GUGAGCCAGAGCCUGAGCUCACAGGUCGGUGCCUGUGCUCCACGGGCAG---GUGACCGGACUGGGUGUUCGGAUGCUG---------AAGAUCUGUGGCCAGGUACUGGCAG ....(((((.(((((.((.((((((((..((((((((...))))))))(---(((.((((((.....)))))).)))).---------..))))))))))))))).))))).. ( -57.40, z-score = -4.59, R) >droSim1.chr2R 3735933 101 + 19596830 GUGAACCGGAGCCUGAGCUCACGGGUCGAUGCCUGUGCUCCACGGGCAG---GUGACCGGACUGGGUGUUCGGAUGCUG---------AAGAUCCGUGGCCAGGUACUGGCAG .....((((.(((((.((.((((((((..((((((((...))))))))(---(((.((((((.....)))))).)))).---------..))))))))))))))).))))... ( -53.90, z-score = -3.95, R) >droWil1.scaffold_181141 5266698 107 + 5303230 ------GGGAAAUGGCACUCAAGGGCUGAUGUUUGUGUUCCACGGGCAACGUGUCGUCAGAUGAGCAGUGGGUGGACUGUGACCUUCUGACCUUAUUGGUCAGAUACUGACAG ------.......((((((((.....)))((((((((...))))))))..)))))(((((....(((((......))))).....(((((((.....)))))))..))))).. ( -36.40, z-score = -0.89, R) >droPer1.super_4 4230208 104 - 7162766 ------GGGAAAUGGCACUGAGGGGAUGAUGUUUAUGCUCCACGGGCAACGUGUCGUCAGAAAAGUUGGUGGUG--CUG-GUCACGAUUACCUUUUUGGCCAGGUAGUGGCAA ------......((.(((((..(((((((..(...((((.....))))..)..))))).((((((.((((.(((--...-..))).)))).))))))..))...))))).)). ( -31.60, z-score = -0.26, R) >dp4.chr3 14683306 104 + 19779522 ------GGGAAAUGGCACUGAGGGGAUGAUGUUUAUGCUCCACGGGUAACGUGUCGUCAGAAAAGUUGGUGGUG--CUG-GUCACGAUUACCUUUUUGGCCAGGUAGUGGCAA ------......((.(((((.(((.((((...)))).)))(((.(.((((...((....))...)))).).)))--(((-((((.((......)).))))))).))))).)). ( -29.90, z-score = -0.03, R) >consensus GUGA_CCGGAGCCUGAGCUCACGGGUCGAUGCCUGUGCUCCACGGGCAG___GUGACCGGACUGGGUGUUCGGAUGCUG_________AAGAUCCGUGGCCAGGUACUGGCAG ...................(((((.....((((((((...))))))))...................((......))................)))))(((((...))))).. (-14.68 = -14.07 + -0.61)

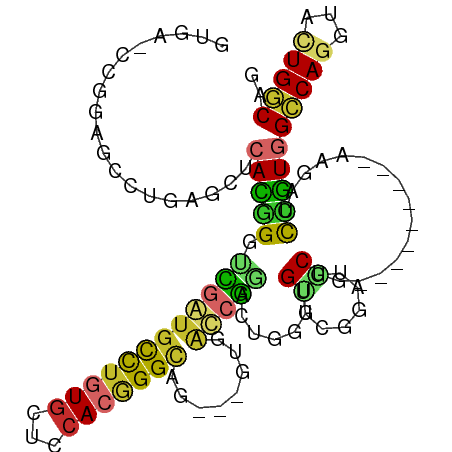
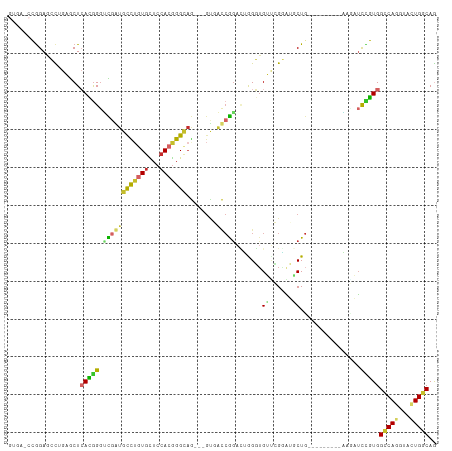
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:09:44 2011