| Sequence ID | dm3.chr2R |
|---|---|
| Location | 4,983,187 – 4,983,287 |
| Length | 100 |
| Max. P | 0.921455 |

| Location | 4,983,187 – 4,983,287 |
|---|---|
| Length | 100 |
| Sequences | 11 |
| Columns | 107 |
| Reading direction | forward |
| Mean pairwise identity | 85.63 |
| Shannon entropy | 0.30390 |
| G+C content | 0.42225 |
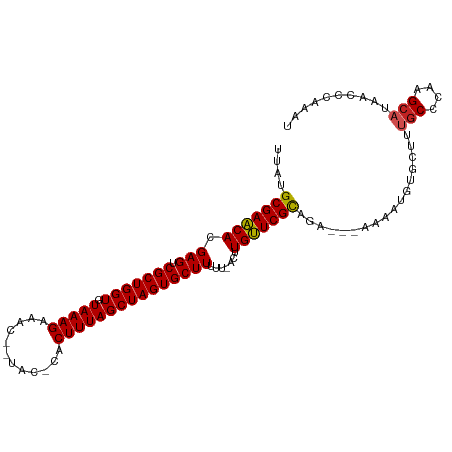
| Mean single sequence MFE | -26.75 |
| Consensus MFE | -20.90 |
| Energy contribution | -20.48 |
| Covariance contribution | -0.42 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.78 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.29 |
| SVM RNA-class probability | 0.921455 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
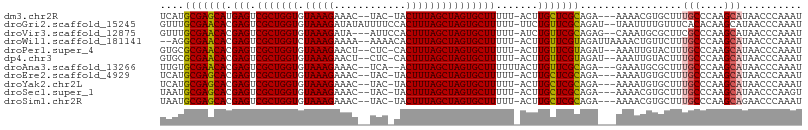
>dm3.chr2R 4983187 100 + 21146708 UCAUGCGAGCAUGAGUCGCUGGUGUAAAGAAAC--UAC-UACUUUAGCUAGUGCUUUUU-ACUUGCUCGCAGA---AAAACGUGCUUUGCCCAAGCAUAACCCAAAU ((.((((((((.(((.(((((((.(((((....--...-..)))))))))))))))...-...))))))))))---.....((((((.....))))))......... ( -30.40, z-score = -2.79, R) >droGri2.scaffold_15245 10600399 104 + 18325388 GUUUGCGAACACGAGUCGCUGGUGUAAAGAUAUAUUUUCCACUUUAGCUAGUGCUUUUU-UUCUGUUCGCAGAU--UAAUUUUGUUUCACACAAGCAUAACCCAAAU (((((((((((.(((.(((((((.(((((............)))))))))))))))...-...)))))))))))--......(((((.....))))).......... ( -24.20, z-score = -2.28, R) >droVir3.scaffold_12875 20243985 101 - 20611582 GUUUGCGAACACGAGUCGCUGGUGUAAAGAUA---AUUCCACUUUAGCUAGUGCUUUUU-AUCUGUUCGCAGAG--CAAAUGCGCUUCGCCCAAGCAUAACCCAAAU .((((((((((.(((.(((((((.(((((...---......)))))))))))))))...-...))))))))))(--(....))((((.....))))........... ( -27.50, z-score = -1.93, R) >droWil1.scaffold_181141 3772850 102 + 5303230 --AGGCGAACACGAGUCGCUGGUCUAAAGAAAA--AAAACACUUUAGCUAGUGCUUUUU-ACUUGUUCGUAGAUUAAAACUGUUCUUUGCCCAAGCAUAACCCAAAU --.((((((((.(((.((((((.((((((....--......)))))))))))))))(((-((......))))).......)))))...)))................ ( -22.80, z-score = -1.45, R) >droPer1.super_4 5020465 101 + 7162766 GUGCGCGAACACGAGUCGCUGGUGUAAAGAACU--CUC-CACUUUAGCUAGUGCUUUUU-ACUUGUUCGUAGAU--AAAUUGUACUUUGCCCAAGCAUAACCCAAAU ((((((((.......))))(((.((((((.((.--...-((((......))))...(((-((......))))).--.....)).))))))))).))))......... ( -23.10, z-score = -1.09, R) >dp4.chr3 9695762 101 + 19779522 GUGCGCGAACACGAGUCGCUGGUGUAAAGAACU--CUC-CACUUUAGCUAGUGCUUUUU-ACUUGUUCGUAGAU--AAAUUGUACUUUGCCCAAGCAUAACCCAAAU ((((((((.......))))(((.((((((.((.--...-((((......))))...(((-((......))))).--.....)).))))))))).))))......... ( -23.10, z-score = -1.09, R) >droAna3.scaffold_13266 16370308 100 - 19884421 UUGUGCGAACACGAGUCGCUGGUGUAAAGAAAC--UCA--ACUUUAGCUAGUGCUUUUUUACUUGUUCGCAGA---GAAAUGCGCUUUGCCCAAGCAUAACCCAAAU (((((((((((.(((.(((((((.(((((....--...--.))))))))))))))).......)))))(((((---(.......))))))....))))))....... ( -27.10, z-score = -1.54, R) >droEre2.scaffold_4929 8974828 100 - 26641161 UCAUGCGAGCACGAGUCGCUGGUGUAAAGAAAC--UAC-UACUUUAGCUAGUGCUUUUU-ACUUGCUCGCAGA---AAAAUGUGCUUUGCCCAAGCAUAACCCAAAU ((.((((((((.(((.(((((((.(((((....--...-..)))))))))))))))...-...))))))))))---....(((((((.....)))))))........ ( -30.10, z-score = -2.69, R) >droYak2.chr2L 17645258 100 + 22324452 UCAUGCGAGCACGAGUCGCUGGUGUAAAGAAAC--UAC-UACUUUAGCUAGUGCUUUUU-ACUUGCUCGCAGA---AAAAUGUGCUUUGCCCAAGCAUAACCCAAAU ((.((((((((.(((.(((((((.(((((....--...-..)))))))))))))))...-...))))))))))---....(((((((.....)))))))........ ( -30.10, z-score = -2.69, R) >droSec1.super_1 2621435 100 + 14215200 UAAUGCGAGCACGAGUCGCUGGUGUAAAGAAAC--UAC-UACUUUAGCUAGUGCUUUUU-ACUUGCUCGCAGA---AAAACGUGCUUUGCCCAAGCAUAACCCAAGU ...((((((((.(((.(((((((.(((((....--...-..)))))))))))))))...-...))))))))..---.....((((((.....))))))......... ( -28.60, z-score = -1.70, R) >droSim1.chr2R 3653819 100 + 19596830 UAAUGCGAGCACGAGUCGCUGGUGUAAAGAAAC--UAC-UACUUUAGCUAGUGCUUUUU-ACUUGCUCGCAGA---AAAACGUGCUUUGCCCAAGCAGAACCCAAAU ...((((((((.(((.(((((((.(((((....--...-..)))))))))))))))...-...))))))))..---......(((((.....))))).......... ( -27.20, z-score = -1.45, R) >consensus UUAUGCGAACACGAGUCGCUGGUGUAAAGAAAC__UAC_CACUUUAGCUAGUGCUUUUU_ACUUGUUCGCAGA___AAAAUGUGCUUUGCCCAAGCAUAACCCAAAU ....(((((((.(((.(((((((.(((((............))))))))))))))).......))))))).................(((....))).......... (-20.90 = -20.48 + -0.42)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:09:37 2011