
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 4,771,123 – 4,771,322 |
| Length | 199 |
| Max. P | 0.998517 |

| Location | 4,771,123 – 4,771,242 |
|---|---|
| Length | 119 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.30 |
| Shannon entropy | 0.27198 |
| G+C content | 0.51288 |
| Mean single sequence MFE | -46.88 |
| Consensus MFE | -32.85 |
| Energy contribution | -32.85 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.30 |
| Mean z-score | -3.83 |
| Structure conservation index | 0.70 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.39 |
| SVM RNA-class probability | 0.998517 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 4771123 119 - 21146708 AUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUGGAGAGACGUGCCUGAUGGAAGAGCAGCUUCUGCUUCUCCAUCUCUUGUUCCUG- ..(((((((((....)))))))))((.(((((((((....))).)))))).))....(((....)))((..(((.....((((((..(((((...)))))..))))))...)))..)).- ( -49.70, z-score = -4.35, R) >droSim1.chr2R 3444090 119 - 19596830 AUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUGGAGAGACGUGCCUGAUGGAAGAGCAGCUUCUGCUUCUCCAUCUCUUGUUCCUG- ..(((((((((....)))))))))((.(((((((((....))).)))))).))....(((....)))((..(((.....((((((..(((((...)))))..))))))...)))..)).- ( -49.70, z-score = -4.35, R) >droSec1.super_1 2408985 119 - 14215200 AUCUCCGAAGGUAUACCUUUGGACGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUGGAGAGACGUGCCUGAUGGAAGAGCAGCUUCUGCUUCUCCAUCUCUUGUUCCUG- ...((((((((....)))))))).((.(((((((((....))).)))))).))....(((....)))((..(((.....((((((..(((((...)))))..))))))...)))..)).- ( -46.60, z-score = -3.91, R) >droYak2.chr2L 17429625 119 - 22324452 AUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUGGAGAGACGUGCCUGAUGGAAGAGCAGUUUCUGCUUCUCCAUCUCUUGUUCCUG- ..(((((((((....)))))))))((.(((((((((....))).)))))).))....(((....)))((..(((.....((((((..(((((...)))))..))))))...)))..)).- ( -49.70, z-score = -4.50, R) >droEre2.scaffold_4929 8760841 119 + 26641161 AUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUGGAGAGACGUGCCUGGUGGAAGAGCAGUUUCUGCUUCUCCAUCUCUUGUUCCUG- ..(((((((((....)))))))))((.(((((((((....))).)))))).))....(((....)))((..(((.....((((((..(((((...)))))..))))))...)))..)).- ( -49.10, z-score = -3.90, R) >droAna3.scaffold_13266 18636806 119 + 19884421 AUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACACCACGAUUGGAGAGACGUGCCUGAUGGAAGAGCAGUUUCUGCUUUUCCAUCUCUUGUUCCUG- ..(((((((((....)))))))))((.(((((((((....))).)))))).))....(((....)))((..(((.....(((((((((((((...)))))))))))))...)))..)).- ( -52.60, z-score = -5.89, R) >droWil1.scaffold_180697 1450979 119 + 4168966 AUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCGCGGUUCGAAAGACGAGCCUGAUGGAAUAAUAGCUUUUGCUUCUCCAUCUCUUGUUCCUA- ..(((((((((....)))))))))((((((((((((....))).))))))(....)..)))((((((.....)))))).((((((.....(((....)))..))))))...........- ( -47.40, z-score = -4.67, R) >droGri2.scaffold_15112 3847351 119 - 5172618 AUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUCGAGAGUCGAGCCUGAUGGAACAGCAGCUUCUGUUUCUCCAUUUCUUGUUCCUG- ..(((((((((....)))))))))((.(((((((((....))).)))))).))........((((....))))((((..((((((..(((((...)))))..))))))....))))...- ( -43.00, z-score = -3.21, R) >droMoj3.scaffold_6496 13129734 119 + 26866924 AUCUCCGAAGGUAUACCUUUGGAGGCGGAUAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUCGACAGACGCGCUUGAUGGAACAGUAGCUUCUGUUUCUCCAUUUCUUGUUCCUG- .((((((((((....))))))))))(((((..((((....))))......(((...)))...))))).(((..(((...((((((((((......))))...))))))...)))..)))- ( -38.50, z-score = -2.59, R) >droVir3.scaffold_12875 1754926 119 - 20611582 AUCUCCGAAGGUAUACCUUUGGAGGCGGAUAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUCGAAAGACGCGCUUGAUGGAAGAGCAACUUCUGUUUCUCCAUUUCUUGUUCCUA- ..(((((((((....)))))))))(((((.((((((....))).))).))(((...))).....((....)))))((..((((((..((((.....))))..))))))....)).....- ( -40.20, z-score = -3.14, R) >droPer1.super_4 5762580 119 + 7162766 AUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUCGAGAGGCGGGCCUGAUGGAAGAGCAGCUUCUGCUUUUCCAUCUCUUGUUCCUA- ..(((((((((....)))))))))((.(((((((((....))).)))))).))...(((.(.......).)))((..(.(((((((((((((...)))))))))))))....)..))..- ( -55.30, z-score = -5.51, R) >dp4.chr3 13918737 119 - 19779522 AUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUCGAGAGGCGGGCCUGAUGGAAGAGCAGCUUCUGCUUUUCCAUCUCUUGUUCCUA- ..(((((((((....)))))))))((.(((((((((....))).)))))).))...(((.(.......).)))((..(.(((((((((((((...)))))))))))))....)..))..- ( -55.30, z-score = -5.51, R) >anoGam1.chr3L 8436342 119 - 41284009 AUCUCGGACGGGACGCCCUUCGAGGCGGAAAUGUGCAGCAGCACCAUCUCCGCGACGCCACGGUUGGCCAGACGUGCCUGGUGGAACAGGAGCUUCUGCUUUUCCAUUUCUUGUUCCUG- .....((((((((.(((......)))(((((...((((.(((.((...(((((...((((....))))(((......))))))))...)).))).)))).)))))...))))))))...- ( -45.20, z-score = -1.12, R) >apiMel3.Group2 3467167 120 - 14465785 AUCUCGCUGGGAACACCUUUGCAGGCUGAGAUGUGAAGCAGUACCAUCUCUGCCACGCCACGAUUCGCGAGUCGCGCUUGGUGGAAAAGAAGCUUCUGCUUUUCCAUUUCUUGUUCCUAG ......((((((((((((....)))..((((((.(((((((...(.((((..(((.((..(((((....))))).)).)))..))...)).)...)))))))..)))))).))))))))) ( -41.80, z-score = -2.15, R) >triCas2.ChLG3 11732883 119 + 32080666 AUUUCUGAGGGGACGCCUUUGCAUGCUGAUAUGUGCAGAAGGACCAUUUCUGCUACGCCUCGUUUGGCCAGUCUUGCUUGGUUGAAAAGCAACUUCUGCUUUUCCAUUUCUUGCUCCUG- .......(((((.((((...(((.((((....(((((((((.....))))))).))(((......)))))))..)))..))).((((((((.....))))))))........)))))).- ( -39.10, z-score = -2.62, R) >consensus AUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUGGAGAGACGUGCCUGAUGGAAGAGCAGCUUCUGCUUCUCCAUCUCUUGUUCCUG_ ..(((((((((....)))))))))((.((.((((((....))).))).)).))..........................((((((..(((((...)))))..))))))............ (-32.85 = -32.85 + 0.00)

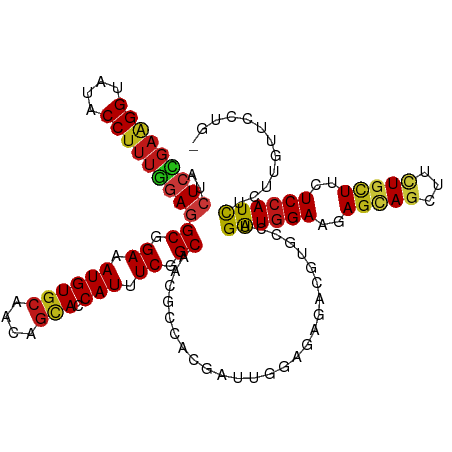
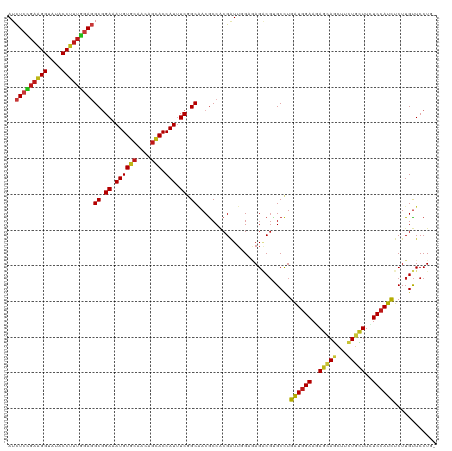
| Location | 4,771,162 – 4,771,282 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 86.20 |
| Shannon entropy | 0.32773 |
| G+C content | 0.53889 |
| Mean single sequence MFE | -43.45 |
| Consensus MFE | -21.48 |
| Energy contribution | -22.13 |
| Covariance contribution | 0.65 |
| Combinations/Pair | 1.30 |
| Mean z-score | -2.32 |
| Structure conservation index | 0.49 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.31 |
| SVM RNA-class probability | 0.638086 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 4771162 120 + 21146708 CAGGCACGUCUCUCCAAUCGUGGCGUUGCCGAAAUGGUGCUGUUGCACAUUUCCGCCUCCAAAGGUAUACCUUCGGAGAUGGUAAUGACGACACUCAAUCUCGGCAUCGCCAUUUUGCGC ..............(((..((((((.((((((.((.(.(.(((((..((((.(((.((((.((((....)))).)))).))).)))).)))))).).)).)))))).)))))).)))... ( -43.50, z-score = -2.85, R) >droSim1.chr2R 3444129 120 + 19596830 CAGGCACGUCUCUCCAAUCGUGGCGUUGCCGAAAUGGUGCUGUUGCACAUUUCCGCCUCCAAAGGUAUACCUUCGGAGAUGGUAAUGACGACACUCAAUCUCGGCAUAGCCAUUUUGCGC ..((((((((.(.......).)))).))))((((((.(((....)))))))))(((((((.((((....)))).))))(((((.(((.(((.........))).))).)))))...))). ( -42.20, z-score = -2.57, R) >droSec1.super_1 2409024 120 + 14215200 CAGGCACGUCUCUCCAAUCGUGGCGUUGCCGAAAUGGUGCUGUUGCACAUUUCCGCGUCCAAAGGUAUACCUUCGGAGAUGGUAAUGACGACACUCAAUCUCGGCAUAGCCAUUUUGCGC ..((((((((.(.......).)))).))))((((((.(((....)))))))))(((((((.((((....)))).)))((((((.(((.(((.........))).))).)))))).)))). ( -41.30, z-score = -2.06, R) >droYak2.chr2L 17429664 120 + 22324452 CAGGCACGUCUCUCCAAUCGUGGCGUUGCCGAAAUGGUGCUGUUGCACAUUUCCGCCUCCAAAGGUAUACCUUCGGAGAUGGUAAUGACGACACUCAAUCUCGGCAUCGCCAUUUUGCGC ..............(((..((((((.((((((.((.(.(.(((((..((((.(((.((((.((((....)))).)))).))).)))).)))))).).)).)))))).)))))).)))... ( -43.50, z-score = -2.85, R) >droEre2.scaffold_4929 8760880 120 - 26641161 CAGGCACGUCUCUCCAAUCGUGGCGUUGCCGAAAUGGUGCUGUUGCACAUUUCCGCCUCCAAAGGUAUACCUUCGGAGAUGGUAAUGACGACACUCAAUCUCGGCAUCGCCAUUCUGCGC ...(((((..........)))((((.((((((.((.(.(.(((((..((((.(((.((((.((((....)))).)))).))).)))).)))))).).)).)))))).)))).....)).. ( -43.50, z-score = -2.82, R) >droAna3.scaffold_13266 18636845 120 - 19884421 CAGGCACGUCUCUCCAAUCGUGGUGUUGCCGAAAUGGUGCUGUUGCACAUUUCCGCCUCCAAAGGUAUACCUUCGGAGAUGGUAAUGACGACACUCAAUCUCGGCAUCGCCAUCCUGAGA ((((.(((..........)))((((.((((((.((.(.(.(((((..((((.(((.((((.((((....)))).)))).))).)))).)))))).).)).)))))).))))..))))... ( -43.90, z-score = -2.90, R) >droWil1.scaffold_180697 1451018 120 - 4168966 CAGGCUCGUCUUUCGAACCGCGGCGUUGCCGAAAUGGUGCUGUUGCACAUUUCCGCCUCCAAAGGUAUACCUUCGGAGAUGGUAAUGACAACACUCAAUCUCGGCAUUGCCAUUCUACGU ..((.(((.....))).))..((((.((((((.((.(.(.(((((..((((.(((.((((.((((....)))).)))).))).)))).)))))).).)).)))))).))))......... ( -44.90, z-score = -3.82, R) >droGri2.scaffold_15112 3847390 120 + 5172618 CAGGCUCGACUCUCGAAUCGUGGCGUUGCCGAAAUGGUGCUGUUGCACAUUUCCGCCUCCAAAGGUAUACCUUCGGAGAUGGUCAUGACGACACUCAAUUUGGGCAUCGCCAUAUUGCGG ..(..(((.....)))..)((((((.((((.((((.(.(.(((((..(((..(((.((((.((((....)))).)))).)))..))).)))))).).)))).)))).))))))....... ( -46.30, z-score = -3.05, R) >droMoj3.scaffold_6496 13129773 120 - 26866924 CAAGCGCGUCUGUCGAAUCGUGGCGUUGCCGAAAUGGUGCUGUUGCACAUAUCCGCCUCCAAAGGUAUACCUUCGGAGAUGGUCAUGACGACACUCAAUUUGGGCAUCGCCAUACUGCGU ...((((.((....))...((((((.((((.((((.(.(.(((((..(((..(((.((((.((((....)))).)))).)))..))).)))))).).)))).)))).))))))...)))) ( -46.20, z-score = -2.65, R) >droVir3.scaffold_12875 1754965 120 + 20611582 CAAGCGCGUCUUUCGAAUCGUGGCGUUGCCGAAAUGGUGCUGUUGCACAUAUCCGCCUCCAAAGGUAUACCUUCGGAGAUGGUCAUGACGACACUCAAUUUGGGCAUCGCCAUAUUGCGU ...(((((((....))...((((((.((((.((((.(.(.(((((..(((..(((.((((.((((....)))).)))).)))..))).)))))).).)))).)))).))))))..))))) ( -46.20, z-score = -3.06, R) >droPer1.super_4 5762619 120 - 7162766 CAGGCCCGCCUCUCGAAUCGUGGCGUUGCCGAAAUGGUGCUGUUGCACAUUUCCGCCUCCAAAGGUAUACCUUCGGAGAUGGUAAUGACGACACUCAAUCUCGGCAUCGCCAUAUUGCGC .(((....)))..((....((((((.((((((.((.(.(.(((((..((((.(((.((((.((((....)))).)))).))).)))).)))))).).)).)))))).))))))....)). ( -46.10, z-score = -3.17, R) >dp4.chr3 13918776 120 + 19779522 CAGGCCCGCCUCUCGAAUCGUGGCGUUGCCGAAAUGGUGCUGUUGCACAUUUCCGCCUCCAAAGGUAUACCUUCGGAGAUGGUAAUGACGACACUCAAUCUCGGCAUCGCCAUAUUGCGC .(((....)))..((....((((((.((((((.((.(.(.(((((..((((.(((.((((.((((....)))).)))).))).)))).)))))).).)).)))))).))))))....)). ( -46.10, z-score = -3.17, R) >anoGam1.chr3L 8436381 120 + 41284009 CAGGCACGUCUGGCCAACCGUGGCGUCGCGGAGAUGGUGCUGCUGCACAUUUCCGCCUCGAAGGGCGUCCCGUCCGAGAUGGUGAUGCGUACGCUCGAGCUCGGAAUUGCCGUGCUGCGG ..(((.......)))..(((..(((..(((((((((.(((....))))))))))))..)....((((.....((((((....(((.((....)))))..))))))..))))..))..))) ( -53.70, z-score = -0.84, R) >apiMel3.Group2 3467207 120 + 14465785 CAAGCGCGACUCGCGAAUCGUGGCGUGGCAGAGAUGGUACUGCUUCACAUCUCAGCCUGCAAAGGUGUUCCCAGCGAGAUGGUGAUGAAGACCUUGGAAUUGGGAAUCUCCAUCCUACGC .....(((.(((((..(((...(((.(((.((((((...........)))))).))))))...))).......))))).(((.((((.((((((.......)))..))).)))))))))) ( -38.20, z-score = -0.05, R) >triCas2.ChLG3 11732922 120 - 32080666 CAAGCAAGACUGGCCAAACGAGGCGUAGCAGAAAUGGUCCUUCUGCACAUAUCAGCAUGCAAAGGCGUCCCCUCAGAAAUGGUAAUGAAAACACUCGAACUGGGCAUUUCUAUCCUCCGU ...(((.(.((((((......)))((.((((((.......))))))))....)))).)))..(((.((.((((((....)))...(((......)))....))).)).)))......... ( -26.10, z-score = 1.03, R) >consensus CAGGCACGUCUCUCCAAUCGUGGCGUUGCCGAAAUGGUGCUGUUGCACAUUUCCGCCUCCAAAGGUAUACCUUCGGAGAUGGUAAUGACGACACUCAAUCUCGGCAUCGCCAUUUUGCGC ...................((((((..((.((((((.(((....))))))))).))((((...((....))...)))).............................))))))....... (-21.48 = -22.13 + 0.65)

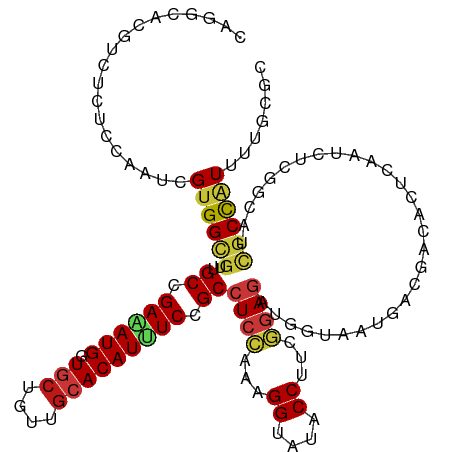
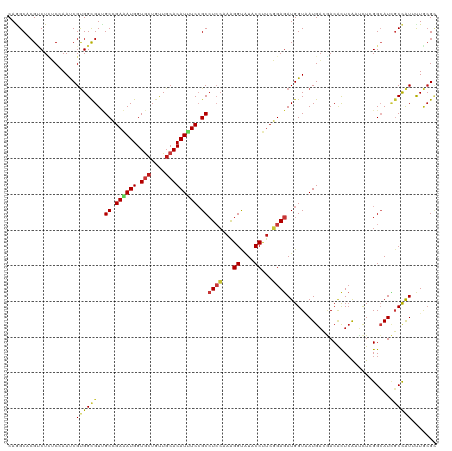
| Location | 4,771,162 – 4,771,282 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 86.20 |
| Shannon entropy | 0.32773 |
| G+C content | 0.53889 |
| Mean single sequence MFE | -47.97 |
| Consensus MFE | -30.87 |
| Energy contribution | -31.24 |
| Covariance contribution | 0.37 |
| Combinations/Pair | 1.33 |
| Mean z-score | -3.65 |
| Structure conservation index | 0.64 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.36 |
| SVM RNA-class probability | 0.998447 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 4771162 120 - 21146708 GCGCAAAAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUGGAGAGACGUGCCUG ((((....(((((.(((((((((.(.((((.((((((.((..(((((((((....)))))))))..)).))))....)).)))))))))))))).))))).....(......)))))... ( -52.80, z-score = -5.15, R) >droSim1.chr2R 3444129 120 - 19596830 GCGCAAAAUGGCUAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUGGAGAGACGUGCCUG ((((....((((..(((((((((.(.((((.((((((.((..(((((((((....)))))))))..)).))))....)).))))))))))))))..)))).....(......)))))... ( -48.80, z-score = -4.21, R) >droSec1.super_1 2409024 120 - 14215200 GCGCAAAAUGGCUAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGACGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUGGAGAGACGUGCCUG ((((....((((..(((((((((.(.((((.((((((.((...((((((((....))))))))...)).))))....)).))))))))))))))..)))).....(......)))))... ( -43.40, z-score = -2.85, R) >droYak2.chr2L 17429664 120 - 22324452 GCGCAAAAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUGGAGAGACGUGCCUG ((((....(((((.(((((((((.(.((((.((((((.((..(((((((((....)))))))))..)).))))....)).)))))))))))))).))))).....(......)))))... ( -52.80, z-score = -5.15, R) >droEre2.scaffold_4929 8760880 120 + 26641161 GCGCAGAAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUGGAGAGACGUGCCUG ((((....(((((.(((((((((.(.((((.((((((.((..(((((((((....)))))))))..)).))))....)).)))))))))))))).))))).....(......)))))... ( -52.80, z-score = -4.98, R) >droAna3.scaffold_13266 18636845 120 + 19884421 UCUCAGGAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACACCACGAUUGGAGAGACGUGCCUG ((((..(((((((((((((....)).))))))))))).(((.(((((((((....)))))))))((.(((((((((....))).)))))).))...........))).))))........ ( -48.20, z-score = -3.86, R) >droWil1.scaffold_180697 1451018 120 + 4168966 ACGUAGAAUGGCAAUGCCGAGAUUGAGUGUUGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCGCGGUUCGAAAGACGAGCCUG .........(((..(((((((((.(.(((((((((((.((..(((((((((....)))))))))..)).))))....)))))))))))))))))..)))..((((((.....)))))).. ( -56.20, z-score = -6.51, R) >droGri2.scaffold_15112 3847390 120 - 5172618 CCGCAAUAUGGCGAUGCCCAAAUUGAGUGUCGUCAUGACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUCGAGAGUCGAGCCUG .(((..(((((((((((.(.....).))))))))))).....(((((((((....))))))))))))(((((((((....))).))))))(((...))).(((((....)))))...... ( -48.00, z-score = -4.16, R) >droMoj3.scaffold_6496 13129773 120 + 26866924 ACGCAGUAUGGCGAUGCCCAAAUUGAGUGUCGUCAUGACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAUAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUCGACAGACGCGCUUG .(((....(((((.((((.((((.(.((((.((((((.((..(((((((((....)))))))))..)).))))....)).))))))))).)))).)))))....((....)).))).... ( -46.00, z-score = -3.34, R) >droVir3.scaffold_12875 1754965 120 - 20611582 ACGCAAUAUGGCGAUGCCCAAAUUGAGUGUCGUCAUGACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAUAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUCGAAAGACGCGCUUG .(((....(((((.((((.((((.(.((((.((((((.((..(((((((((....)))))))))..)).))))....)).))))))))).)))).)))))....((....)).))).... ( -49.00, z-score = -4.61, R) >droPer1.super_4 5762619 120 + 7162766 GCGCAAUAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUCGAGAGGCGGGCCUG .(((....(((((.(((((((((.(.((((.((((((.((..(((((((((....)))))))))..)).))))....)).)))))))))))))).))))).....(....))))...... ( -53.00, z-score = -4.52, R) >dp4.chr3 13918776 120 - 19779522 GCGCAAUAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUCGAGAGGCGGGCCUG .(((....(((((.(((((((((.(.((((.((((((.((..(((((((((....)))))))))..)).))))....)).)))))))))))))).))))).....(....))))...... ( -53.00, z-score = -4.52, R) >anoGam1.chr3L 8436381 120 - 41284009 CCGCAGCACGGCAAUUCCGAGCUCGAGCGUACGCAUCACCAUCUCGGACGGGACGCCCUUCGAGGCGGAAAUGUGCAGCAGCACCAUCUCCGCGACGCCACGGUUGGCCAGACGUGCCUG .........((((..((.(.((.(((.(((..((.((.....((((((.(((...)))))))))(((((.((((((....))).))).))))))).)).))).)))))).))..)))).. ( -44.70, z-score = -0.26, R) >apiMel3.Group2 3467207 120 - 14465785 GCGUAGGAUGGAGAUUCCCAAUUCCAAGGUCUUCAUCACCAUCUCGCUGGGAACACCUUUGCAGGCUGAGAUGUGAAGCAGUACCAUCUCUGCCACGCCACGAUUCGCGAGUCGCGCUUG ((((..((((((((((...........)))))))))).....(((((.((.....))...((.(((.((((((.(........))))))).)))..))........)))))..))))... ( -38.10, z-score = -0.81, R) >triCas2.ChLG3 11732922 120 + 32080666 ACGGAGGAUAGAAAUGCCCAGUUCGAGUGUUUUCAUUACCAUUUCUGAGGGGACGCCUUUGCAUGCUGAUAUGUGCAGAAGGACCAUUUCUGCUACGCCUCGUUUGGCCAGUCUUGCUUG ..(((((.((((((((....((..(((....)))...)))))))))).(....).)))))(((.((((....(((((((((.....))))))).))(((......)))))))..)))... ( -32.80, z-score = 0.15, R) >consensus GCGCAAAAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACAGCACCAUUUCGGCAACGCCACGAUUGGAGAGACGUGCCUG ..((..((((((((((((......).))))))))))).....(((((((((....)))))))))((.((.((((((....))).))).)).))......................))... (-30.87 = -31.24 + 0.37)

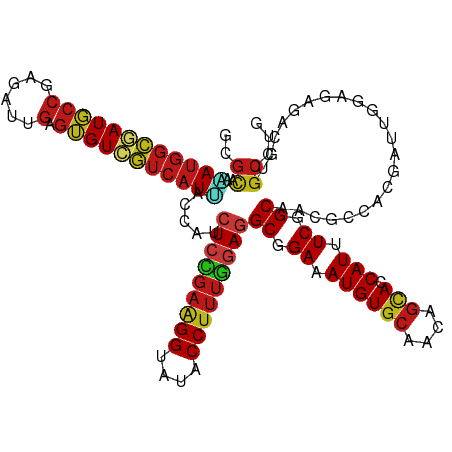
| Location | 4,771,202 – 4,771,322 |
|---|---|
| Length | 120 |
| Sequences | 15 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.44 |
| Shannon entropy | 0.33786 |
| G+C content | 0.49778 |
| Mean single sequence MFE | -41.76 |
| Consensus MFE | -27.86 |
| Energy contribution | -28.55 |
| Covariance contribution | 0.69 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.67 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.42 |
| SVM RNA-class probability | 0.938046 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 4771202 120 - 21146708 AGUGGUUCAGCAUGCCCAUUUGGAUGUCGAUGUUGCCGCCGCGCAAAAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACA .(((((((.((((.((.....)))))).))....))))).(((((.(((((((((((((....)).))))))))))).((..(((((((((....)))))))))..))...))))).... ( -48.80, z-score = -3.35, R) >droSim1.chr2R 3444169 120 - 19596830 AGUGGUUCAGCAUGCCCAUUUGGAUGUCGAUGUUUCCGCCGCGCAAAAUGGCUAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACA .((((.((.((((.((.....)))))).)).....)))).(((((.((((((.((((((....)).)))).)))))).((..(((((((((....)))))))))..))...))))).... ( -41.70, z-score = -1.89, R) >droSec1.super_1 2409064 120 - 14215200 AGUGGUUCAGCAUGCCCAUUUGGAUGUCGAUGUUUCCGCCGCGCAAAAUGGCUAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGACGCGGAAAUGUGCAACA ............(((.((((((((..........))).(((((....((((..(((.(((.((...)).))).)))..)))).((((((((....)))))))))))))))))).)))... ( -37.40, z-score = -0.79, R) >droYak2.chr2L 17429704 120 - 22324452 AGUGGUUCAGCAUGCCCAUUUGGAUGUCGAUGUUGCCACCGCGCAAAAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACA .(((((((.((((.((.....)))))).))....))))).(((((.(((((((((((((....)).))))))))))).((..(((((((((....)))))))))..))...))))).... ( -49.20, z-score = -3.69, R) >droEre2.scaffold_4929 8760920 120 + 26641161 AGUGGUUCAGCAUGCCCAUUUGGAUGUCGAUGUUGCCACCGCGCAGAAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACA .(((((((.((((.((.....)))))).))....))))).(((((.(((((((((((((....)).))))))))))).((..(((((((((....)))))))))..))...))))).... ( -49.30, z-score = -3.57, R) >droAna3.scaffold_13266 18636885 120 + 19884421 GAUGGUUCAGCAUGCCCAUUUGGAUGUCGAUGUUGCCGCCUCUCAGGAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACA .........(((....((((..(.(.(((..(((((((((.....)).)))))))..))).))..))))....((((.((..(((((((((....)))))))))..)).))))))).... ( -44.50, z-score = -2.04, R) >droWil1.scaffold_180697 1451058 120 + 4168966 GAUGAUUUAACAUACCCAUUUGGAUGUCAAUGUUACCGCCACGUAGAAUGGCAAUGCCGAGAUUGAGUGUUGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACA (.((((...((((.((.....))))))....)))).)((.((((..(((((((((((((....)).))))))))))).((..(((((((((....)))))))))..))..)))))).... ( -37.50, z-score = -2.47, R) >droGri2.scaffold_15112 3847430 120 - 5172618 GAUGAUUGAGCAUGCCCAUUUGGAUGUCGAUGUUGCCACCCCGCAAUAUGGCGAUGCCCAAAUUGAGUGUCGUCAUGACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACA ....(((((.(((.((.....))))))))))(((((((..((((..(((((((((((.(.....).))))))))))).....(((((((((....)))))))))))))...)).))))). ( -48.20, z-score = -4.23, R) >droMoj3.scaffold_6496 13129813 120 + 26866924 AGUGAUUCAACAUACCCAUUUGAAUGUCGAUGUUACCGCCACGCAGUAUGGCGAUGCCCAAAUUGAGUGUCGUCAUGACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAUAUGUGCAACA .(((....(((((...(((....)))...))))).....)))(((.(((((((((((.(.....).))))))))))).((..(((((((((....)))))))))..)).....))).... ( -40.20, z-score = -2.53, R) >droVir3.scaffold_12875 1755005 120 - 20611582 AAUGAUUCAGCAUGCCCAUCUGUAUAUCAAUGUUUCCGCCACGCAAUAUGGCGAUGCCCAAAUUGAGUGUCGUCAUGACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAUAUGUGCAACA ..((((.(((.((....)))))...))))((((.((((((......(((((((((((.(.....).)))))))))))......((((((((....)))))))))))))).))))...... ( -41.80, z-score = -3.04, R) >droPer1.super_4 5762659 120 + 7162766 GAUGAUUGAGCAUGCCCAUUUGGAUGUCGAUGUUGCCGCCGCGCAAUAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACA ....(((((.(((.((.....))))))))))((((((.((((.....((((((((((((....)).))))))))))......(((((((((....)))))))))))))....).))))). ( -44.80, z-score = -2.63, R) >dp4.chr3 13918816 120 - 19779522 GAUGAUUGAGCAUGCCCAUUUGGAUGUCGAUGUUGCCGCCGCGCAAUAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACA ....(((((.(((.((.....))))))))))((((((.((((.....((((((((((((....)).))))))))))......(((((((((....)))))))))))))....).))))). ( -44.80, z-score = -2.63, R) >anoGam1.chr3L 8436421 120 - 41284009 GAUGGUUCAACAUUCCCAUCUGGAUGUCGAUGUUACCACCCCGCAGCACGGCAAUUCCGAGCUCGAGCGUACGCAUCACCAUCUCGGACGGGACGCCCUUCGAGGCGGAAAUGUGCAGCA ..((((..((((((..(((....)))..))))))))))...((.(((.(((.....))).))))).((...(((((..((..((((((.(((...)))))))))..))..)))))..)). ( -40.00, z-score = -0.61, R) >apiMel3.Group2 3467247 120 - 14465785 AAUGGUUCAGCAUUCCUCGUUGAAUCUCUAUAUUACCGCCGCGUAGGAUGGAGAUUCCCAAUUCCAAGGUCUUCAUCACCAUCUCGCUGGGAACACCUUUGCAGGCUGAGAUGUGAAGCA ...((((((((.......))))))))..((((((.(.((((((.((((((((((((...........)))))))))).(((......)))......)).))).))).).))))))..... ( -32.60, z-score = -0.28, R) >triCas2.ChLG3 11732962 120 + 32080666 AAUGGUUUAACAUUCCCAUUUGGAUGUCGAAGUUACCACCACGGAGGAUAGAAAUGCCCAGUUCGAGUGUUUUCAUUACCAUUUCUGAGGGGACGCCUUUGCAUGCUGAUAUGUGCAGAA ...((((..((((((......))))))..).....((.((.((((((.((....)).))......((((....))))......)))).))))..)))(((((((........))))))). ( -25.60, z-score = 1.25, R) >consensus AAUGGUUCAGCAUGCCCAUUUGGAUGUCGAUGUUGCCGCCGCGCAAAAUGGCGAUGCCGAGAUUGAGUGUCGUCAUUACCAUCUCCGAAGGUAUACCUUUGGAGGCGGAAAUGUGCAACA ........(((((...(((....)))...)))))........(((.((((((((((((......).))))))))))).((.((((((((((....)))))))))).)).....))).... (-27.86 = -28.55 + 0.69)
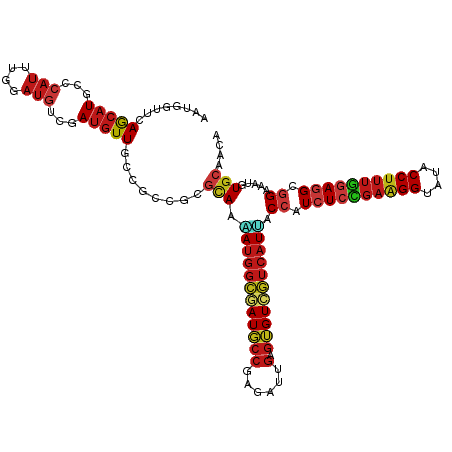
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:09:14 2011