| Sequence ID | dm3.chr2R |
|---|---|
| Location | 4,754,477 – 4,754,593 |
| Length | 116 |
| Max. P | 0.610992 |

| Location | 4,754,477 – 4,754,593 |
|---|---|
| Length | 116 |
| Sequences | 15 |
| Columns | 122 |
| Reading direction | reverse |
| Mean pairwise identity | 70.83 |
| Shannon entropy | 0.66138 |
| G+C content | 0.45070 |
| Mean single sequence MFE | -31.38 |
| Consensus MFE | -10.81 |
| Energy contribution | -10.13 |
| Covariance contribution | -0.69 |
| Combinations/Pair | 1.56 |
| Mean z-score | -1.48 |
| Structure conservation index | 0.34 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.610992 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 4754477 116 - 21146708 UACUCACCAUAUCACUGUUGUGGUGACGCAGCAAAAUGGCAUUGCCAUAGAGCAAUGUCCUGUGUCCCGAUCCGGUGCCUUUGCCCUAGGA-----GUUUGUGGGAAAGCCAUUAAAAAAU- ...((((((((.......))))))))(((((......((((((((......)))))))))))))(((((.(((((.((....))))..)))-----.....)))))...............- ( -35.80, z-score = -1.73, R) >apiMel3.Group2 3449065 115 - 14465785 UAUUUACCAUGUCGCUGUUCUGAUGCCUUAGUAAAAUUGCGUUACCGUACAACAACGUUCUGUGUCCGGAUCCUGUACCCUUUCCCUACAAA---UGCACAGAGAUAUAGCGAUUGAA---- .......((.((((((((((((.(((....)))....((((((.(((.(((.((......))))).)))....((((.........))))))---))))))))...))))))))))..---- ( -24.50, z-score = -1.46, R) >droSim1.chr2R 3431633 116 - 19596830 UACUCACCAUAUCACUGUUGUGAUGUCGCAGCAAAAUGGCAUUGCCAUAGAGCAAUGUCCUGUGUCCCGAUCCUGUGCCUUUGCCCUAGGA-----AUUUGUGGAAAAGCCAUUAGAAAGU- ......(((((.((((((((((....)))))))....((((((((......))))))))..)))......(((((.((....))..)))))-----...))))).................- ( -29.80, z-score = -1.09, R) >droSec1.super_1 2392288 116 - 14215200 UACUCACCAUAUCACUGUUGUGGUGGCGCAGCAAAAUGGCAUUGCCAUAGAGCAAUGUCCUGUGUCCCGAUCCGGUGCCUUUGCCCUAGGA-----AUUUGUGGGAAAGCCAUUAGAAAGU- ....(((((((.......)))))))((((((......((((((((......)))))))))))))).....((..(((.((((.(((.....-----......))))))).)))..))....- ( -35.60, z-score = -0.84, R) >droYak2.chr2L 17413044 116 - 22324452 UACUCACCAUAUCACUGUUGUGAUGGCGCAGCAAAAUGGCAUUGCCGUAGAGCAAUGUCCUGUGUCCCGAUCCGGUGCCUUUGCCCUAUGA-----AUUUUUGAGAACGCCGUUAAAAAUU- ....((((..(((((....)))))(((((((......((((((((......))))))))))))))).......)))).............(-----((((((((........)))))))))- ( -30.20, z-score = -1.24, R) >droEre2.scaffold_4929 8744173 116 + 26641161 UACUCACCAUAUCACUGUUGUGAUGGCGCAACAAAAUGGCAUUGCCGUAGAGCAAUGUCCUGUGUCCCGAUCCGGUGCCUUUGCCCUAGGA-----AUUUUGGGGAAAGCCAUUAAAAAUG- ....((((.((((((....)))))).((..(((....((((((((......))))))))...)))..))....)))).((((.((((((..-----...))))))))))............- ( -32.70, z-score = -0.83, R) >droAna3.scaffold_13266 18619425 114 + 19884421 -ACUCACCAUGUCACUAUUGUGAUGACGGAGCAGAAUGGCAUUGCCGUAGAGCAAUGUCCUGUGUCCGGAUCCAGUUCCCUUGCCCUAGGA-----AUUGUUGG-UAAGCGAUUAAUAAGU- -.((.((((.(((((....)))))..((((((((...((((((((......)))))))))))).))))....(((((((.........)))-----)))).)))-).))............- ( -34.70, z-score = -1.85, R) >dp4.chr3 13902704 118 - 19779522 CACUUACCAUAUCACUGUUGUGAUGUCGGAGCAAAAUGGCAUUGCCGUAGAGCAAUGUCCUAUGUCCCGAUCCGGUACCUUUGCCCUAUCG----AAUAUCGGAAAUGAUUGAUUAAUGACU .........((((((....))))))((((..((....((((((((......))))))))...))..))))(((((((..((((......))----))))))))).................. ( -31.70, z-score = -1.86, R) >droPer1.super_4 5746410 118 + 7162766 CACUUACCAUAUCACUGUUGUGAUGUCGGAGCAAAAUGGCAUUGCCGUAGAGCAAUGUCCUAUGUCCCGAUCCGGUACCUUUGCCCUAUCG----AAUAUCGGAAAUGAUUGAUUAAUGACU .........((((((....))))))((((..((....((((((((......))))))))...))..))))(((((((..((((......))----))))))))).................. ( -31.70, z-score = -1.86, R) >droWil1.scaffold_180697 1434439 121 + 4168966 GACUUACCAUAUCACUAUUGUGAUGUCUUAAUAAAAUGGCAUUGCCAUAUAGCAAUGUCCUGUGACCGGAGCCAGUGCCUUUACCCUAAAGUUGUUGUUUUGGGUUAAAGCGUUAAAUAAU- (((...(((((((((....)))))))...........((((((((......))))))))........)).......((.((((.(((((((......))))))).))))))))).......- ( -28.60, z-score = -1.30, R) >droVir3.scaffold_12875 1739138 112 - 20611582 -GCUUACCAUAUCGCUGUUGUGAUGCCUGAGAAGUAUGGCAUUGCCAUAGAGUAGUGUUCUAUGGCCAGAUCCAGUGCCUUUACCCUGAAG-----AUUU--G--AAAAUUAAUAAGUAAGC -((((((..((((((....)))))).((.....(((.(((((((((((((((.....))))))))).......))))))..))).....))-----....--.--...........)))))) ( -31.11, z-score = -2.78, R) >droMoj3.scaffold_6496 13113790 112 + 26866924 -GCUUACCAUGUCGCUAUUGUGAUGCCGCAGGAGUAUGGCAUUGCCAUAGAGCAGGGUUCUAUGGCCGGAUCCUGUGCCCUUGCCCUGUAG-----AUUU--G--AAUAGUAAUUAGUAAGU -((((((...((..(((((..(((.(..((((.(((.(((((.((((((((((...)))))))))).(....).)))))..)))))))..)-----))).--.--)))))..))..)))))) ( -41.60, z-score = -2.96, R) >droGri2.scaffold_15112 3829117 114 - 5172618 -GCUCACCAUAUCGCUGUUGUGAUGCCUCAGGAGUAUCGCGUUGCCAUAGAGUAGUGUUCUAUGGCCAGAUCCGGUGCCUUUACCCUAAAG-----AUUUUCA--AAAAAUAAUUACUAACU -...((((..(((......((((((((....).)))))))...(((((((((.....)))))))))..)))..)))).(((((...)))))-----.......--................. ( -27.40, z-score = -1.41, R) >anoGam1.chr3L 8416901 116 - 41284009 UACUUACCAUGUCACUGUUGUUGUGACGCAACAGGAUAGCAUUGCCGUAAAGCAGCGUACGAUGACCGGAUCCGGUACCCUUUCCCUGUCG-----UUUUGUUUAUGCGAUACCGAAAAGU- .........((((.(((((((......))))))))))).(((((.(((......)))..)))))((((....))))....((((..(((((-----(.........))))))..))))...- ( -28.80, z-score = -0.42, R) >triCas2.ChLG3 11659788 102 + 32080666 -AUUUACCAUAUCACUGUUUUGAUGGCGUAAAAGGAUGGCAUUACCAUACAAAAGGGUGCGGUGUCCAGAUCCUGUACCUUUACCCUAGAAG---GGAGUAAGCAA---------------- -.(((((..(((((......)))))..)))))......((.((((.......((((((((((..(....)..)))))))))).(((.....)---)).))))))..---------------- ( -26.50, z-score = -0.49, R) >consensus UACUUACCAUAUCACUGUUGUGAUGCCGCAGCAAAAUGGCAUUGCCAUAGAGCAAUGUCCUGUGUCCCGAUCCGGUGCCUUUGCCCUAGAA_____AUUUUUGGAAAAACCAUUAAAAAAU_ ....(((..((((((....)))))).............(((((((......)))))))................)))............................................. (-10.81 = -10.13 + -0.69)

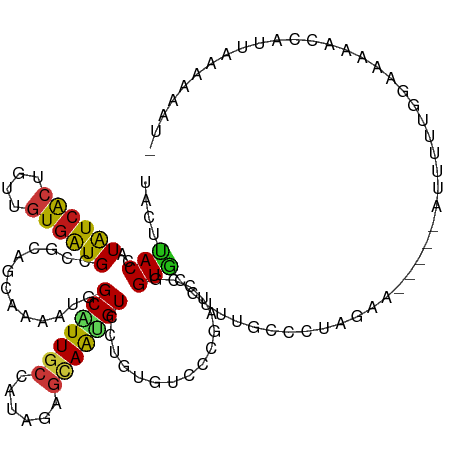

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:09:07 2011