
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 3,343,405 – 3,343,525 |
| Length | 120 |
| Max. P | 0.608341 |

| Location | 3,343,405 – 3,343,525 |
|---|---|
| Length | 120 |
| Sequences | 14 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.89 |
| Shannon entropy | 0.47792 |
| G+C content | 0.51131 |
| Mean single sequence MFE | -40.12 |
| Consensus MFE | -21.88 |
| Energy contribution | -20.72 |
| Covariance contribution | -1.16 |
| Combinations/Pair | 1.68 |
| Mean z-score | -1.28 |
| Structure conservation index | 0.55 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.608341 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
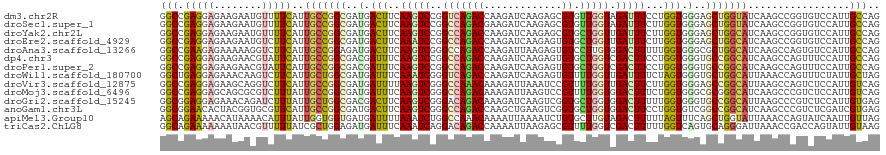
>dm3.chr2R 3343405 120 - 21146708 GGCCGAGGAGAAGAAUGUUUUCAUUGCCGGCGAUGACUUCAAGUCCGGUCAGACCAAGAUCAAGAGCGUGUUGGUAGAUUUCCUGGUGGGAGCUGGUAUCAAGCCGGUGUCCAUUGCCAG (((..((((((.(((....))).((((((((.(((.(((...(((.((.....))..)))...)))))))))))))).))))))(((((..((((((.....))))))..)))))))).. ( -40.10, z-score = -1.07, R) >droSec1.super_1 990348 120 - 14215200 GGCCGAGGAGAAGAAUGUUUUCAUUGCCGGCGAUGACUUCAAGUCCGGCCAGACGAAGAUCAAGAGCGUGUUGGUAGAUUUCUUGGUGGGAGCUGGUAUCAAGCCGGUGUCCAUUGCCAG (((...((((((((...)))))...((((((..(((..(((..(((.((((((.....(((((.......))))).....)).)))).)))..)))..))).)))))).)))...))).. ( -39.90, z-score = -0.96, R) >droYak2.chr2L 16022045 120 - 22324452 GGCCGAGGAGAAGAAUGUUUUCAUUGCCGGCGAUGACUUCAAGUCCGGCCAGACCAAGAUCAAGAGCGUGCUGGUUGAUUUCUUGGUGGGAGCUGGCAUCAAGCCGGUGUCCAUUGCCAG (((....(((((.....)))))...)))(((((((((......(((.(((((....(((((((.((....))..))))))).))))).)))((((((.....)))))))).))))))).. ( -42.30, z-score = -0.75, R) >droEre2.scaffold_4929 7386547 120 + 26641161 GGCCGAGGAGAAGAAUGUCUUCAUUGCCGGCGAUGACUUCAAAUCCGGCCAGACCAAGAUCAAGAGUGUGCUGGUUGAUUUCUUGGUGGGAGCUGGCAUCAAGCCGGUGUCCAUUGCCAG (((...(((((......)))))...)))(((((((((......(((.(((((....(((((((.((....))..))))))).))))).)))((((((.....)))))))).))))))).. ( -44.20, z-score = -1.38, R) >droAna3.scaffold_13266 6811224 120 - 19884421 GGCCGAAGAGAAAAAGGUCUUCAUUGCCGGAGAUGACUUCAAGUCGGGCCAGACCAAGAUUAAGAGUGUCCUUGUGGACUUUUUGGUGGGCGCUGGCAUCAAGCCAGUGUCCAUUGCCAG ((((((((((.....(((((.....(((.((..((....))..)).))).))))).............(((....))))))))))((((((((((((.....)))))))))))).))).. ( -48.80, z-score = -3.28, R) >dp4.chr3 13086777 120 + 19779522 GGCCGAGGAGAAGAACGUAUUCAUUGCCGGCGACGAUUUCAAGUCCGGCCAGACCAAGAUCAAGAGUGUGCUGGUCGACUUCCUGGUGGGUGCCGGCAUCAAGCCAGUUUCCAUUGCCAG (((...(((((.(((....)))..((((((((.(.(((..(((((.((((((..((..........))..)))))))))))...))).).)))))))).........)))))...))).. ( -41.70, z-score = -0.99, R) >droPer1.super_2 2121513 120 + 9036312 GGCCGAGGAGAAGAACGUAUUCAUUGCCGGCGACGAUUUCAAGUCCGGCCAGACCAAGAUCAAGAGUGUGCUGGUCGACUUCCUGGUGGGUGCCGGCAUCAAGCCAGUUUCCAUUGCCAG (((...(((((.(((....)))..((((((((.(.(((..(((((.((((((..((..........))..)))))))))))...))).).)))))))).........)))))...))).. ( -41.70, z-score = -0.99, R) >droWil1.scaffold_180700 475848 120 + 6630534 GGCUGAGGAGAAACAAGUCUUCAUUGCUGGCGAUGAUUUCAAAUCGGGUCAGACCAAGAUCAAGAGUGUUUUGGUUGAUUUUCUAGUGGGUGCUGGCAUUAAACCAGUUUCUAUUGCUAG ...((((((........))))))...((((((((........)))(((((((.(((((((.......))))))))))))))...((((((.(((((.......))))).))))))))))) ( -31.50, z-score = -0.18, R) >droVir3.scaffold_12875 10867868 120 - 20611582 GGCCGAGGAGAAGCAGGUCUUCAUUGCCGGCGAUGAUUUUAAGUCGGGCCAAACAAAGAUUAAAUCCGUUUUGGUUGACUUCUUGGUGGGAGCCGGCAUUAAGCCAGUCUCCAUUGUCAG (((...(((((....((.(((...(((((((..(.(((..(((((((.(((((....(((...)))...))))))))))))...))).)..)))))))..)))))..)))))...))).. ( -40.90, z-score = -1.82, R) >droMoj3.scaffold_6496 10797960 120 + 26866924 GGCCGAGGAGCAGCGCGUCUUUAUUGCCGGCGAUGAUUUCAAGUCGGGCCAGACAAAGAUUAAGUCCGUUUUGGUGGACUUUCUGGUGGGCGCGGGCAUCAAGCCCGUCUCCAUUGUCAG (((...(((((...((((((......(((((..((....)).)))))((((((........(((((((......)))))))))))))))))))((((.....))))).))))...))).. ( -48.10, z-score = -2.23, R) >droGri2.scaffold_15245 3972185 120 - 18325388 GGCGGAGGAGAAACAGAUCUUUAUUGCUGGCGACGACUUCAAGUCGGGACAGACAAAGAUCAAGUCGGUGCUGGUGGACUUUUUGGUGGGUGCCGGCAUUAAGCCCGUCUCCAUUGUGAG .((((.(((((....(..(((...((((((((.(.(((..(((((.(..(((.((..(((...)))..))))).).)))))...))).).))))))))..)))..).))))).))))... ( -41.00, z-score = -1.05, R) >anoGam1.chr3L 33203620 120 - 41284009 GGCGGAACACUACGGUGCGUUCAUUGCCGGCGAUGACUUCAAGUCGGGCCAGACCAAGCUGAAGUCGGUGCUGGUGGACUUCCUGGUGUCGGCCGGCAUCAAGCCCGUCUCGAUCGUGAG (((.((((((....))..))))...)))(((...(((((((.((..((.....))..)))))))))(((((((((.(((........))).)))))))))..)))...((((....)))) ( -50.20, z-score = -1.26, R) >apiMel3.Group10 9569085 120 + 11440700 AGCAGAAAAACAUAAAACAUUUAUUGGUGGUGAUGAUUUUAAAUCUGGCCAAACAAAAUUAAAAUCUGUGCUUGUAGACUUUUUAGUUUCAGCUGGUAUUAAACCAGUAUCAAUUGUUAG ((((((((....(((((..(((((....(((...((((((((.(.((......)).).))))))))...))).)))))..))))).)))).((((((.....))))))......)))).. ( -22.30, z-score = -0.42, R) >triCas2.ChLG8 12776631 120 + 15773733 GGCAGAAAAAAAUAACGUUUUUAUCGCUGGAGAUGAUUUCAAAUCAGGACAGACCAAAAUUAAGAGCGUUUUGGUCGACUUUUUGGUCAGUGCAGGGAUUAAACCGACCAGUAUUGUAAG .(((((.((((((...)))))).))(((((.(((........))).(.((.(((((((((.......)))))))))((((....)))).)).).((.......))..)))))..)))... ( -29.00, z-score = -1.57, R) >consensus GGCCGAGGAGAAGAAUGUCUUCAUUGCCGGCGAUGACUUCAAGUCCGGCCAGACCAAGAUCAAGAGCGUGCUGGUGGACUUCUUGGUGGGAGCUGGCAUCAAGCCAGUCUCCAUUGCCAG (((.(((((........)))))..(((((((..(.(((..(((((..(((((((.............)).))))).)))))...))).)..))))))).................))).. (-21.88 = -20.72 + -1.16)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:05:33 2011