
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 3,184,601 – 3,184,707 |
| Length | 106 |
| Max. P | 0.624861 |

| Location | 3,184,601 – 3,184,707 |
|---|---|
| Length | 106 |
| Sequences | 12 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 69.40 |
| Shannon entropy | 0.65707 |
| G+C content | 0.56515 |
| Mean single sequence MFE | -38.22 |
| Consensus MFE | -14.85 |
| Energy contribution | -13.77 |
| Covariance contribution | -1.07 |
| Combinations/Pair | 1.80 |
| Mean z-score | -1.22 |
| Structure conservation index | 0.39 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.28 |
| SVM RNA-class probability | 0.624861 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
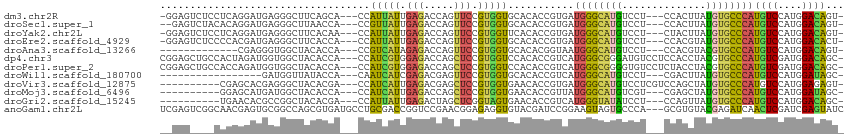
>dm3.chr2R 3184601 106 + 21146708 -GGAGUCUCCUCAGGAUGAGGGCUUCAGCA---CCAUUAUUGAGACCAGUUCCGUGGUGCACACCGUGAUGGGCAUGUCCU---CCACUUAUGUGCCCAUGUCCAUGGACAGU- -(((((..((((.....))))))))).(((---((((.((((....))))...)))))))...(((((((((((((((...---......))))))))))...))))).....- ( -40.30, z-score = -1.45, R) >droSec1.super_1 828515 105 + 14215200 --GAGUCUACACAGGAUGAGGGCUUAACCA---CCGUUAUUGAGACCAGUUCCGUGGUGCACACCGUGAUGGGCAUGUCCU---CCACUUAUGUGCCCAUGUCCAUGGACAGU- --..(((((....(((((.((((...((((---(.(..((((....))))..))))))..(((..(((..(((....))).---.)))...))))))).))))).)))))...- ( -33.30, z-score = -0.22, R) >droYak2.chr2L 15868944 106 + 22324452 -GGAGUCUCCUCAGGAUGAGGGCUUCACAA---CCAUUAUUGAGACCAGUUCCGUGGUUCACACCGUGAUGGGCAUGUCCU---CUACUUAUGUGCCCAUGUCCAUGGACAGU- -(((((..((((.....)))))))))..((---((((.((((....))))...))))))....(((((((((((((((...---......))))))))))...))))).....- ( -33.70, z-score = -0.25, R) >droEre2.scaffold_4929 7231867 106 - 26641161 -GGAGUCUCCCCACGAUGAGGGCUUCACCA---CCAUUAUUGAGACCAGUUCCGUGGUGCACACCGUGAUGGGCAUGUCCU---CCACGUAUGUGCCCAUGUCCAUGGACACU- -((((((((.....((((..((.....)).---.))))...)))))....)))((((.(((((.((((..(((....))).---.))))..)))))))))(((....)))...- ( -36.70, z-score = -0.64, R) >droAna3.scaffold_13266 3397378 94 + 19884421 -------------CGAGGGUGGCUACACCA---CCGUCAUAGAGACCAGUUCCGUGGUGCACACGGUAAUGGGCAUGUCCU---CCACGUACGUGCCCAUGUCCAUGGACAGU- -------------.((.(((((.....)))---)).)).............(((((.....)))))..((((((((((.(.---....).))))))))))(((....)))...- ( -38.40, z-score = -1.66, R) >dp4.chr3 1453882 110 + 19779522 CGGAGCUGCCACUAGAUGGUGGCUACACCA---CCAUCGUGGAGACCAGCUCCGUGGUCCACACCGUCAUGGGCGGGAUGUCCUCCACCUACGUGCCCAUGUCGAUGGACAGC- ((((((((((((..((((((((.....)))---)))))))))....))))))))..(((((...((.((((((((((.((.....)))))....))))))).)).)))))...- ( -55.50, z-score = -3.77, R) >droPer1.super_2 1627134 110 + 9036312 CGGAGCUGCCACCAGAUGGUGGCUACACCA---CCAUCGUGGAGACCAGCUCCGUGGUCCACACCGUCAUGGGCGGGGUGUCCUCUACCUACGUGCCCAUGUCGAUGGACAGC- ((((((((((((..((((((((.....)))---)))))))))....))))))))..(((((...((.(((((((((((((.....)))))...)))))))).)).)))))...- ( -58.80, z-score = -4.08, R) >droWil1.scaffold_180700 2828072 90 + 6630534 -----------------GAUGGUUAUACCA---CAAUCAUCGAGACGAGUUCCGUGGUGCACACCGUCAUGGGCAUGUCCU---CGACUUAUGUGCCCAUGUCCAUGGAUAGC- -----------------((((((..(((((---(.....(((...))).....))))))...))))))(((((((((((..---.)))....))))))))(((....)))...- ( -30.00, z-score = -1.16, R) >droVir3.scaffold_12875 19045015 100 - 20611582 ----------CGAGCACGAGGGCUACACGA---CCAUCAUUGAGACGAGCUCCGUGGUGAACACCGUGAUGGGCAUGUCCUCGUCCAGCUAUGUGCCCAUGUCCAUGGAGAGU- ----------..(((((((((((.......---(((((((....(((.....)))(((....))))))))))....))))))))...)))......((((....)))).....- ( -31.60, z-score = 0.42, R) >droMoj3.scaffold_6496 22192013 97 - 26866924 ----------GGAGCAUGAUGGCUACACCA---CCAUCAUUGAGACCAGCUCCGUGGUGAACACCGUUAUGGGCAUGUCGU---CGAGCUAUGUGCCCAUGUCCAUGGAUAGC- ----------((((((((((((........---)))))))((....)))))))..(((....)))....(((((((((.(.---....).)))))))))((((....))))..- ( -34.30, z-score = -1.09, R) >droGri2.scaffold_15245 9435328 97 + 18325388 ----------UGAACACGCCGGCUACACGA---CCAUUAUUGAGACUAGCUCGGUAGUGAACACCGUCAUGGGUAUAUCCU---CCAGUUAUGUGCCCAUGUCCAUGGACAGC- ----------....((((((((((((.(((---......))).)..)))).)))).)))....(((((((((((((((...---......)))))))))))...)))).....- ( -28.20, z-score = -0.82, R) >anoGam1.chr2L 47761502 111 + 48795086 UCGAGUCGGCAACGAGUGCGGCCAGCGUGAUGCCUGCGACCGGUCCGAACGGAGAGGUGUACGAUCCGGAAGUAGUGCCCA---GCGUGUACGAGAUCAACUCGAUCGAGUAUC ((((.(((....))).).)))...((((...((((((..((((..((.(((......))).))..))))..)))).))...---))))((((..((((.....))))..)))). ( -37.80, z-score = 0.05, R) >consensus __GAG____CACAGGAUGAGGGCUACACCA___CCAUCAUUGAGACCAGCUCCGUGGUGCACACCGUGAUGGGCAUGUCCU___CCACUUAUGUGCCCAUGUCCAUGGACAGU_ ...................................(((((.(((.....))).)))))...........((((((((..............))))))))((((....))))... (-14.85 = -13.77 + -1.07)


Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:05:19 2011