
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 2,835,230 – 2,835,329 |
| Length | 99 |
| Max. P | 0.997117 |

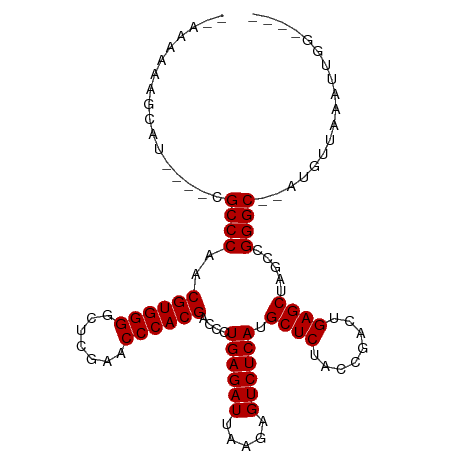
| Location | 2,835,230 – 2,835,329 |
|---|---|
| Length | 99 |
| Sequences | 13 |
| Columns | 107 |
| Reading direction | forward |
| Mean pairwise identity | 83.62 |
| Shannon entropy | 0.37405 |
| G+C content | 0.53247 |
| Mean single sequence MFE | -32.75 |
| Consensus MFE | -30.32 |
| Energy contribution | -30.32 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.66 |
| Structure conservation index | 0.93 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 3.04 |
| SVM RNA-class probability | 0.997117 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
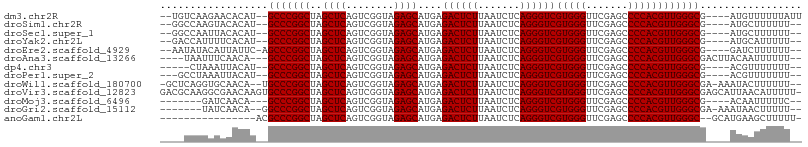
>dm3.chr2R 2835230 99 + 21146708 AAUAAAAAACAU----CGCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGC--AUGUGUUCUUGACA-- ........((((----.((((..((((((.......))))))....((((((.....)))))).((((........)))).....))))--))))..........-- ( -33.70, z-score = -3.25, R) >droSim1.chr2R 1651958 97 + 19596830 --AAAAAAGCAU----CGCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGC--AUGUGUACUUGGCC-- --......((((----.((((..((((((.......))))))....((((((.....)))))).((((........)))).....))))--))))..........-- ( -34.00, z-score = -2.44, R) >droSec1.super_1 517287 97 + 14215200 --AAAAAAGCAU----CGCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGC--AUGUGUAAUUGGCC-- --......((((----.((((..((((((.......))))))....((((((.....)))))).((((........)))).....))))--))))..........-- ( -34.00, z-score = -2.60, R) >droYak2.chr2L 15567447 97 + 22324452 --AAAAAUGCAU----CGCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGC--AUGUGAAAAUGGUC-- --......((((----.((((..((((((.......))))))....((((((.....)))))).((((........)))).....))))--))))..........-- ( -33.90, z-score = -2.81, R) >droEre2.scaffold_4929 20278928 98 + 26641161 --AAAAAAGAUC----CGCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGCU-GAAUAAUGUAUAUU-- --........((----.((((..((((((.......))))))....((((((.....)))))).((((........)))).....)))).-))............-- ( -32.10, z-score = -3.33, R) >droAna3.scaffold_13266 9158470 98 + 19884421 --AAAAAAUUGUAAGUCGCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGC---UGUUGAAAUUA---- --....((((...((..((((..((((((.......))))))....((((((.....)))))).((((........)))).....))))---..))..)))).---- ( -31.30, z-score = -2.50, R) >dp4.chr3 1124948 94 + 19779522 --AAAAAAACGU----CGCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGC--AUGUAAUUUAG----- --......((((----.((((..((((((.......))))))....((((((.....)))))).((((........)))).....))))--)))).......----- ( -33.30, z-score = -3.66, R) >droPer1.super_2 1289196 96 + 9036312 --AAAAAAACGU----CGCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGC--AUGUAAUUUAGGC--- --......((((----.((((..((((((.......))))))....((((((.....)))))).((((........)))).....))))--)))).........--- ( -33.30, z-score = -2.79, R) >droWil1.scaffold_180700 5892402 101 + 6630534 --AAAAAAGUAUUU-UCGCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGCA--UGUUGCACCUGAGC- --......(((...-..((((..((((((.......))))))....((((((.....)))))).((((........)))).....)))).--...)))........- ( -32.00, z-score = -1.92, R) >droVir3.scaffold_12823 845589 106 - 2474545 -AAAAAUGUUAAUGCUCGCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGCACUUGUUCGCCUUGCGUC -............(((((..(..((((((.......))))))....((((((.....)))))).........)..)))))..((.((((........)))).))... ( -32.30, z-score = -1.30, R) >droMoj3.scaffold_6496 25272262 91 + 26866924 --GAAAAAUUGU----CGCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGC---UGUUGAUC------- --..........----.((((..((((((.......))))))....((((((.....)))))).((((........)))).....))))---........------- ( -31.00, z-score = -2.48, R) >droGri2.scaffold_15112 869979 95 + 5172618 --AAAAAGUUAUUU-UCGCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGCC--UGUUGAUA------- --.....((((...-..((((..((((((.......))))))....((((((.....)))))).((((........)))).....)))).--...)))).------- ( -30.80, z-score = -2.37, R) >anoGam1.chr2L 44195362 88 + 48795086 -AAAAAGCUUCAUGC--GCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGCGU---------------- -............((--((((..((((((.......))))))....((((((.....)))))).((((........)))).....))))))---------------- ( -34.10, z-score = -3.18, R) >consensus __AAAAAAGCAU____CGCCCAACGUGGGGCUCGAACCCACGACCCUGAGAUUAAGAGUCUCAUGCUCUACCGACUGAGCUAGCCGGGC__AUGUUAAAUUGG____ .................((((..((((((.......))))))....((((((.....)))))).((((........)))).....)))).................. (-30.32 = -30.32 + 0.00)

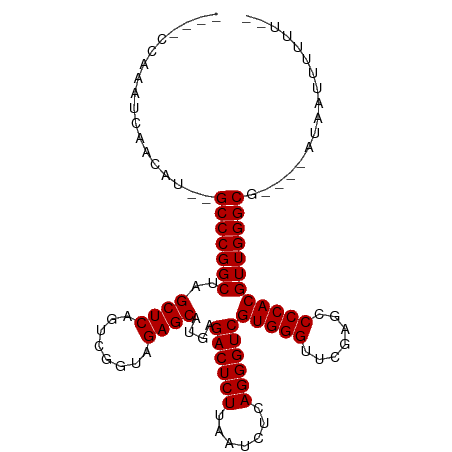
| Location | 2,835,230 – 2,835,329 |
|---|---|
| Length | 99 |
| Sequences | 13 |
| Columns | 107 |
| Reading direction | reverse |
| Mean pairwise identity | 83.62 |
| Shannon entropy | 0.37405 |
| G+C content | 0.53247 |
| Mean single sequence MFE | -34.06 |
| Consensus MFE | -32.61 |
| Energy contribution | -32.61 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.84 |
| Structure conservation index | 0.96 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 2.27 |
| SVM RNA-class probability | 0.987261 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 2835230 99 - 21146708 --UGUCAAGAACACAU--GCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGCG----AUGUUUUUUAUU --....(((((((..(--(((((((..((((........))))....((((((.......))))))(((((.......)))))))))))))----.))))))).... ( -36.70, z-score = -2.64, R) >droSim1.chr2R 1651958 97 - 19596830 --GGCCAAGUACACAU--GCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGCG----AUGCUUUUUU-- --....(((((....(--(((((((..((((........))))....((((((.......))))))(((((.......)))))))))))))----.)))))....-- ( -34.10, z-score = -1.14, R) >droSec1.super_1 517287 97 - 14215200 --GGCCAAUUACACAU--GCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGCG----AUGCUUUUUU-- --(.(((((....(((--((((((((.....))))))....))))).((((((.......))))))(((((.......)))))))))).).----..........-- ( -34.00, z-score = -1.40, R) >droYak2.chr2L 15567447 97 - 22324452 --GACCAUUUUCACAU--GCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGCG----AUGCAUUUUU-- --...........(((--(((((((..((((........))))....((((((.......))))))(((((.......)))))))))))).----))).......-- ( -33.40, z-score = -1.65, R) >droEre2.scaffold_4929 20278928 98 - 26641161 --AAUAUACAUUAUUC-AGCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGCG----GAUCUUUUUU-- --............((-.(((((((..((((........))))....((((((.......))))))(((((.......)))))))))))).----))........-- ( -33.30, z-score = -2.17, R) >droAna3.scaffold_13266 9158470 98 - 19884421 ----UAAUUUCAACA---GCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGCGACUUACAAUUUUUU-- ----...........---(((((((..((((........))))....((((((.......))))))(((((.......))))))))))))...............-- ( -32.90, z-score = -2.35, R) >dp4.chr3 1124948 94 - 19779522 -----CUAAAUUACAU--GCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGCG----ACGUUUUUUU-- -----..........(--(((((((..((((........))))....((((((.......))))))(((((.......)))))))))))))----..........-- ( -33.00, z-score = -2.07, R) >droPer1.super_2 1289196 96 - 9036312 ---GCCUAAAUUACAU--GCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGCG----ACGUUUUUUU-- ---((((((....(((--((((((((.....))))))....))))).((((((.......))))))(((((.......))))).)))))).----..........-- ( -35.50, z-score = -2.35, R) >droWil1.scaffold_180700 5892402 101 - 6630534 -GCUCAGGUGCAACA--UGCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGCGA-AAAUACUUUUUU-- -((......))....--((((((((..((((........))))....((((((.......))))))(((((.......))))))))))))).-............-- ( -34.70, z-score = -1.36, R) >droVir3.scaffold_12823 845589 106 + 2474545 GACGCAAGGCGAACAAGUGCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGCGAGCAUUAACAUUUUU- ...((...((......))(((((((..((((........))))....((((((.......))))))(((((.......))))))))))))..))............- ( -35.30, z-score = -0.92, R) >droMoj3.scaffold_6496 25272262 91 - 26866924 -------GAUCAACA---GCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGCG----ACAAUUUUUC-- -------........---(((((((..((((........))))....((((((.......))))))(((((.......)))))))))))).----..........-- ( -32.90, z-score = -2.11, R) >droGri2.scaffold_15112 869979 95 - 5172618 -------UAUCAACA--GGCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGCGA-AAAUAACUUUUU-- -------........--.(((((((..((((........))))....((((((.......))))))(((((.......))))))))))))..-............-- ( -32.70, z-score = -1.99, R) >anoGam1.chr2L 44195362 88 - 48795086 ----------------ACGCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGC--GCAUGAAGCUUUUU- ----------------.((((((((..((((........))))....((((((.......))))))(((((.......))))))))))))--).............- ( -34.30, z-score = -1.79, R) >consensus ____CCAAAUCAACAU__GCCCGGCUAGCUCAGUCGGUAGAGCAUGAGACUCUUAAUCUCAGGGUCGUGGGUUCGAGCCCCACGUUGGGCG____AUAAUUUUUU__ ..................(((((((..((((........))))....((((((.......))))))(((((.......))))))))))))................. (-32.61 = -32.61 + -0.00)
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:04:33 2011