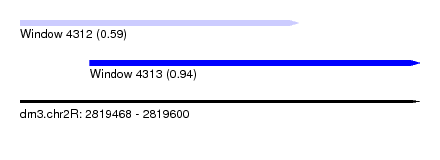
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 2,819,468 – 2,819,600 |
| Length | 132 |
| Max. P | 0.940706 |

| Location | 2,819,468 – 2,819,560 |
|---|---|
| Length | 92 |
| Sequences | 12 |
| Columns | 107 |
| Reading direction | forward |
| Mean pairwise identity | 69.43 |
| Shannon entropy | 0.62901 |
| G+C content | 0.63217 |
| Mean single sequence MFE | -40.39 |
| Consensus MFE | -16.56 |
| Energy contribution | -17.07 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.58 |
| Mean z-score | -1.19 |
| Structure conservation index | 0.41 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.591489 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 2819468 92 + 21146708 ------------GUCCGGCGAGGAGUCCGGCGAACCGGCCAAAUUGGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGCGCG---UGGGGCUGCGGAU ------------(((((.(....(((((.(((..((.(((.....))).)).....(((((((((.((((.....))))))))))))).))---).))))))))))) ( -48.90, z-score = -1.82, R) >droSim1.chr2R 1635923 92 + 19596830 ------------GUCCGGCGAGGAGUCCGGCGAACCGGCCAAAUUGGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGCGCG---UGUGGCUGCGGAU ------------.(((.....)))(((((((......)))......((((.((((((((((((((.((((.....)))))))))))))).)---).)).)))))))) ( -46.00, z-score = -1.06, R) >droSec1.super_1 501517 92 + 14215200 ------------GUCCGGCGAGGAGUCCGGCGAACCGGCCAAAUUGGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGCGCG---UGUGGCUGCGGAU ------------.(((.....)))(((((((......)))......((((.((((((((((((((.((((.....)))))))))))))).)---).)).)))))))) ( -46.00, z-score = -1.06, R) >droYak2.chr2L 15552698 92 + 22324452 ------------GUCCGGCGAGGAGUCCGGUGAAGCGGCUAAAUUCGUGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGGAGCGCG---UGGGGCUGCGGAU ------------((((((((((.((.(((......)))))...))))).(((.((((((.(((((.((((.....))))))))).)))).)---)...))).))))) ( -40.40, z-score = -0.29, R) >droEre2.scaffold_4929 6910834 92 - 26641161 ------------GUCCGGCGAGGAGUCCGGUGAGCCGGUUAAAUUCGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGCGCG---UGGGGCUGCGGAU ------------((((((((((....((((....)))).....))))).(((.((((((((((((.((((.....)))))))))))))).)---)...))).))))) ( -49.20, z-score = -2.45, R) >droAna3.scaffold_13266 9145459 91 + 19884421 -------------UCCGGCGAGGAGUCCGGCGUUCCAGCCAGGUUCGUCUGCAGCUGCUGCAUCUCGCGCAUCUCGUGCAGCUGUAUCGCC---UGUGGCUGGGGAU -------------.(((((.....).))))..(((((((((.(..((..(((((((((.((.....))((.....))))))))))).))..---).))))))))).. ( -38.40, z-score = -0.32, R) >dp4.chr3 1099084 92 + 19779522 ---------------GUCCGGCGAGUCCGGGGAUCCGACUAGAUUCGAGGGCAGCUGCUGCAGCUCGCGCAUCUCGUGGAGCUGCAGCUGCGACUGCACCUGGGGGU ---------------((((..((((((..(........)..)))))).)))).((.((((((((((.(((.....))))))))))))).))......(((....))) ( -44.90, z-score = -1.20, R) >droPer1.super_65 158103 91 + 397436 ----------UUUGCCGGCAAGGAGUCCGGCGACCAGGCCAGAUGGAACACCGCCUUCUGCAGCGACAUCUGCAUGAGCGGAGCCAGCUUCG--AGAGGAUGC---- ----------.((((((((.....).)))))))...((((....).....((((.((.(((((......))))).)))))).))).(((((.--...))).))---- ( -30.30, z-score = 0.31, R) >droWil1.scaffold_180700 2338449 89 + 6630534 ------------AUCUGGCGAUGAUUCUGGUGAACCGGCUAGAUUCGUUGGCAAUUGCUGCAAUUCACGCAUCUCGUGCAUUUGCAAU------UGCGAUUGUGGAU ------------((((..((((((.((((((......)))))).))))))(((((((((((((..((((.....))))...)))))..------.)))))))))))) ( -31.50, z-score = -2.10, R) >droVir3.scaffold_12823 826226 89 - 2474545 ---------GCUGCCAUCCGGCGACUCUGGUGAGCCCGUCAGAUUGUCCGGCAGCUGUUGCAGUUCACGCAUCUCGUGCAGCUUCAAU------UGC---UGCGGAU ---------......(((((((((.((((..(....)..)))))))))..(((((.((((.((((((((.....)))).)))).))))------.))---))))))) ( -34.30, z-score = -1.01, R) >droMoj3.scaffold_6496 25936970 87 - 26866924 -----------UGCCAUCUGGUGAUUCAGGUGAACCAGCCAGAUUGUCAGGCAGCUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGU------UGC---UGCGGAU -----------..(((((((((..(((....)))...)))))))......(((((.(((((((((.((((.....)))))))))))))------.))---))))).. ( -45.20, z-score = -3.15, R) >droGri2.scaffold_15245 16383262 98 + 18325388 ACCACCACUACCACCAUCUGGGGAUUCGGGCGAGCCACUCAGAUUGUCGGGCAACUGCUGCAAUUCACGCAUCUCGUGCAGCUGCAAC------UGC---UGCGGAU .((.((....((.((....))))....))(((((.......(((((((((....)))..)))))).......)))))(((((......------.))---))))).. ( -29.64, z-score = -0.07, R) >consensus ____________GUCCGGCGAGGAGUCCGGCGAACCGGCCAGAUUGGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGCGCG___UGCGGCUGCGGAU .........................((((((......)))................(((((((((((((.....)))).)))))))))...............))). (-16.56 = -17.07 + 0.50)
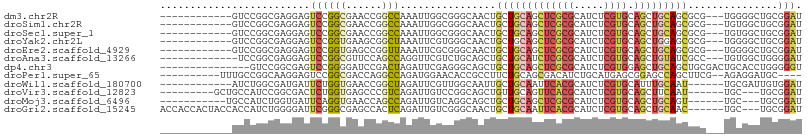
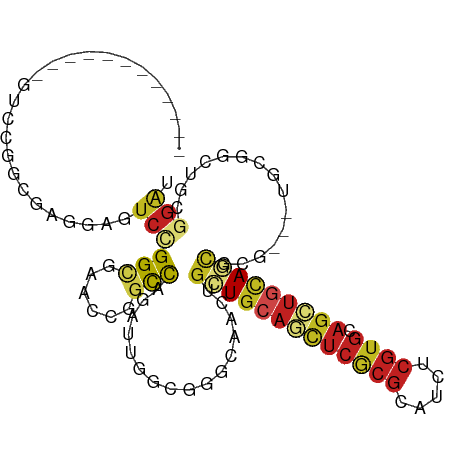
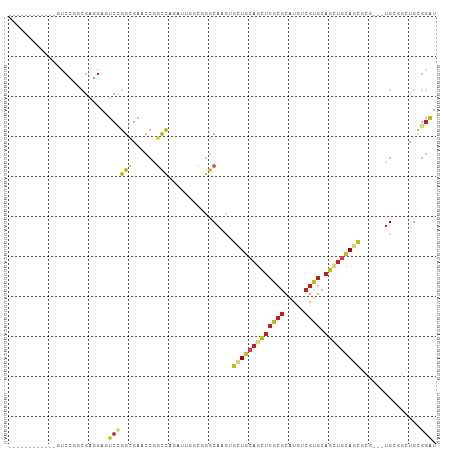
| Location | 2,819,491 – 2,819,600 |
|---|---|
| Length | 109 |
| Sequences | 12 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 73.50 |
| Shannon entropy | 0.59443 |
| G+C content | 0.58507 |
| Mean single sequence MFE | -45.87 |
| Consensus MFE | -15.69 |
| Energy contribution | -16.48 |
| Covariance contribution | 0.79 |
| Combinations/Pair | 1.67 |
| Mean z-score | -2.37 |
| Structure conservation index | 0.34 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 1.47 |
| SVM RNA-class probability | 0.940706 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 2819491 109 + 21146708 CGGCCAAAUUGGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGC---GCGUGGGGCUGCGGAUCCUUAGUCCGCAGCACUACAAUCUCAGAGUUCUGCUUCAC ..(((.....)))(((((((((((((((((.((((.....)))))))))))))---..((((.(((((((((.....))))))))).))))........)))..)))))... ( -55.00, z-score = -4.20, R) >droSim1.chr2R 1635946 109 + 19596830 CGGCCAAAUUGGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGC---GCGUGUGGCUGCGGAUCCUUGGUCCGCAGCACUACAAUCUCGGAGUUCUGCUUCAC (.(((.....))).)(((..((((((((((.((((.....)))))))))))))---)..))).(((((((((.....)))))))))...........((((.....)))).. ( -54.00, z-score = -3.46, R) >droSec1.super_1 501540 109 + 14215200 CGGCCAAAUUGGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGC---GCGUGUGGCUGCGGAUCCUUGGUCCGCAGCACUACAAUCUCGGAGUUCUGCUUCAC (.(((.....))).)(((..((((((((((.((((.....)))))))))))))---)..))).(((((((((.....)))))))))...........((((.....)))).. ( -54.00, z-score = -3.46, R) >droYak2.chr2L 15552721 109 + 22324452 CGGCUAAAUUCGUGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGGAGC---GCGUGGGGCUGCGGAUCCUUGGUCCGCAGCACCACAAUCUCGGAGUUCUGCUUCAC .(((..(((((((((....))(((.(((((.((((.....))))))))).)))---))((((.(((((((((.....))))))))).)))).......)))))..))).... ( -54.50, z-score = -3.91, R) >droEre2.scaffold_4929 6910857 109 - 26641161 CGGUUAAAUUCGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGC---GCGUGGGGCUGCGGAUCCUUGGUCCGCAGCACUACAAUCUCGGAGUUCUGCUUCAC .(((..(((((((((....))(((((((((.((((.....)))))))))))))---))((((.(((((((((.....))))))))).)))).......)))))..))).... ( -57.50, z-score = -5.35, R) >droAna3.scaffold_13266 9145481 109 + 19884421 CAGCCAGGUUCGUCUGCAGCUGCUGCAUCUCGCGCAUCUCGUGCAGCUGUAUC---GCCUGUGGCUGGGGAUCCUUGGUGCGCAGCAUCACUAUCUCCGAGUUCUGCUUCAC .(((((.(..((..(((((((((.((.....))((.....))))))))))).)---)..).)))))((((((...((((((...))))))..)))))).............. ( -38.50, z-score = -0.07, R) >dp4.chr3 1099104 112 + 19779522 CGACUAGAUUCGAGGGCAGCUGCUGCAGCUCGCGCAUCUCGUGGAGCUGCAGCUGCGACUGCACCUGGGGGUCCUUGGUGCGCAGCAGGACUAUCUCCGAGUUCUGCUUGAC (((......)))...((.((.((((((((((.(((.....))))))))))))).))....(((((.(((...))).)))))))(((((((((.......))))))))).... ( -53.20, z-score = -1.95, R) >droPer1.super_65 158128 101 + 397436 AGGCCAGAUGGAACACCGCCUUCUGCAGCGACAUCUGCAUGAGCGGAGCCAGC-----------UUCGAGAGGAUGCGCUCGGUGCGGUUUUCCAUGUGGGUCAGGCUGGAC .(((((.((((((.(((((...(((.((((.(((((.......(((((....)-----------))))...)))))))))))).))))).)))))).).))))......... ( -43.70, z-score = -1.59, R) >droWil1.scaffold_180700 2338472 106 + 6630534 CGGCUAGAUUCGUUGGCAAUUGCUGCAAUUCACGCAUCUCGUGCAUUUGCAAU------UGCGAUUGUGGAUCUUUUGUGCGUAAGACAACUAUUUCGGAAUUCUGUUUAAC .(((..((((((((.(((((((((((((..((((.....))))...)))))..------.)))))))).)))...((((.(....)))))........)))))..))).... ( -27.40, z-score = -0.72, R) >droVir3.scaffold_12823 826252 103 - 2474545 CCGUCAGAUUGUCCGGCAGCUGUUGCAGUUCACGCAUCUCGUGCAGCUUCAAU------UGC---UGCGGAUCUUUGGUGCGCAAUAGCACAAUUUCCGAGUUCUGUUUCAC ....((((...((..(((((.((((.((((((((.....)))).)))).))))------.))---)))(((......((((......))))....)))))..))))...... ( -33.30, z-score = -1.64, R) >droMoj3.scaffold_6496 25936994 103 - 26866924 CAGCCAGAUUGUCAGGCAGCUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGU---------UGCUGCGGAUCUUUGGUGCGCAUCAGCACAAUCUCCGAGUUCUGCUUGGC ..(((((...(.((((((((.(((((((((.((((.....)))))))))))))---------.)))))(((......((((......))))....))).....))))))))) ( -48.50, z-score = -2.17, R) >droGri2.scaffold_15245 16383297 103 + 18325388 CACUCAGAUUGUCGGGCAACUGCUGCAAUUCACGCAUCUCGUGCAGCUGCAAC---------UGCUGCGGAUCCUUGUGGCGCAACAGCACAAUCUCCGAGUUCUGCUUGAC .......((((((((....)))..)))))(((.(((.(((..(((((......---------.)))))(((...(((((.(......))))))..))))))...))).))). ( -30.90, z-score = 0.07, R) >consensus CGGCCAGAUUGGCGGGCAACUGCUGCAGCUCGCGCAUCUCGUGCAGCUGCAGC___GC_UG_GGCUGCGGAUCCUUGGUGCGCAGCACCACAAUCUCCGAGUUCUGCUUCAC .............(((((((((((((((((((((.....)))).)))))))))...........(((.(.((.....)).).)))..............)))..)))))... (-15.69 = -16.48 + 0.79)

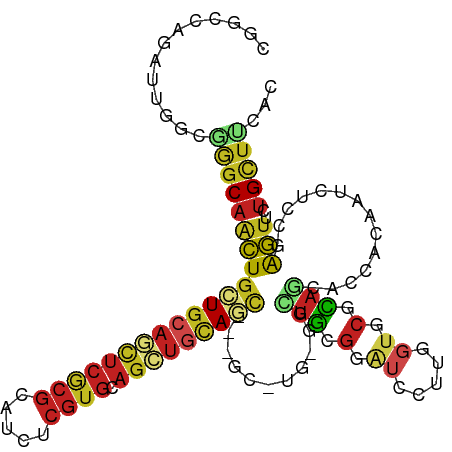
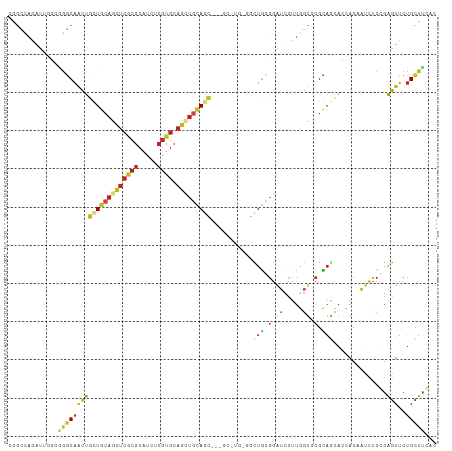
Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:04:26 2011