
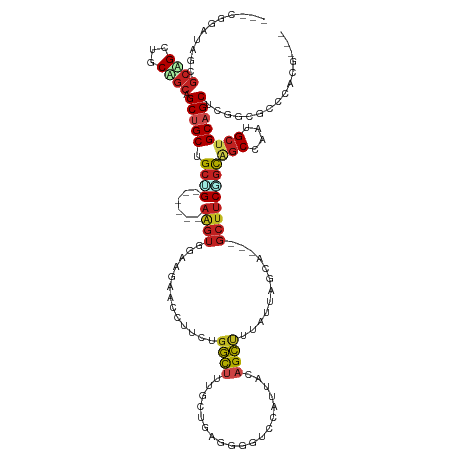
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 1,249,531 – 1,249,649 |
| Length | 118 |
| Max. P | 0.845955 |

| Location | 1,249,531 – 1,249,649 |
|---|---|
| Length | 118 |
| Sequences | 12 |
| Columns | 133 |
| Reading direction | forward |
| Mean pairwise identity | 70.09 |
| Shannon entropy | 0.62839 |
| G+C content | 0.58937 |
| Mean single sequence MFE | -49.56 |
| Consensus MFE | -15.59 |
| Energy contribution | -16.71 |
| Covariance contribution | 1.12 |
| Combinations/Pair | 1.39 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.31 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.81 |
| SVM RNA-class probability | 0.822962 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 1249531 118 + 21146708 ---CGGAUAGCGCUGCUGCAGCCGCUGCUGCGG------AGGUGGAAGAACCAUCUGGCUUUGCAGAGGGGUCCAUUACAGCUUUAUUAGUA---GCUUCAGCAGCUAAUGCUGCAGCUCGGCGCCCGCG--- ---(((...(((((((((((((.(((((((..(------..(((((....((.((((......)))).)).)))))..)((((........)---))).)))))))....))))))))..))))))))..--- ( -53.60, z-score = -1.61, R) >droSim1.chr2R 193451 118 + 19596830 ---CGGAUAGAGCUGCUGCAGCAGCUGCUGCGG------AAGUGGAAGAACCAUCGGGCUUUGCAGAGGGGUCCAUUACAGCUUUGUUAGUA---GCUUCCGCAGCUAAUGCUGCAGCUCGGCGCCCGCG--- ---(((.....((((((((((((...(((((((------((((((.....)).....(((..((((((..((.....))..)))))).))).---)))))))))))...)))))))))..)))..)))..--- ( -54.20, z-score = -2.01, R) >droSec1.super_59 152075 118 + 165940 ---CGGAUAGAGCUGCUGCAGCAGCUGCUGCGG------AAGUGGAAGAACCAUCGGGCUUUGCAGAUGGGUCCAUUACAGCUUUGUUAGUA---GCUUCCGCAGCUAAUGCUGCAGCUCGGCGCCCGCG--- ---(((.....((((((((((((...(((((((------(((((((....(((((..((...)).))))).)))......(((.....))).---)))))))))))...)))))))))..)))..)))..--- ( -58.10, z-score = -3.38, R) >droYak2.chr2L_random 3370795 118 + 4064425 ---CGGAUAGCGCAGAUGCAGCCGCUGCUGCGG------AAGUUGAAGAGCCAUCUGGCUUUGCAGAGGGGUCCAUUACAGCUUUAUUAGUA---GCUUCGGCAGCUAAUGCUGCAGCUCGGCGCCCCCG--- ---(((...((((....)).((((..(((((((------(((((((((..((.((((......))))..))..).)).))))))(((((((.---((....)).)))))))))))))).))))))..)))--- ( -49.80, z-score = -1.22, R) >droEre2.scaffold_4929 5349380 118 - 26641161 ---CGGAUAGCGCAGCUGCAGCCGCUGCUGCGG------AAGUGGAAGAACCAUCUGGCUUUGCAGAGGGGUCCAUUACAGCUUUAUUAGUA---GCUUCGGCAGCUAACGCUGCAGCUCGGCGCCCGCG--- ---(((...(((((((((((((....(((((.(------(((((((....((.((((......)))).)).)))......(((.....))).---))))).)))))....)))))))))..)))))))..--- ( -54.80, z-score = -2.21, R) >droAna3.scaffold_13280 66263 112 - 1428709 ------CAAGUGCAG---CGGCUGCUGCAGCUG------AAGUAGAGGAACCUUCUGGUCUGGAUAAGGGGUCCAUUACAGCUUUAUUUGCU---GCUUCUGCAGCCACUGCUGCAGCUCUGCGUCCCCG--- ------.....((((---.((((((.((((.((------((((((((..(((....))).(((((.....)))))......))))))))(((---((....))))))))))).)))))))))).......--- ( -44.70, z-score = -1.11, R) >dp4.Unknown_group_488 5194 121 - 14360 UCUCCGAAAAUGCAGCAGCUGCUGCUGCAGCUG------AGGUUGAAGAGCCUUCUGGCUUAGCUGUUGGAUCCAUUACAGCCUUAUUAGCA---GCUUCGGCAGCCACUGCUGCAGCGCGACGACCACG--- ((((((....((((((((..(((((((.(((((------....(((.(((((....))))).(((((((....))..))))).)))....))---))).)))))))..)))))))).)).)).)).....--- ( -52.70, z-score = -2.78, R) >droPer1.super_2 210594 120 + 9036312 -UCUCGAAAAUGCAGCAGCUGCUGCUGCAGCUG------AGGUUGAAGAGCCUUCUGGCUUAGCUGUUGGAUCCAUUACAGCCUUAUUAGCA---GCUUCGGCAGCCACUGCUGCAGCGCGACGACCACG--- -(((((....((((((((..(((((((.(((((------....(((.(((((....))))).(((((((....))..))))).)))....))---))).)))))))..))))))))...))).)).....--- ( -53.80, z-score = -3.01, R) >droWil1.scaffold_180569 1050537 127 + 1405142 CCUCACUGAGCGCCGCAGCGGCAGCCGCUGCUGCCUUCGAAGUGGAUGAACCUUCUGGCUUGGCUGUGGGAUCCAUAACAGCCUUAUUAACU---GCUUCAGCAGCCACUGCUGCAGCACGACGUCCUCU--- ......((((.((.(((((((...))))))).)).)))).((.(((((..((....))...((((((.(....)...))))))........(---(((.((((((...)))))).))))...))))).))--- ( -48.70, z-score = -1.43, R) >droVir3.scaffold_12875 12313253 118 + 20611582 UUUCAUGAAACGCAG---CUGCAGCUGCUGCCG------AAGUUGAGGAACCUGCAGGCUUAGCUGUUGGAUCCAUGACAGCUUUAUUGACC---GCUUCGGCAGCGACAGCCGCAGCACGACGACCACG--- ..............(---((((.((((((((((------((((.(.(((.((.((((......)))).)).)))....(((.....))).).---)))))))))))...))).)))))............--- ( -45.00, z-score = -2.49, R) >droMoj3.scaffold_6496 12309546 118 - 26866924 CUUCGUUGAAUGCAG---CUACAGCUGCUGCUG------AAGUUGAGGAACCUGGGGGCUUUGCUGUAGGAUCCAUAACAGCCUUAUUAACC---GCUUCGGCGGCAACAGCUGCAGCACGACGGCCACG--- ..((((((..(((((---((.((((....))))------..((((.(((.((((.((......)).)))).)))................((---((....)))))))))))))))...)))))).....--- ( -48.20, z-score = -2.71, R) >anoGam1.chr2L 21716009 112 - 48795086 ------CGGAUCCCGC-ACUGUCGCCGCUGCUG--------------CCACCCUCCAUCUUAACCGGCCGCUUCUCGCUCGACCCACCGGCUCCGGCAUCGUUACCCGACGCCGCCGCCCGCCGCUUCCCCUU ------.(((..(.((-...((((.((..((.(--------------((................))).))....))..)))).....(((..((((.(((.....))).))))..))).)).)..))).... ( -31.09, z-score = -0.88, R) >consensus ___CGGAUAGCGCAGCUGCAGCAGCUGCUGCUG______AAGUGGAAGAACCUUCUGGCUUUGCUGAGGGGUCCAUUACAGCUUUAUUAGCA___GCUUCGGCAGCCAAUGCUGCAGCUCGGCGCCCACG___ ...........((((...)))).(((((.............(((((((........((((...................)))).............)))))))(((....))))))))............... (-15.59 = -16.71 + 1.12)

| Location | 1,249,531 – 1,249,649 |
|---|---|
| Length | 118 |
| Sequences | 12 |
| Columns | 133 |
| Reading direction | reverse |
| Mean pairwise identity | 70.09 |
| Shannon entropy | 0.62839 |
| G+C content | 0.58937 |
| Mean single sequence MFE | -48.39 |
| Consensus MFE | -17.54 |
| Energy contribution | -16.92 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.62 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.36 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.89 |
| SVM RNA-class probability | 0.845955 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>dm3.chr2R 1249531 118 - 21146708 ---CGCGGGCGCCGAGCUGCAGCAUUAGCUGCUGAAGC---UACUAAUAAAGCUGUAAUGGACCCCUCUGCAAAGCCAGAUGGUUCUUCCACCU------CCGCAGCAGCGGCUGCAGCAGCGCUAUCCG--- ---....(((((...((((((((....(((((((.(((---(........)))).....(((((..((((......)))).)))))........------...))))))).)))))))).))))).....--- ( -50.00, z-score = -2.19, R) >droSim1.chr2R 193451 118 - 19596830 ---CGCGGGCGCCGAGCUGCAGCAUUAGCUGCGGAAGC---UACUAACAAAGCUGUAAUGGACCCCUCUGCAAAGCCCGAUGGUUCUUCCACUU------CCGCAGCAGCUGCUGCAGCAGCUCUAUCCG--- ---...((((.....(((((((((...((((((((((.---..........((((((..((...))..)))..)))....(((.....))))))------)))))))...))))))))).))))......--- ( -46.90, z-score = -1.79, R) >droSec1.super_59 152075 118 - 165940 ---CGCGGGCGCCGAGCUGCAGCAUUAGCUGCGGAAGC---UACUAACAAAGCUGUAAUGGACCCAUCUGCAAAGCCCGAUGGUUCUUCCACUU------CCGCAGCAGCUGCUGCAGCAGCUCUAUCCG--- ---...((((.....(((((((((...((((((((((.---.....(((....)))..((((.(((((.((...))..)))))....)))))))------)))))))...))))))))).))))......--- ( -50.20, z-score = -2.82, R) >droYak2.chr2L_random 3370795 118 - 4064425 ---CGGGGGCGCCGAGCUGCAGCAUUAGCUGCCGAAGC---UACUAAUAAAGCUGUAAUGGACCCCUCUGCAAAGCCAGAUGGCUCUUCAACUU------CCGCAGCAGCGGCUGCAUCUGCGCUAUCCG--- ---(((((((((.((..((((((....(((((.(((((---(........))))((...(((.((.((((......)))).)).)))...)).)------).)))))....)))))))).))))).))))--- ( -45.40, z-score = -1.00, R) >droEre2.scaffold_4929 5349380 118 + 26641161 ---CGCGGGCGCCGAGCUGCAGCGUUAGCUGCCGAAGC---UACUAAUAAAGCUGUAAUGGACCCCUCUGCAAAGCCAGAUGGUUCUUCCACUU------CCGCAGCAGCGGCUGCAGCUGCGCUAUCCG--- ---....(((((..((((((((((((.(((((.(((((---(........)))).....(((((..((((......)))).))))).......)------).)))))))).)))))))))))))).....--- ( -49.60, z-score = -1.91, R) >droAna3.scaffold_13280 66263 112 + 1428709 ---CGGGGACGCAGAGCUGCAGCAGUGGCUGCAGAAGC---AGCAAAUAAAGCUGUAAUGGACCCCUUAUCCAGACCAGAAGGUUCCUCUACUU------CAGCUGCAGCAGCCG---CUGCACUUG------ ---.(....)(((....))).(((((((((((....((---(((.........((...((((.......))))...))((((.........)))------).))))).)))))))---)))).....------ ( -43.80, z-score = -1.66, R) >dp4.Unknown_group_488 5194 121 + 14360 ---CGUGGUCGUCGCGCUGCAGCAGUGGCUGCCGAAGC---UGCUAAUAAGGCUGUAAUGGAUCCAACAGCUAAGCCAGAAGGCUCUUCAACCU------CAGCUGCAGCAGCAGCUGCUGCAUUUUCGGAGA ---((((.....)))).(((((((((.(((((.(.(((---((.......((((((..((....)))))))).((((....)))).........------))))).).))))).))))))))).......... ( -48.90, z-score = -0.83, R) >droPer1.super_2 210594 120 - 9036312 ---CGUGGUCGUCGCGCUGCAGCAGUGGCUGCCGAAGC---UGCUAAUAAGGCUGUAAUGGAUCCAACAGCUAAGCCAGAAGGCUCUUCAACCU------CAGCUGCAGCAGCAGCUGCUGCAUUUUCGAGA- ---((((.....)))).(((((((((.(((((.(.(((---((.......((((((..((....)))))))).((((....)))).........------))))).).))))).))))))))).........- ( -48.90, z-score = -1.03, R) >droWil1.scaffold_180569 1050537 127 - 1405142 ---AGAGGACGUCGUGCUGCAGCAGUGGCUGCUGAAGC---AGUUAAUAAGGCUGUUAUGGAUCCCACAGCCAAGCCAGAAGGUUCAUCCACUUCGAAGGCAGCAGCGGCUGCCGCUGCGGCGCUCAGUGAGG ---.....((...((((((((((.(..(((((((....---.........((((((...((...))))))))..(((.((((.........))))...)))..)))))))..).))))))))))...)).... ( -56.20, z-score = -2.15, R) >droVir3.scaffold_12875 12313253 118 - 20611582 ---CGUGGUCGUCGUGCUGCGGCUGUCGCUGCCGAAGC---GGUCAAUAAAGCUGUCAUGGAUCCAACAGCUAAGCCUGCAGGUUCCUCAACUU------CGGCAGCAGCUGCAG---CUGCGUUUCAUGAAA ---(((((.(((...(((((((((...((((((((((.---.(.......((((((..((....)))))))).((((....))))...)..)))------)))))))))))))))---).)))..)))))... ( -53.30, z-score = -3.63, R) >droMoj3.scaffold_6496 12309546 118 + 26866924 ---CGUGGCCGUCGUGCUGCAGCUGUUGCCGCCGAAGC---GGUUAAUAAGGCUGUUAUGGAUCCUACAGCAAAGCCCCCAGGUUCCUCAACUU------CAGCAGCAGCUGUAG---CUGCAUUCAACGAAG ---(((.........(((((((((((((((((....))---))).......((((....(((.(((...((...))....))).))).......------)))))))))))))))---)........)))... ( -40.83, z-score = -0.99, R) >anoGam1.chr2L 21716009 112 + 48795086 AAGGGGAAGCGGCGGGCGGCGGCGUCGGGUAACGAUGCCGGAGCCGGUGGGUCGAGCGAGAAGCGGCCGGUUAAGAUGGAGGGUGG--------------CAGCAGCGGCGACAGU-GCGGGAUCCG------ ..(((...(((((.(((..((((((((.....))))))))..))).))..((((.((.....((.((((.((........)).)))--------------).)).))..))))..)-))....))).------ ( -46.60, z-score = -1.72, R) >consensus ___CGCGGGCGCCGAGCUGCAGCAGUAGCUGCCGAAGC___UGCUAAUAAAGCUGUAAUGGACCCCUCAGCAAAGCCAGAAGGUUCUUCAACUU______CAGCAGCAGCUGCAGCAGCUGCACUAUCCG___ ...............(((((.((..(((((......(......)......)))))..................((((....)))).................)).))))).((((...))))........... (-17.54 = -16.92 + -0.61)



Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:01:53 2011