
| Sequence ID | dm3.chr2R |
|---|---|
| Location | 1,182,672 – 1,182,762 |
| Length | 90 |
| Max. P | 0.791080 |

| Location | 1,182,672 – 1,182,762 |
|---|---|
| Length | 90 |
| Sequences | 10 |
| Columns | 93 |
| Reading direction | forward |
| Mean pairwise identity | 77.35 |
| Shannon entropy | 0.46748 |
| G+C content | 0.63042 |
| Mean single sequence MFE | -42.88 |
| Consensus MFE | -25.23 |
| Energy contribution | -24.75 |
| Covariance contribution | -0.48 |
| Combinations/Pair | 1.44 |
| Mean z-score | -1.41 |
| Structure conservation index | 0.59 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.70 |
| SVM RNA-class probability | 0.791080 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
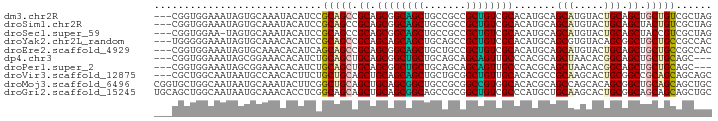
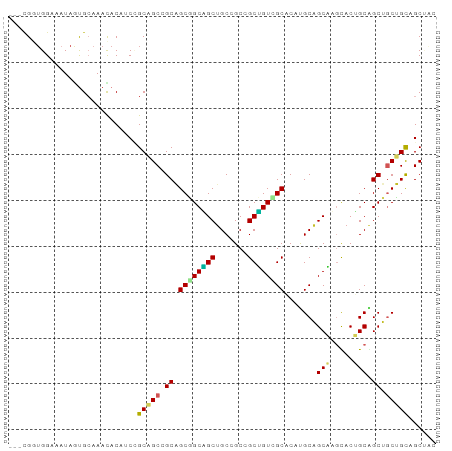
>dm3.chr2R 1182672 90 + 21146708 ---CGGUGGAAAUAGUGCAAAUACAUCCGCAGCCGCAGCGGCAGCUGCCGCCGCUGUCGCACAUGCAGCAUGUACUGCAGCUGCUGUCGCUAG ---..(((((..((.......))..))))).((.((((((((.......)))))))).))(((.(((((.((.....))))))))))...... ( -34.80, z-score = 0.38, R) >droSim1.chr2R 129854 90 + 19596830 ---CGGUGGAAAUAGUGCAAAUACAUCCGCAGCCGCAGCGGCAGCUGCCGCCGCUGUCGCACAUGCAGCAUGUACUGCAGCUACUGUCGCUAG ---.(((((...(((((((..((((((.(((((.((((((((.......)))))))).))...))).).))))).)))).)))...))))).. ( -35.10, z-score = -0.35, R) >droSec1.super_59 109251 89 + 165940 ---CGGUGGAA-UAGUGCAAAUACAUCCGCAGCCGCAGCGGCAGCUGCCGCCGCUGUCGCACAUGCAGCAUGUACUGCAGCUACCGUCGCUAG ---((((((..-...((((..((((((.(((((.((((((((.......)))))))).))...))).).))))).)))).))))))....... ( -35.60, z-score = -0.60, R) >droYak2.chr2L_random 764427 90 - 4064425 ---UGGGGGAAAUAGUGCAAACACAUCCGCAGCCGCAGCAGCAGCUGCAGCCGCUGUCGCACAUGCAGCGUGUACAGCGGCUGCUGCCGCCAC ---(((.(......(((....)))....(((((.(((((....)))))(((((((((.((((.......))))))))))))))))))).))). ( -44.10, z-score = -1.91, R) >droEre2.scaffold_4929 21345429 90 + 26641161 ---CGGUGGAAAUAGUGCAAACACAUCAGCAGCCGCAGCGGCAGCUGCUGCCGCUGUCGCACAUGCAGCAUGUACUGCAGCUGCUGCCGCCAC ---.(((((.....(((....)))..((((((((((((((((((...))))))))).))....(((((......))))))))))))))))).. ( -46.90, z-score = -2.87, R) >dp4.chr3 19689006 87 - 19779522 ---CGGUGGAAAUAGCGGAAACACAUCUGCAGCUGCAGCGGCUGCUGCAGCAGCAGUUGCCCACGCAGCUAACACGGCAGCUGCUGCAGC--- ---...........(((((......))))).(((((((((((((((((....))((((((....)))))).....)))))))))))))))--- ( -44.90, z-score = -2.73, R) >droPer1.super_2 8757717 87 - 9036312 ---CGGUGGAAAUAGCGGAAACACAUCUGCAGCUGCAGCGGCUGCUGCAGCAGCAGUUGCCCACGCAGCUAACACGGCAGCUGCUGCAGC--- ---...........(((((......))))).(((((((((((((((((....))((((((....)))))).....)))))))))))))))--- ( -44.90, z-score = -2.73, R) >droVir3.scaffold_12875 8270579 90 - 20611582 ---CGCUGGCAAUAAUGCCAACACUUCUGCUGCAGCUGCAGCAGCUGCUGCGGCUGUUGCACACGCCGCAAGCACUGCGGCCGCAGCAGCAGC ---.(.(((((....))))).).((.(((((((.(((((((....(((((((((((.....)).))))).))))))))))).))))))).)). ( -48.70, z-score = -2.06, R) >droMoj3.scaffold_6496 14528279 93 + 26866924 CGGUGCUGGCAAUAAUGCAAAUACUUCGGCUGCAGCUGCAGCGGCUGCCGCGGCCGUGGCACACGCAGCCAGCACAGCGGCUGCAGCAGCUGC ..((((((((.....(((..........(((((....)))))(..((((((....))))))..)))))))))))).(((((((...))))))) ( -46.90, z-score = -0.54, R) >droGri2.scaffold_15245 8014436 93 + 18325388 UGCAGCUGGCAAUAAUGCAAACACCUCGGCAGCAGCUGCAGCGGCAGCCGCGGCUGUCGCCCAUGCUGCAAGCACUGCGGCAGCAGCAGCUGC .((((((((((....))).........(((.((((((((.((....)).)))))))).)))..(((((((.....)))))))....))))))) ( -46.90, z-score = -0.70, R) >consensus ___CGGUGGAAAUAGUGCAAACACAUCCGCAGCCGCAGCGGCAGCUGCCGCCGCUGUCGCACAUGCAGCAAGCACUGCAGCUGCUGCAGCUAC ............................(((((.((.((((((((.......)))))))).......(((.....))).)).)))))...... (-25.23 = -24.75 + -0.48)

| Location | 1,182,672 – 1,182,762 |
|---|---|
| Length | 90 |
| Sequences | 10 |
| Columns | 93 |
| Reading direction | reverse |
| Mean pairwise identity | 77.35 |
| Shannon entropy | 0.46748 |
| G+C content | 0.63042 |
| Mean single sequence MFE | -45.05 |
| Consensus MFE | -26.10 |
| Energy contribution | -26.02 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.39 |
| Mean z-score | -1.44 |
| Structure conservation index | 0.58 |
| Background model | dinucleotide |
| Decision model | sequence based alignment quality |
| SVM decision value | 0.69 |
| SVM RNA-class probability | 0.786879 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
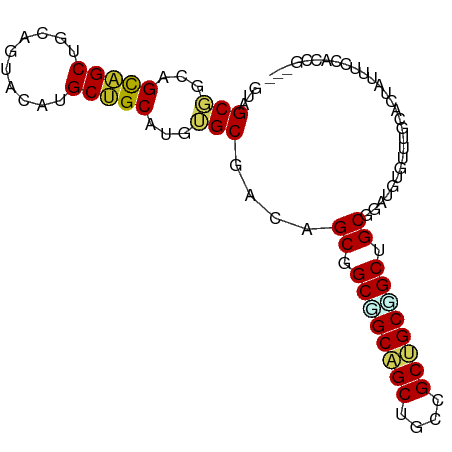
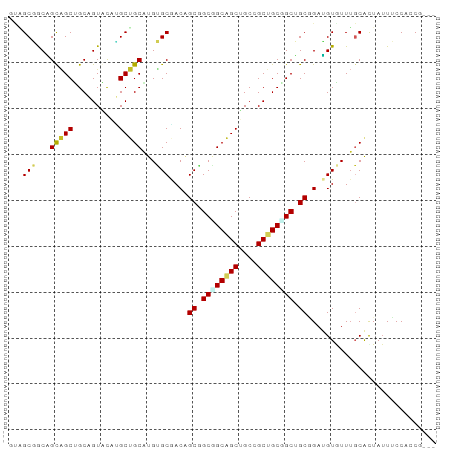
>dm3.chr2R 1182672 90 - 21146708 CUAGCGACAGCAGCUGCAGUACAUGCUGCAUGUGCGACAGCGGCGGCAGCUGCCGCUGCGGCUGCGGAUGUAUUUGCACUAUUUCCACCG--- .........((((((((.(((((((...)))))))...((((((((...))))))))))))))))(((.(((.......))).)))....--- ( -40.10, z-score = -1.22, R) >droSim1.chr2R 129854 90 - 19596830 CUAGCGACAGUAGCUGCAGUACAUGCUGCAUGUGCGACAGCGGCGGCAGCUGCCGCUGCGGCUGCGGAUGUAUUUGCACUAUUUCCACCG--- ...(.((.(((((.((((((((((.(((((.((.((.(((((((((...))))))))))))))))))))))).)))))))))))))....--- ( -39.80, z-score = -1.39, R) >droSec1.super_59 109251 89 - 165940 CUAGCGACGGUAGCUGCAGUACAUGCUGCAUGUGCGACAGCGGCGGCAGCUGCCGCUGCGGCUGCGGAUGUAUUUGCACUA-UUCCACCG--- .......((((((.((((((((((.(((((.((.((.(((((((((...))))))))))))))))))))))).))))))).-....))))--- ( -39.00, z-score = -0.90, R) >droYak2.chr2L_random 764427 90 + 4064425 GUGGCGGCAGCAGCCGCUGUACACGCUGCAUGUGCGACAGCGGCUGCAGCUGCUGCUGCGGCUGCGGAUGUGUUUGCACUAUUUCCCCCA--- (..((.((((((((.((((((..((((((......).)))))..)))))).)))))))).))..)(((.(((....)))....)))....--- ( -50.20, z-score = -2.83, R) >droEre2.scaffold_4929 21345429 90 - 26641161 GUGGCGGCAGCAGCUGCAGUACAUGCUGCAUGUGCGACAGCGGCAGCAGCUGCCGCUGCGGCUGCUGAUGUGUUUGCACUAUUUCCACCG--- ..((.((((((((((((.(((((((...)))))))...((((((((...))))))))))))))))))..(((....))).....)).)).--- ( -48.00, z-score = -2.23, R) >dp4.chr3 19689006 87 + 19779522 ---GCUGCAGCAGCUGCCGUGUUAGCUGCGUGGGCAACUGCUGCUGCAGCAGCCGCUGCAGCUGCAGAUGUGUUUCCGCUAUUUCCACCG--- ---......(((((((......)))))))(((((((.((((.((((((((....)))))))).)))).)))....))))...........--- ( -40.60, z-score = -1.08, R) >droPer1.super_2 8757717 87 + 9036312 ---GCUGCAGCAGCUGCCGUGUUAGCUGCGUGGGCAACUGCUGCUGCAGCAGCCGCUGCAGCUGCAGAUGUGUUUCCGCUAUUUCCACCG--- ---......(((((((......)))))))(((((((.((((.((((((((....)))))))).)))).)))....))))...........--- ( -40.60, z-score = -1.08, R) >droVir3.scaffold_12875 8270579 90 + 20611582 GCUGCUGCUGCGGCCGCAGUGCUUGCGGCGUGUGCAACAGCCGCAGCAGCUGCUGCAGCUGCAGCAGAAGUGUUGGCAUUAUUGCCAGCG--- (((.((((((((((.((((((((((((((.((.....))))))))..))).))))).)))))))))).)))(((((((....))))))).--- ( -57.30, z-score = -3.11, R) >droMoj3.scaffold_6496 14528279 93 - 26866924 GCAGCUGCUGCAGCCGCUGUGCUGGCUGCGUGUGCCACGGCCGCGGCAGCCGCUGCAGCUGCAGCCGAAGUAUUUGCAUUAUUGCCAGCACCG ((.((((...)))).)).((((((((((((.((((..((((.(((((.((....)).))))).))))..)))).)))).....)))))))).. ( -46.30, z-score = -0.42, R) >droGri2.scaffold_15245 8014436 93 - 18325388 GCAGCUGCUGCUGCCGCAGUGCUUGCAGCAUGGGCGACAGCCGCGGCUGCCGCUGCAGCUGCUGCCGAGGUGUUUGCAUUAUUGCCAGCUGCA ((((((((.((((.(((.((((.....))))..))).)))).))))))))....(((((((..((.(..(((....)))..).))))))))). ( -48.60, z-score = -0.11, R) >consensus GUAGCGGCAGCAGCUGCAGUACAUGCUGCAUGUGCGACAGCGGCGGCAGCUGCCGCUGCGGCUGCGGAUGUGUUUGCACUAUUUCCACCG___ ...(((...(((((..........)))))...)))....((.((((((((....)))))))).))............................ (-26.10 = -26.02 + -0.08)

Generated by rnazCluster.pl (part of RNAz 1.0) on Tue Apr 19 22:01:51 2011